作者简介: 高荣华, 女, 1977年生, 北京市农林科学院信息技术研究中心副研究员 e-mail: gaorh@nercita.org.cn
植物病害的自动早期检测对于作物精确保护至关重要。 提出了一种基于多维光谱序列(multi-dimensional spectral series, MDSS)和加权随机森林(weighted random forest, WRF)的番茄灰霉病早期诊断与鉴别方法。 目的是利用叶片多个观测维度的光谱曲线整体变化趋势建立作物病害检测模型, 以期在肉眼明显可见叶面病斑前对作物病害实现诊断。 将健康叶片接种灰霉病菌第3天作为叶片成功染病第1天。 试验首先采集番茄健康叶片和染病叶片7天内每天的高光谱图像, 提取感兴趣区域并计算平均光谱作为初始光谱数据, 经筛选共得到(156×7)组有效样本。 将样本数据按时间顺序拆分成分别包含1~7个维度的光谱数据形成多维原始光谱序列, 为增加维度间差异性, 相邻原始光谱序列相减构成多维关联光谱序列。 分别采用符号聚合近似估计(symbolic aggregate approximation, SAX)和符号傅里叶近似估计(symbolic Fourier approximation, SFA)两种符号化方法将光谱序列离散成局部辨别性特征。 基于多维光谱序列的局部辨别性特征建立加权随机森林(MDSS-SAX-SFA-WRF)分类模型, 实现病害早期检测。 相应地, 基于单维光谱序列(single-dimensional spectral series, SDSS)的番茄灰霉病识别模型被作为基准模型与MDSS-SAX-SFA-WRF模型比较。 试验结果显示, MDSS-SAX-SFA-WRF检测模型在包含2至7个光谱序列维度的56个测试样本数据中均获得90%以上识别准确率, 在包含5个光谱序列维度测试集中得到最高99%的识别准确率, 较SDSS-SAX-SFA-MRF检测模型在染病第5天的识别率高8.2个百分点。 另外受随机干扰的影响, SDSS-SAX-SFA-MRF模型准确率在染病5~7 d出现大幅度回落至最低84%, MDSS-SAX-SFA-WRF模型识别率在肉眼可见病斑阶段依然保持超过98%的较高检测水准, 未过度回落。 因此, 提出的基于多维光谱曲线整体变化趋势和加权随机森林(MDSS-SAX-SFA-WRF)的分类模型能够有效实现番茄灰霉病早期检测, 并具有较强的鲁棒性, 为染病初期的番茄灰霉病鉴别提供新思路。
Automatic early detection of plant diseases is essential for precision crop protection. This paper proposes an early diagnosis and detection method for tomato gray mold based on multi-dimensional spectral series (MDSS) and weighted random forest (WRF) algorithm. The aim was to establish a crop disease detection model by utilizing the overall trend of the spectral curve among multiple observation dimensions of the target leaves to realize the diagnosis before the leaf spot is visible. Generally, the third day after healthy leaves were inoculated with the Botrytis cinerea was treated as the first day that was successfully infected. Therefore, hyperspectral images were recorded from both healthy and infected leaves for 7 days after infection respectively. Then extracted, the region of interest and calculated the average spectrum to form the original spectral samples, whilst (156×7) groups were obtained in total after selection. The group samples were split into multi-dimensional spectral series with 1~7 dimensions per the course of the disease to make up multi-dimensional original spectral series. In order to increase the difference between dimensions, the adjacent original spectral series were subtracted to generate multi-dimensional related spectral series. Afterwards, two symbolic methods, symbolic aggregate approximation (SAX) and symbolic Fourier approximation (SFA), were employed to discretize each spectral series into local discriminant features. Finally, a weighted random forest classification model (MDSS-SAX-SFA-WRF) based on the local discriminant features of multi-dimensional spectral series is established to realize early disease detection. Accordingly, the model based on single-dimensional spectral series (SDSS) is also built as the benchmark to compare with the MDSS-SAX-SFA-WRF model. The experiment results indicate that the MDSS-SAX-SFA-WRF detection model achieves detection accuracies of more than 90% in 56 testing samples containing 2 to 7 spectral series dimensions, and the highest accuracy up to 99% reached in the 5-dimensional sample data, which is 8.2 percentage higher than that of SDSS-SAX-SFA-MRF detection model on the 5th day of infection. Different from the SDSS-SAX-SFA-MRF model detection performance dropped significantly to the lowest 84% in the 5th~7th days of infection due to random interference. While the discrimination accuracy of the MDSS-SAX-SFA-WRF model still retained a high level of more than 98% in the visible stage of infection without excessive decline. Therefore, the classification model based on the overall change trend of the multi-dimensional spectral curve and weighted random forest (MDSS-SAX-SFA-WRF) proposed in this paper can effectively realize the early detection of tomato gray mold with the strong robustness, which provides a new idea for the early differentiation of crop disease.
番茄灰霉病是一种低温、 高湿病害, 是棚室番茄栽培的常见病害。 该病繁殖速度快、 遗传变异大且适应性强, 在患病早期无法通过肉眼发现, 一旦进入发病期将扩散迅速, 对保护地番茄生产威胁极大, 已成为番茄设施栽培的主要限制因素。 2020年5月, 国务院颁布施行《农作物病虫害防治条例》, 条例中指出监测预警是做好农作物病虫害防控的前提和基础[1]。 虽然国内外对番茄灰霉病的防治研究已取得了一定进展, 但目前生产上仍缺乏该病害早期发现的有效途径。 实现对番茄灰霉病的早期检测对我国作物病害防治具有十分重要意义。
随着高光谱成像技术在作物病害识别中的深入应用, 对黄瓜[2]、 小麦[3]、 水稻[4]、 马铃薯[5]和苹果[6]等作物病害检测获得较高检出率。 高光谱具有唯一性特点, 不同作物中叶绿素等生物量含量不同导致其光反射率不同, 因此不同物质中光谱曲线走势和波峰波谷差异较大, 该种特异性使得基于光谱特征作物病害识别成为可能。 秦立峰[2]等提出融合病害差异信息改进的竞争性自适应重加权算法(competitive adaptive reweighted sampling, CARS)和连续投影算法(successive projections algorithms, SPA), 并运用二次降维寻优得到的特征波段建立了黄瓜霜霉病早期检测模型。 针对番茄病害检测, Sun[7]等利用基于小波变换和最小二乘支持向量机回归(least square support vector machine regression, WT-LSSVR)的方法筛选最佳波长并建立检测模型, 证明了高光谱成像技术在不同镉胁迫下测定番茄叶片重金属含量的可靠性和有效性。 贾方方[8]等利用去包络线法(continuum removal)筛选对叶霉病发病程度识别的敏感波段, 构建基于光谱特征吸收参量的发病程度估测模型。 上述研究基本思路均是首先对所获取作物叶片高光谱图像进行病斑或叶片分割并计算平均光谱曲线, 后利用降维工具或相关光谱植被指数(spectral vegetation indices, SVI)对光谱特征波段筛选和提取[9], 过滤不做分类贡献的波段信息, 最后基于提取的特征波段建立作物病害识别分类器。 Xie[10]等对比了基于全波段和使用特征排序(feature ranking, FR)算法筛选的敏感波段分别建立的K-最近邻(K nearest neighbor, KNN)模型对5级患病程度番茄灰霉病叶片的分类准确率, 在测试集中, 基于全波段KNN的总体分类结果为61.11%, FR-KNN模型为45.83%。 由此可见, 仅用个别波段光谱信息作为分类依据, 忽略了光谱曲线整体变化趋势, 筛选出不合格波段更易导致曲线信息片面化, 尤其患病早期叶片与健康叶片光谱曲线差异不显著, 容易导致识别失误, 错过病害早期最佳防治时间。
借鉴多维时间序列分类算法(multivariate time series classification, MTSC), 以番茄灰霉病患病早期叶片光谱曲线为基础, 从接病第3天(染病第1天)至完全发病连续采集图像, 根据图像不同波段上反射率随时间推移而产生的变化来监测作物病害的发病情况, 完整描述患病区域在不同波段下、 不同发病时期光谱反射率变化趋势, 建立基于多维光谱序列(multi-dimensional spectral series, MDSS)的作物病害分类器, 以实现对番茄灰霉病的早期检测。
实验采用盆栽培育番茄幼苗30株。 仪器采用四川双利合谱科技有限公司的GaiaField-V10内置推扫式的便携式高光谱成像仪, 成像仪光谱范围为400~1 000 nm, 光谱分辨率为4 nm, 选取360个光谱通道, 单幅拍摄速度15 s, 全幅图像像素分辨率为960× 1 101。 为了确保采集的图像清晰且不失真, 经预备实验, 确定曝光时间为7.5 ms, 物距为50 cm, 图像采集速度为16 mm· s-1。
图像采集方式如图1所示, 用支架固定高光谱相机, 设置高光谱镜头背对太阳且斜向下30° 与叶片垂直架设, 避免在采集过程中光谱仪吸收太阳光导致成像不准确。 图像采集时将番茄盆栽置于纯色背景板前, 以去除复杂背景的影响, 同时在地面标定镜头与盆栽的相对位置以保证采集距离不变。 从30盆番茄中选出生长状态良好、 叶片平展宽大的180片叶片作为序列采集对象, 其中90片作为接病实验组, 90片为正常生长健康对照组。 为了探究侵染灰霉病番茄叶片光谱信息随时间推移而产生的变化, 以接种灰霉病菌第3天(染病第1天)作为番茄高光谱序列图像采集的开端, 对选定的叶片以固定姿势和角度连续采集7 d。
由于每天需采集180张叶片, 拍摄过程持续时间较长, 为消除光照强度对高光谱图像成像影响, 尽量选取上下午光照强度相同时间段采集。 此外, 在光照强度较弱时间段的采集过程中采用补光灯对番茄盆栽进行补光, 并根据环境条件变化, 对应采集全白与全黑标定图像, 用作计算不同光照情况下番茄叶片的光谱反射率值。 经过对30盆番茄盆栽连续采集, 共采集了180片叶片360个光谱波段上的7天连续数据。 经过预处理剔除模糊、 过曝等不合格实例后, 剩余171个叶片(实验组86个, 对照组85个)进行光谱信息分析试验。
首先对番茄叶片图像进行镜头积分球矫正, 消减光照影响, 再分别提取叶片病斑部分、 健康部分和整片叶子感兴趣区域, 获得对比光谱反射率曲线如图2所示。 图2(a)展示了染病第6天番茄叶片状态, 可明显看出染病区域呈深褐色, 图2(b)表示三个不同感兴趣区域选取, 橙色为整片叶片, 绿色为健康区域, 红色为病斑区域, 对三个区域光谱反射率做平均处理得到图2(c)。 可知患病区域平均光谱与健康区域有较大差别, 整片叶片光谱曲线受病斑区域影响与健康叶片曲线有轻微差异, 随着时间延长, 该差异逐渐增大, 如图2(d)所示, 由此可从整片叶片光谱曲线随时间延长而产生的变化来判断该叶片患病情况。
作物在染病初期, 如图3所示, 由于无法用肉眼识别患病区域, 对171片叶片进行整片光谱信息提取。 其中部分叶片在不同观察日存在叶片卷曲、 脱落等问题, 为保证多维序列样本完整性, 删除数据维度小于7 d的样本, 共得到156个可供实验样本数据, 其中(83× 7)组患病实验组叶片, (73× 7)组对照组叶片, 全部接病叶片与健康叶片在连续7天的观测下得到的光谱信息如图4所示。 可以看出, 随着接病天数的增加, 叶片的平稳反射率在波长750~1 000 nm区间内越来越接近1, 该变化为病叶判断提供可能。 以番茄叶片高光谱图像的360个通道波长作为序列长度, 将156组试验样本按照近似65:35比例划分为100组训练数据和56组测试数据进行光谱图像分析。
在探究作物病害的高光谱数据中, 光谱序列指一个目标区域在相同波长间隔上连续取反射率值生成的一组数据。 以一维光谱序列为基础, 多维光谱序列涵盖多个观测时间维度, 在执行分类任务时, 不仅需要考虑每个维度内部的时序特征, 也需要考虑多个维度之间的关联特征。 多维光谱序列与病害分类的数学形式表达如下
1.3.1 一维光谱序列
由n个有序的观测值ri|1≤ i≤ n组成的序列R={r1, r2, ..., rn}被称为长度为n的一维光谱序列, n=360。
1.3.2 多维光谱序列
由d个一维光谱序列组成的序列集合M={R1, R2, ..., Rd}被称为维度为d的多维光谱序列, 每个维度序列的长度均为n, d∈ {2, 3, 4, 5, 6, 7}。 多维光谱序列M中第i维序列的第j个观测值用ri, j表示。
1.3.3 基于多维光谱序列病害分类
给定包含m个实例的训练集S={M1, M2, ..., Mm}, 其中每个实例M都是一个长度为n维度为d的多维光谱序列, m=100。 训练集中的实例都属于C个类别之一, C∈ {患病, 健康}。 多维光谱序列分类的目标是在训练集S中学习多维光谱序列观测值到所属类别的映射, 即叶片是否患病。
1.3.4 光谱子序列
给定一个长度为n的光谱序列T, 从第i个观测值起, 截取w个连续观测值组成的新序列{ti, ti+1, ..., ti+w-1}被称为光谱序列T的子序列, 其中1≤ i≤ n-w+1。
本试验数据共有7个特征维度, 分别对应番茄叶片染病灰霉病菌的第1天至第7天, 为了实现番茄灰霉病病害的早期检测, 分别制作包含2~7个特征维度的光谱序列数据。 例如特征维度为3时, 156组实验样本只包含实验叶片染病第1天至染病第3天和相同观测日期的对照叶片数据, 其他维度同理。
1.3.5 多维关联光谱序列
维度随机选择, 即在构建每棵决策树时随机选择D维光谱序列中的d个维度, D=7, 生成初始光谱序列, 从而降低计算复杂度, 同时增加决策树之间的差异性。
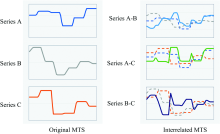
为了提取初始光谱序列中各个维度之间的关联特征, 计算初始序列中任意两条序列之间对应属性点的差值, 生成与初始序列长度相同的关联光谱序列, 初始序列TA与TB之间的关联序列TI的计算公式如式(1)
初始序列维度为d时, 转换后的关联序列维度为d'=d(d-1)/2。 图5(a, b)分别展示了d=3时初始序列转换为关联序列的过程。 建模时, 在平衡样本维度与计算开销情况下, 序列维度d取值为4, 即样本维度≥ 4时, 随机选取其中4维参与决策树的构建, 样本维度< 4时则用原维度。
建立分类器时, 直接采用光谱序列整体的相似性进行分类的方法时间复杂度较高, 因此为减小模型复杂度, 提取代表光谱序列局部辨别性的特征进行分类以缩减模型运算规模。 SAX-VSM算法(the symbolic aggregate approximation-vector space model)采用符号聚合近似估计(symbolic aggregate approximation, SAX)技术将时间序列转换到时域空间, 生成单词特征; BOSS(the bag of SFA symbols)算法采用符号傅里叶近似(symbolic Fourier approximation, SFA)技术将时间序列转换为频域空间的单词特征集合。 该两种符号化方法允许在离散后的符号表示上定义距离, 运行机器学习算法, 同时产生与对原始数据进行操作的算法相同的结果。 该性质使得该两种方法在降维的同时还能够保留原始序列的大体形状, 因此在各类时间序列任务中被广泛应用。
1.4.1 光谱序列符号化
给定一个长度为n的光谱序列T, 利用光谱序列符号化技术将序列T或其子序列转换为离散符号组成的字符串, 这个过程被称为光谱序列符号化。
1.4.2 SAX方法符号化光谱序列
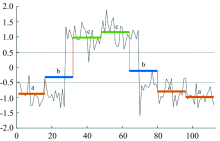
给定长度为w的光谱序列或其子序列T、 字母表大小a、 单词长度l, 将序列分为等长的l个子序列, 分别计算每个子序列中各个点的平均值, 基于高斯分布将平均值划分为a个区域, 每个区域对应一个字符, 将平均值序列离散化为对应字符, 得到序列T对应的字符串, 这个过程就是SAX方法。 图6展示了采用SAX方法将长度为112的序列转换为长度为7的字符串的过程。
1.4.3 SFA方法符号化光谱序列
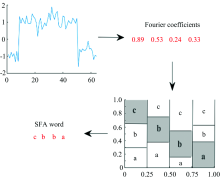
SFA方法与SAX方法类似, 区别在于SFA方法不计算序列的平均值, 而是对序列进行傅里叶变换后将傅里叶系数离散处理, 为每个系数计算合适的离散化分割点。 图7展示了采用SFA方法将长度为64的序列转换为长度为4的字符串的过程。
随机森林能较好地学习多个特征维度之间的潜在关系, 且复杂度较低、 抗干扰能力强, 因此常被用来处理高维度数据。 例如时间序列森林(time series forest, TSF)算法采用时间序列的间隔特征作为决策树的节点, 构建随机森林进行分类。 但这类算法只能提取到一条时间序列上的辨别性特征, 未考虑到多个维度之间相关性。 因此结合作物光谱曲线连续性, 本工作选择使用一种用于多维时间序列分类的随机森林算法作为识别作物病害的分类器。 在构建随机森林中的每棵决策树时, 首先随机选择多维光谱序列中的d条维度作为初始序列, 以增加决策树之间的差异性并降低时间复杂度; 然后将初始序列转换为与原序列长度相等的关联光谱序列, 从而提取光谱序列中多个维度之间的关联特征。 通过SAX方法和SFA方法, 提取初始序列与关联序列的时域与频域特征。 将转换后的光谱序列特征集合作为决策树的输入。 在每个结点为每个类别选择代表特征, 以待预测光谱序列特征与代表特征之间的相似性作为决策树的分支依据。 在独立构造每棵树之后, 计算每棵树的权重以进行加权分类, 实现番茄灰霉病识别的目标。
1.5.1 随机森林模型构建
借鉴邻近森林[11]的思想, 按照待分类实例与每个结点的代表实例之间的相似度进行结点分割。 首先在决策树的每个结点中随机选择多个代表不同类别的光谱序列字典特征实例, 然后计算当前结点的数据集中各个实例与各个代表实例的余弦相似度, 将与某一代表实例最相似的实例划分到该代表实例所属的结点分支。 不断重复这个划分过程, 直到某一结点中的全部实例都属于同一个类标, 则将这一结点作为叶子结点。 算法1展现了决策树的具体构建过程, 重复这一过程构建多颗决策树生成随机森林。
算法1 决策树构建算法buildMTSTree(S)
输入: 符号化的多维光谱序列数据集S
输出: 决策树根结点T
01. IF S中的实例都属于同一个类
02. 将该结点设为叶子结点, 结点类标为S中任一实例的类标
03. END IF
04. 创建结点T与实例集合数组E[]
05. FOR 0≤ i< S. numClasses
06. E[i]=Random(Si), Si为数据集S中类标为i的实例集合
07. END FOR
08. FOR e∈ E
09. 将与实例e距离最近的实例放入集合Se中
10. t=buildMTSTree(Se)
11. Se={s∈ S|argmindist(s, e)}在当前树结点T中添加分支(e, t)
12. END FOR
13. RETURN T
1.5.2 加权分类过程
传统的随机森林算法采用多数投票法决定分类结果, 这种方法操作简单, 但在一定程度上忽略了决策树之间的差异。 所采用的加权方式, 根据每棵决策树的结构, 计算其权重, 可以反映出当前决策树随机选择的维度、 特征提取等是否适合当前数据集, 从而提高分类准确率。
决策树的原理可以理解为通过某些特征选择方式, 将训练数据集划分为几个部分, 使得划分后的数据子集相比与划分之前, 具有更高的纯度, 即划分后的数据子集中各示例所属的类别不确定性更小。 因此在度量决策树的权重时, 可以通过度量决策树中每个结点划分前后数据集的纯度来实现。 本文选择了计算方式较为简单的基尼指数。 基尼指数也被称为基尼不纯度, 表示在实例集合中任意一个实例在分类时被分到错误类标的概率, 基尼指数的数值越小说明实例集合中实例分类错误的概率越小, 即实例集合的纯度越高。 其计算公式如式(2)
式(2)中, pk表示任意一个实例的类标为k的概率, K为实例集合中所有存在的类标数。
每个决策树的结点包含多个分支, 因此计算结点权重时需要分别计算每个分支的基尼指数。 然后将每个分支的基尼指数乘以每个分支的数据子集占结点总数据集的比例[式(3)], 累加起来得到结点的权重[式(4)]。 将决策树中所有结点的权重加起来, 作为整棵决策树的权重。 这时决策树的权重数值越小, 说明这棵决策树的总体划分效果越好, 在随机森林中占的投票权重应该越高, 因此采用归一化指数函数[式(5)]处理得到的权重值。 权重的计算公式如式(3)— 式(5)
式中, wt为最终得到的决策树权重, p为决策树的结点数, n为当前结点的分支数。
在分类阶段, 对于单棵决策树, 待分类实例首先按照当前决策树的符号化方式将多维光谱序列转换为局部辨别性字典特征, 然后从决策树的根结点开始, 选择与待分类实例最接近的代表实例所属的分支, 重复此过程直到到达叶子结点, 将叶子结点类别作为待分类实例的类别。 最后, 通过加权投票得到最后的分类结果, 实现基于光谱序列的番茄灰霉病识别。
未接种番茄灰霉病毒的植株在整个试验期间保持健康, 接种后的植株没有症状的情况下, 经过一段潜伏期后出现典型症状。 灰霉病的发病症状随着接种后病菌培育时间延长逐渐显著, 染病4 d后, 叶片染病从叶尖或叶缘开始, 发生不定形的湿润状、 灰褐色病斑。 染病6 d后, 小病斑逐渐发展成湿腐, 并长出一层鼠灰色茸毛状的霉层, 此为病菌的分生孢子梗和分生孢子。 按照可见光条件下的病程发展, 染病1~4 d划分为肉眼不可见, 染病5~7 d分别划分为肉眼可见颜色变化、 肉眼可见小病斑、 肉眼可见明显病斑。
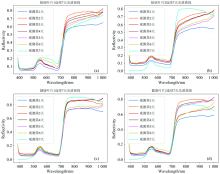
对比健康叶片与染病叶片的光谱曲线(图8), 染病叶片的光谱曲线在550~700和800~1 000 nm两个区间内均与健康叶片呈现不同特点与差异, 该差异不仅体现在单一波段上, 更体现在曲线整体的变化趋势上。 在550~700 nm波段区间内, 染病叶片的光谱反射率之间不密集[如图8(a, b)], 呈现分散的特点, 而健康叶片的较为紧凑, 如图8(c, d)。 在800~1 000 nm波段区间内, 染病叶片光谱曲线趋势形成带有正斜率向上延伸的曲线, 健康叶片呈水平波动或略有抬升的走向特点。 该差异特点为基于光谱曲线整体变化趋势特征的早期灰霉病识别提供了基本条件。
为验证模型从样本中识别出染病叶片的能力, 将健康叶片样本作为正例, 7个不同染病阶段的样本叶片分别作为反例, 建立基于单维光谱原始序列的早期番茄灰霉病检测模型。 模型的参数选择如表1所示。 根据预实验结果, 在符号化算法SAX和SFA的字典特征提取中, 字母表大小a的取值范围为3~5, 单词长度l的取值范围为3~6, 滑动窗口大小w的取值范围为光谱序列长度的20%~60%, 且以减少随机波动干扰增强鲁棒性为目的, 以上3个参数的数值随机选择参与序列符号化的构建。
| 表1 符号化方法与随机森林模型构建的参数 Table 1 Parameters of symbolic methods and weighted random forest model |
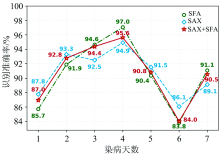
为验证模型稳定性, 基于SFA, SAX与SAX+SFA三种符号化方法建模, 重复实验20次, 将20次实验的识别率求平均得到结果如图9所示, 其中识别准确率=
基于单维原始光谱序列的番茄灰霉病检测模型的分类准确率随着疾病严重程度的增加先增加后降低。 在肉眼可见染病叶片明显颜色变化前(染病1~4 d), 分类准确率在三种符号化方法中均先呈上升趋势, 并且在染病第4天达到顶峰, 识别准确率最高97%。 然而随着染病程度加深, 基于单维原始光谱序列的模型识别效果出现回落, 染病第5天和第7天落回到90%附近, 染病第6天的识别率降到最低, 仅有83.8%。 分析此现象的原因是染病5~7 d的叶片光谱曲线产生随机波动[1], 模型识别准确率受到影响, 因此基于单维光谱序列的分类器无法作为番茄灰霉病害的早期检测模型。
为解决染病严重时模型识别准确率偏低的问题, 从染病第1天开始, 将单维光谱序列依照染病进程顺序, 分别组合成1~7个特征序列的多维度数据, 离散成局部辨别特征后, 基于该特征建立分类模型, 以此实现番茄灰霉病早期检测, 模型参数与表1设置相同。 结果如图10(a, b, c)所示, 基于多维光谱序列模型在测试集上的识别准确率逐步上升, 对染病2天到染病7天后发病严重等6个阶段的识别均超过90%, 相较于基于单维光谱原始序列的模型均有提高。 在5维序列时达到最高识别率99%, 并且在维度6和7中未发生过度回落, 准确率依然保持在98%左右, 表明基于多维光谱序列特征能有效避免光谱干扰性波动造成的识别效果不稳定的情况。
对模型的三种符号化方法进一步分析, 基于原始光谱序列的分类器检出率整体优于基于关联序列和组合序列的分类器; 基于关联光谱序列的模型检测效果在三种序列表达中的表现最差, 整体准确率不高于80%; 基于组合光谱序列的模型识别准确率介于其他两种序列表达方法之间, 分布于86.5%~94.9%。 随着序列维度的增加, 基于关联序列的识别模型识别效果随时间维度增加提升较大, 最大增幅37.7%, 并均在维度为7时达到最高点。 说明维度特征对关联序列表达尤为重要, 叶片光谱特征信息越丰富, 关联光谱信息越能表达番茄灰霉病的健康信息与患病信息。 然而, 在低维度光谱序列中, 原始序列则能最大程度保留番茄灰霉病的光谱特征并有效表达, 在稳定性和识别准确率中均优于其他两种序列表达方式。
对比基于多维光谱序列和单维光谱序列在最优符号化方式下模型的准确率(图11), 在光谱维度为2~3时, SDSS-SAX-SFA-WRF模型检测效果略优于MDSS-SAX-SFA-WRF模型, 差异基本控制在1.5%以内。 然而, 在光谱维度为4(染病第4天)后, MDSS-SAX-SFA-WRF番茄灰霉病早期检测模型则相较SDSS-SAX-SFA-WRF模型一直保持优势, 维持不低于98%的识别准确率, 走势较平稳。 即使在染病第1天, MDSS-SAX-SFA-WRF模型同样达到87.4%的检测准确率, 将作物病害的检测时间大大提前。 随着观测维度增加, MDSS-SAX-SFA-WRF模型的识别准确率每日增幅3%, 直至染病第5天(肉眼可见颜色变化)达到最高。 然而即使在该阶段和维度6~7 d时, 植保人员依然无法仅凭肉眼通过病叶表面颜色的变化对植株所患疾病确诊, 仍需待病情进一步发展或从植株中摘取叶片进行化学鉴定, 因此在该阶段, 作物病害检测模型的准确识别依然发挥重要作用。
借鉴多维时间序列的思想, 将多个观测日的光谱曲线累积形成多维光谱序列, 为减小模型计算量, 采用符号化方法提取光谱序列的局部辨别特征, 并且依据该累积局部辨别特征建立加权随机森林模型MDSS-SAX-SFA-WRF, 学习健康叶片与染病叶片在不同观测维度间的差异信息表达, 实现番茄灰霉病的早期检测, 同时在数据维度逐渐增加情况下, 能够在吸收新维度特征的同时最大化保存之前观测维度的辨别特征, 并将新旧特征有效结合形成累积辨别特征, 将所观测到多维度的光谱曲线特征融合确保模型识别的精准度。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|