作者简介: 李 伟, 1988年生, 山西农业大学农业工程学院博士研究生 e-mail: jamesallenlw@163.com
有机磷农药毒死蜱是目前农业生产中使用最广泛的农药之一, 但有机磷农药过度使用导致的农药残留却给自然环境和人类生命健康造成严重威胁, 因此, 开发一种快捷、 准确、 经济的毒死蜱农药在农产品表面残留的直接检测方法意义重大。 配制4组不同体积浓度(1:200, 1:500, 1:800, 1:1 000)的毒死蜱农药溶液, 对照组为纯净水, 分别浸泡甘蓝叶片3 min, 每组采集30个叶片样本, 5组共计150个样本。 采用可见近红外光谱仪获取其谱图信息, 然后开展不同浓度毒死蜱农药在甘蓝叶片上残留的可见近红外光谱定性分析研究。 建模时, 将每组数据中24个样本, 5组共计120个样本作为建模训练集, 剩下每组6个样本, 5组共计30个样本作为预测集。 鉴于甘蓝叶面不平整、 皱褶较多, 叶片颜色深浅不一等因素会给近红外光谱分析带来干扰, 给预测模型的建立增加难度, 提出一种光谱全波段平均分组积分(求和)预处理方法, 将光谱波段平均分成 n组, 再对分组后每组数据积分求和, 用预处理后的数据训练BP神经网络。 实验表明, 光谱全波段平均分组积分(求和)预处理方法, 对光谱反射率一阶导数(FD)且分组数为25的神经网络训练效果最好, 建模集识别准确率为97.50%, 预测集识别准确率为96.67%, 建模效果优于通常采用的提取光谱敏感、 特征波段建模方法(建模集识别准确率为91.67%)。 光谱全波段平均分组积分预处理方法在保留光谱数据更多特征波段的同时探索更多潜在敏感波段, 能够降低光谱数据维度, 减小单个光谱数据噪声对建模效果的影响, 选择合适的分组数 n, 能取得较好的建模预测效果。
Chlorpyrifos is one of the most widely used organophosphorus pesticides (OPs) in agricultural production. However, pesticide residues caused by excessive OPs pose a serious threat to the natural environment and human life and health. Therefore, it is of great significance to develop a rapid, accurate, convenient, and economic method for directly detecting OPs residues in agricultural products. Four groups of chlorpyrifos pesticide solutions with different volume concentrations (1:200, 1:500, 1:800, 1:1 000) were prepared, the control group was treated with pure water.Cabbage leaves were soaked in chlorpyrifos pesticide solutionsfor 3 minutes, 30 leaf samples were collected from each group, and 150 samples were collected from 5 groups. The spectrum information of Chlorpyrifos in cabbage leaves was obtained by visible near-infrared spectroscopy (NIR), and the qualitative analysis of chlorpyrifos pesticide residues in cabbage leaves was carried out. In modeling, 24 samples in each group, 120 samples of 5 groups are taken as modeling training set, 6 samples in each group and 30 samples of 5 groups are taken as prediction set. The near-infrared spectrum analysis will be interfered with by factors such as uneven leaf surface, more wrinkles and different color of cabbage leaves, which makes the establishment of prediction model more difficult. In this paper, an all-band average grouping integration preprocessing method is proposed. The spectral bands are averagely divided into n groups, and then each group of data is integrated as new data for neural network modeling. The experimental results show that the all-band average grouping integration preprocessing method proposed in this paper has the best modeling effect using the spectral reflectance first derivative (FD) when the group number is n=25. The modeling set recognition accuracy is 97.50%, and the prediction set recognition accuracy is 96.67%. The modeling effect is better than the commonly used spectral sensitive and characteristic band modeling method (with modeling set recognition accuracy 91.67%). The all-band average grouping integration preprocessing method can retain more characteristic bands of spectral data and reduce the dimension of spectral data, reducing the impact of single spectral data noise on the modeling effect. Selecting the appropriate grouping number could achieve good modeling and prediction effect. The results of this study can provide a reference for the application of visible near-infrared spectroscopy in the detection of chlorpyrifos pesticide residues.
甘蓝(Cabbage), 又名卷心菜, 属于十字花科, 其茎叶营养丰富、 口感清脆深受人们喜爱, 是一种非常重要的茎叶类蔬菜。 为防止病虫害、 提高农作物产量, 蔬菜生产过程中需要多次喷洒农药, 甘蓝叶片层层包裹, 容易形成农药残留和累积[1]。 毒死蜱(Chlorpyrifos)是一种结晶体有机磷农药, 是目前农业生产中应用最广泛的有机磷农药之一[2, 3]。 有机磷农药的过度使用造成的农药残留问题会破坏生态环境, 给人类生命健康造成严重威胁[4]。 采用近红外光谱进行农作物无损检测已经成为国内外学者研究热点问题[5, 6, 7, 8, 9]。
目前对可见近红外光谱检测水果、 蔬菜等农产品表面农药残留的研究主要包括两类, 即农药残留种类鉴别[5, 6]和农药微量浓度残留定性判别[7]。 孙俊[5]等通过连续投影算法选出光谱的10个特征波长, 用支持向量机(SVM)与10折交叉验证法, 建立了桑叶农药残留的近红外光谱检测模型, 在SVM算法中引入自适应提升算法获得更好的建模效果, 结果表明Ada-SVM算法预测准确率达97.78%。 陈淑一[6]等将对比主成分分析算法应用到近红外光谱数据降维模型, 结果表明对比主成分分析降维算法能够识别水果表面是否喷洒农药的特征。
近红外光谱数据对农药残留浓度定性判定过程中有很多干扰因素: 不同果蔬表面差异较大, 有些蔬菜叶面不平整, 比如甘蓝叶片褶皱多, 会对光谱反射率产生干扰, 另外甘蓝叶片颜色深浅不一, 比如甘蓝外层叶片颜色深, 而内层叶片颜色浅, 也会干扰光谱数据。 另一方面, 大多关于近红外光谱的研究都在提取分析物质的特征敏感波段, 不同文献对毒死蜱提取的敏感波长、 使用的敏感波段并不完全相同[10]。
本文提出一种全波段平均分组积分光谱数据预处理方法: 选择可见-近红外光谱数据全部波段(350~2 500 nm), 在保留多个敏感光谱波段的同时以探索更多潜在敏感波段, 将全波段平均分为n组, 分别对每组光谱数据积分求和, 以达到对光谱数据降维压缩的目的, 而且极大消除了单个波长光谱数据的随机误差对建模效果的影响。
以甘蓝叶片为检测对象, 将甘蓝叶片在不同体积浓度毒死蜱农药中浸泡3 min, 后置通风处晾晒3 h, 用可见近红外光谱仪获取甘蓝叶片谱图信息。 经全波段平均分组积分预处理后的数据用于训练神经网络。 一般而言, 神经网络输入数据的维度要小于神经网络的训练样本数, 全波段平均分组积分预处理方法能够选择n方便的控制输入数据维度。 通过实验对比寻求最优的建模方案, 建立甘蓝叶片农药残留浓度定性判别预测模型。
毒死蜱农药配制: 毒死蜱农药是山东东远生物科技有限公司生产的45%浓度的毒死蜱乳液, 不同体积浓度的毒死蜱溶液由纯净水稀释而成, 分别稀释至200倍(1:200)、 500倍(1:500)、 800倍(1:800)和1 000倍(1:1 000), 对照组为纯净水, 将各组溶液分别标注为5, 4, 3, 2和1类。
从市场购买普通的甘蓝, 将甘蓝叶片裁剪成直径5 cm左右近似圆形, 共150个叶片样本, 分为5组, 每组30个样本。 首先将裁剪好的近圆形叶片使用纯净水做超声波清洗处理, 然后分别将各组叶片在1, 2, 3, 4和5类溶液中浸泡3 min。 后将甘蓝叶片移至通风处晾晒3 h, 至毒死蜱农药溶液的水分蒸发干。
使用ASD公司生产的FieldSpec3光谱仪采集光谱数据: 光谱仪光源为卤素灯, 光源与甘蓝叶片距离15 cm, 卤素灯光与水平线呈45° 夹角。 光谱采集范围350~2 500 nm, 光谱分辨率为1 nm, 每个样本扫描3次求平均值, 光谱采集在实验室室温[(23± 0.5) ℃]下进行。 原始光谱数据在ViewSpecPro软件中进行预处理, 包括求原始光谱数据平均值, 拼接校正, 分别求原始光谱数据的一阶导数和二阶导数。 处理后的数据导出, 使用MATLAB2011a进行进一步的数据处理和建模分析。
设原始光谱反射率为
式(1)中, 光谱反射率f(λ )是光谱波长λ 的离散函数, 不同波长λ 对应的反射率分别为f1, f2, …, fj, 光谱反射率波长总数为j。
光谱反射率的一阶导数可以表示为
光谱反射率的二阶导数可以表示为
将整个光谱波段平均分成n等份, 则每一等份的光谱波长数为$m=\lfloor j/n \rfloor$, 分别对每一组分积分求和得到处理后的数据, 原光谱反射率(R)、 光谱反射率一阶导数(FD)、 光谱反射率二阶导数(SD)的平均分组积分预处理计算过程分别如式(4), 式(5)和式(6)所示
其中, Σ (n), Σ '(n), Σ ″(n)分别表示光谱反射率(R)、 光谱反射率一阶导数(FD)、 光谱反射率二阶导数(SD)做全波段平均分组积分处理后的数据, 它们均为n维数组。 Σ 1, Σ 2, Σ 3, …, Σ n表示Σ (n)数组中的n个元素; Σ '1, Σ '2, Σ '3, …, Σ 'n表示Σ '(n)数组中的n个元素; Σ ″1, Σ ″2, Σ ″3, …, Σ ″表示Σ ″(n)数组中的n个元素。
分别将Σ (n), Σ '(n), Σ ″(n)作为神经网络的输入, 建立k层神经网络模型如下(以Σ (n)为例):
输入层
隐藏层一
隐藏层二
输出层
其中mi表示第i层网络的节点个数, Y(i)表示第i层网络的输出, 也表示第i+1层网络的输入。
定义第k层神经网络的激活函数为h(k), 每一层神经网络的权重矩阵W和偏置向量b定义如下
那么第k层神经网络的输出Y(k)的计算式(14)所示
其中net(k)表示第k层神经网络的神经元的输入向量, 也表示第k层权重矩阵W(k)与第(k-1)层神经网络的输出乘积再加上第k层偏置矩阵b(k)。 神经网络输出层Y即为甘蓝叶片毒死蜱残留浓度的判定标准。
神经网络模型是通过模拟动物神经网络特征的一种建模算法, 适用于复杂的非线性多维数据建模分析模型[11, 12]。 选择典型BP神经网络, 设置输入层、 1个隐藏层(50个节点)、 输出层。 将预处理后的n维数据作为BP神经网络的输入, 各溶液组的类别标号作为神经网络的输出, 神经网络的输出层Y为一维数据, 且数值只能取1, 2, 3, 4和5。 将各组数据中前24个样本, 5组共计120个样本作为建模训练集, 剩下每组6个样本, 5组共计30个样本作为预测集。
神经网络模型训练结果最主要的评价指标就是模型对建模集的识别准确率p, 定性判别时识别准确率越高, 建模效果越好。 模型识别准确率的计算公式如式(15)所示
式(15)中, Yi是所建模型对样本组别的预测值,
图1是不同浓度毒死蜱残留甘蓝叶片的平均光谱曲线。 从图1中可以看到, 不同组别的甘蓝叶片原始平均光谱反射率曲线变化规律相近, 平均光谱反射率曲线在数值上略有差异, 但整个光谱曲线在多个局部有细微随毒死蜱农药残留浓度变化特征明显的波段, 如图2所示。
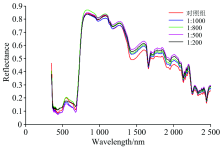 | 图1 不同浓度毒死蜱残留甘蓝叶片平均光谱曲线Fig.1 Average spectra of cabbage leaves with different chlorpyrifos concentrations |
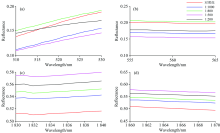 | 图2 不同浓度毒死蜱残留甘蓝叶片平均光谱曲线特征明显波段Fig.2 Obvious characteristics bands of average spectra of cabbage leaves with different chlorpyrifos concentrations residues |
图2是不同浓度毒死蜱平均光谱曲线的四个局部细节, 分别为510~530, 555~565, 1 830~1 840和1 860~1 870 nm等四个特征明显波段。 在这些特征明显波段内, 光谱反射率(R)曲线的斜率随农药残留浓度的变化有明显特征。 这种特征在光谱反射率曲线的一阶导数(FD)曲线中表现得更加明显, 如图3所示。
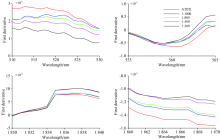 | 图3 不同浓度毒死蜱残留甘蓝叶片平均光谱曲线一阶导数特征明显波段Fig.3 Obvious characteristics bands of first derivative average spectra of cabbage leaves with different chlorpyrifos concentrations residues |
图3是原始平均光谱反射率一阶导数(FD)曲线的四个局部特征明显波段。 如图3所示, 在特征明显波段510~530 nm, 随毒死蜱农药残留浓度的增大光谱曲线的斜率变小, 光谱反射率的一阶导数(FD)变小; 而在波段555~565, 1 830~1 840和1 860~1 870 nm等特征明显波段, 随农药残留浓度的升高原始平均光谱反射率(R)曲线的斜率增大, 光谱反射率一阶导数(FD)增大。
从光谱反射率(R)曲线中共找到四段随农药残留浓度变化特征明显的光谱波段, 提取特征明显波段分别为510~530, 555~565, 1 830~1 840和1 860~1 870 nm, 共计54个特征明显波长。 这些特征明显波段与之前的文献报道有相近之处[10]。 从光谱反射率(R)曲线中发现更多特征明显波段, 将这54个特征明显波长作为神经网络的输入, 训练神经网络, 统计神经网络建模预测准确率如表1所示, 可见, 光谱反射率一阶导数(FD)提取的特征明显波段建模效果最好, 建模集识别准确率可以达到91.67%, 效果良好。
| 表1 特征明显波段建模效果 Table 1 Modeling effects of obvious characteristics bands |
提取随浓度变化特征明显波段虽然能找到和农药残留浓度直接相关的光谱波段, 但光谱数据庞杂, 有些变化明显或敏感波段难以从光谱曲线中直接发现。 为此提出一种新的光谱全波段平均分组积分预处理方法, 将全波段光谱平均分为n组, 后对每一组进行积分求和处理, 组成新的数据, 进行神经网络建模。 具体算法如1.3中所描述。 光谱仪光谱采集数据光谱范围为350~2 500 nm, 总波长数j=2 151, 处理后的数据维度为n。 n取不同数值(10, 15, 20, 25, 30, 35和40)时, 对应不同的数据维度m如表2所示。
| 表2 全波段平均分组积分预处理建模效果 Table 2 All-band grouping integration preprocessing modeling effects |
分别选择原光谱反射率(R)、 光谱反射率一阶导数(FD)和光谱反射率二阶导数(SD)进行全波段平均分组积分预处理, 处理后的n维数据作为训练神经网络输入, 农药残留叶片样本组标号(标号1, 2, 3, 4, 5)作为神经网络的输出。 神经网络输出Y的取值通过式(16)确定
如表2所示, 整体而言, 光谱反射率一阶导数(FD)全波段平均分组积分(求和)预处理, BP神经网络模型训练效果最好, 建模集识别准确率较高, 其次是光谱反射率(R), 光谱反射率二阶导数(SD)建模效果最差, 建模集识别准确率最低。 对光谱反射率(R)、 光谱反射率一阶导数(FD)、 光谱反射率二阶导数(SD)预处理建模识别准确率都随平均分组数n的增大呈先增大后减小的趋势, 其中光谱反射率(R)在分组数为30时建模效果最好, 建模集识别准确率最高为81.67%。 光谱反射率一阶导数(FD)和二阶导数(SD)均在分组数为25时取得最好建模效果, 建模集识别准确率最高分别为97.50%和73.33%。 全波段平均分组积分预处理方法选择全部光谱波段范围, 在保留特征明显光谱波段的同时试图从原始光谱数据中探索更多潜在特征明显和敏感波段; 光谱数据分组是对光谱数据的一种压缩, 能够有效降低神经网络输入数据维度; 光谱数据积分将组内所有光谱数据求和, 能够有效地减小单个光谱数据随机误差对建模带来的干扰; 调整分组数n的大小能够找到最佳分组效果。
以普通甘蓝为研究对象, 将甘蓝叶片在不同体积浓度毒死蜱农药溶液中浸泡3 min, 经过3 h的通风晾晒。 获取甘蓝
叶片可见近红外光谱信息, 通过全波段平均分组积分预处理后建立神经网络模型, 与选取特征明显波段建模效果对比, 结论如下:
(1)有毒死蜱农药残留的甘蓝叶片的光谱反射率曲线中发现四个与毒死蜱农药溶液体积浓度相关的特征明显波段, 分别为510~530, 555~565, 1 830~1 840和1 860~1 870 nm。 特征明显波段光谱反射率一阶导数(FD)曲线随农药残留浓度变化特征最显著。
(2)分别提取光谱反射率(R)、 光谱反射率一阶导数(FD)和光谱反射率二阶导数(SD)光谱特征明显波段进行神经网络建模训练, 对建模集识别准确率分别为74.17%, 91.67%和70.83%, 其中光谱反射率一阶导数(FD)训练效果最好, 这与光谱反射率一阶导数(FD)曲线随毒死蜱农药残留浓度特征最显著的结果一致。
(3)分别对原光谱反射率(R)、 光谱反射率一阶导数(FD)、 光谱反射率二阶导数(SD)进行光谱全波段平均分组积分(求和)预处理后建模, 其中, 光谱反射率一阶导数(FD)全波段平均分组积分预处理后建模效果最好, 其中当分组数为25时效果最好, 最好建模效果对建模集的识别准确率为97.50%, 对预测集识别准确率可以达到96.67%。
(4)对不同的光谱数据(R, FD, SD)做平均分组处理能极大程度压缩光谱数据, 分组后的数据做积分求和处理能够降低单个光谱波长随机噪声对建模效果的干扰, 在保留光谱数据特征明显波段的同时又进一步探索更多在光谱反射率曲线上表现不明显的潜在敏感波段, 调整参数n能取得良好的建模效果。
通过可见近红外光谱数据对甘蓝叶片毒死蜱农药残留浓度进行定性检测判定, 快速, 有效, 经济, 本研究提出的全波段平均分组积分预处理方法能有效提高对甘蓝叶片毒死蜱残留浓度的判定准确度, 对可见近红外光谱检测毒死蜱农药在农产品表面残留浓度具有重要的参考价值。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|