作者简介: 桂江生, 1978年生, 浙江理工大学信息学院副教授 e-mail: jsgui@zstu.edu.cn
为降低大豆食心虫对大豆产量以及品质的影响, 实现对大豆食心虫虫害的快速检测, 提出了一种基于三维关系网络小样本元学习(3D-RN)模型的大豆食心虫虫害的检测方法。 首先分别对附着虫卵的, 附着食心虫幼虫的, 被啃食的及正常的大豆各20颗进行高光谱图像采集, 提取感兴趣区, 建立基于高光谱图像的3D-RN模型。 最终模型的正确率达82%±2.50%。 对比与模型无关的元学习和匹配网络元学习模型, 3D-RN模型能够充分度量样本特征间的距离, 识别效果大大提升。 研究表明, 基于高光谱图像的3D-RN模型能够在少量样本情况下实现对大豆食心虫虫害的检测, 将小样本元学习与高光谱结合的方法为虫害检测提供一种新思路。
In order to reduce the influence of leguminivora glycinivorella on soybean production and quality, and to realize the rapid detection of leguminivora glycinivorella, this paper proposed a leguminivora glycinivorella detection model based on 3D-Realtion Network (3D-RN) model. Firstly, collect the hyperspectral images of 20 soybeans that are attached to eggs, larvae, gnawed and normal soybeans, respectively, and extract the region of interest (ROI) to establish a 3D-RN model based on hyperspectral images. The accuracy of the final model reached 82%±2.50%. Compared to the Model-Agnostic Meta-Learning (MAML) and Matching Network (MN) models, the 3D-RN model can fully measure the distance between sample features, and the recognition effect is greatly improved. Thus, this research shows that the 3D-RN model based on the hyperspectral image can detect leguminivora glycinivorella in a small number of samples. The method of combining few-shot meta-learning with hyperspectral provides a new idea for pest detection.
大豆是世界上最重要的油料作物和人类最主要的植物蛋白源, 在两市供给和农业生产中占重要地位[1]。 然而, 在大豆生长以及存储过程中, 由于病虫害导致大豆的产量下降。 中国大豆害虫达400余种, 虫害一直是影响大豆产量和质量的重要因素[2]。 大豆在生长存储过程主要面临的虫害主要有大豆食心虫、 大豆蚜虫以及豆象科害虫等。
目前的作物病虫害检测方法主要有: 人工感官判断, 生物化学方法[3, 4, 5], 图像处理和光谱数据检测[6, 7, 8]。 其中, 人工感官判断具有一定的局限性和主观性。 生物化学方法对检测样本要求高, 且耗时长、 成本高以及不利于大规模操作。 图像处理需要采集大量的样本数据支持, 耗费大量的人力以及时间等。 光谱数据的检测方法只对样本的光谱维度进行分析, 缺少空间维度的数据分析, 导致检测效果不佳。 随着图像空间和光谱结合的高光谱技术的快速发展, 高光谱在农作物病虫害检测的应用越来越广泛。 Zheng[9]等提出了基于选取的植被指数的损失分量与芦苇绿叶干重量损失之间的关系建立了蝗虫损失估计模型。 李翠玲[10]等搭建了高光谱成像系统, 利用判别分析方法建立番茄叶片斑潜蝇虫害的判别模型。 Lin[11]等利用R-CNNs开发了一个基于知识的植物病虫害识别系统。 表明高光谱结合神经网络的方法检测虫害具有较为光明的前景。
目前大多数高光谱图像采用卷积神经网络进行分类。 但是深度学习需要采集大量数据, 针对这个问题, 本文提出了一种三维关系网络小样本元学习模型的大豆食心虫虫害检测方法, 通过高光谱技术, 利用元学习关系网络具有在少量标记样本下处理数据和三维卷积神经网络高效提取高光谱图像特征的特点, 采用基于高光谱建立大豆食心虫虫害的三维小样本元学习模型, 对大豆食心虫虫害进行检测, 旨在探索高光谱图像处理技术以及小样本元学习模型在大豆食心虫虫害检测方面的应用。
实验用的大豆样本以及大豆食心虫幼虫来源于实际农业机构。 将大豆食心虫幼虫放入温暖潮湿的箱子中等待大豆食心虫长成成虫后, 将20只成虫放入大豆中使其于大豆上产卵, 5 d后采集附着虫卵的大豆, 10 d后采集附着大豆食心虫幼虫的大豆, 30 d后采集被啃食的大豆。 分别对正常的大豆以及上述三种大豆拍摄高光谱图像, 每类样本数量为20颗大豆种子。
实验使用的高光谱成像系统如图1所示, 主要包括高光谱成像仪(Imperx IPX-2M30)、 CCD相机、 电控平移台、 4个150 W的卤素灯以及一台计算机。 其中采集的光谱范围为383.70~1 032.70 nm, 共包含256个光谱波段, 光谱分辨率为2.73 nm, 使用SpectraVIEW软件采集获得高光谱图像。 为避免环境光对采集到的图像产生影响, 整个试验采集过程在暗箱中完成。
1.3.1 光谱预处理及感兴趣区提取
在采集图像过程中可能会由于光照不均匀而对实验结果造成影响, 所以对采集到的图像进行黑白校正。 采集过程中为了降低物体漫反射, 样本不均匀, 基线漂移等问题, 避免噪声对实验结果的影响, 对大豆光谱维度数据进行Savitzky-Golay滤波处理, 使光谱更加平滑, 并保持光谱的形状和宽度不变, 对后续的建模和检测具有重要的意义。
高光谱图像由很多窄波段图像组成, 波段间的相关性比较大, 从而造成数据冗余, 造成大量的重复计算。 为了避免数据冗余的影响, 对大豆高光谱图像进行主成分分析法降维处理, 选取前30个主成分高光谱波段作为特征波段。 以样本为中心选取50×50像素的正方形区域作为感兴趣区域, 将数据转换成50×50×30的高光谱数据。
1.3.2 小样本元学习模型原理
小样本元学习模型有规定的训练模式, 小样本元学习包含元训练数据和元测试数据, 两者均包含支撑集和查询集。 给定一个N-way K-shot分类任务, 支撑集中含有N个类别, 且每个类别含有K个被标签的样本, 查询集中同样也包含N个类别以及未被标签的样本。
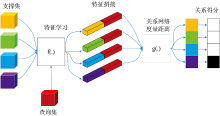
三维关系网络(3D-relation network, 3D-RN)[12]是一种基于度量的元学习方法, 训练好模型后无需对模型进行调整, 也能对新的类别进行分类。 3D-RN主要是训练一个卷积神经网络来学习距离度量的方式, 而不是采用线性度量方法来度量距离。
将所有数据分成多个任务p(T), 每个任务包含支撑集S={(xi, yi)
式(1)中, f(·)为三维卷积神经网络学习样本的特征, g(·)为3D-RN网络度量两个样本间的距离, C(·)将两个特征拼接。 3D-RN的网络结构如图2。
与模型无关的元学习(model-agnostic meta learning, MAML)[13]是一种基于模型的元学习方法, 通过训练一个模型, 使得这个模型能够最大程度的适应多分类任务, 即使面对未训练过的对象, 也不需要构建新的模型, 使用一样的学习模型也可以完成新任务的分类, 基本思想是寻找到对于模型更好的初始参数, 使得模型能够在该初始参数下快速高效的学习新的任务。
匹配网络(matching network, MN)[14]是一种基于度量的元学习方法, 训练好一个模型后, 不需要对模型做调整, 也能对新的类别即训练中未出现的类别进行分类, 基本思想类似于端到端的近邻分类器。
高光谱图像采用SpectraVIEW软件采集, 采集图像时的曝光时间为18 ms, 平台移动速度1.50 cm·s-1, 4盏卤素灯与平台的夹角为50°, 首先采集白板图像W和暗背景图像B, 并依次对大豆样本进行图像采集。 采集到的大豆样本如图3。
使用CAVE, iCVL和NUS数据集作为元训练数据集。 CAVE数据集是一个包含32个场景的哥伦比亚大学收集的多光谱数据集。 iCVL数据集是一个欧洲计算机视觉会议收集的高光谱数据集, 数据集覆盖了室内、 公园、 植物、 农村以及城市。 NUS数据集是一个包含一般场景和水果两大类的高光谱数据集。 本文采集的四类大豆样本数据作为目标测试数据集。 每个任务包含4个类, 每个类共包含20个样本, 分别对训练集包含每个类1个样本和5个样本两种情况进行研究。
3D-RN模型由特征学习模块、 特征拼接和关系网络组成, 特征学习模块使用的网络模型为ResNet18或Conv4。 ResNet18模型由3D卷积层、 BatchNormal归一化层、 Relu激活函数、 最大池化层、 常规残差模块和平均池化层组成。 其中第一层的3D卷积层的卷积核为7×7×7, 共包含8个常规残差模块, 常规残差模块中卷积层的卷积核为3×3×3, 最大池化层的卷积核为3×3×3, 损失函数为交叉熵损失函数。 Conv4模型由3D卷积层、 BatchNormal归一化层、 Relu激活函数和最大池化层组成, 共4个卷积层, 其中3D卷积层的卷积核为3×3×3, 最大池化层的卷积核为3×3×3。 关系网络由2个卷积层组成, 两层的卷积核均为3×3×3。 每轮迭代方式为: 卷积运算, 归一化操作, 再叠加两层全连接层, 激活函数为Relu。 最后使用均方误差损失函数输出关系得分。 MAML模型使用的网络模型为ResNet18或Conv4。 MN模型特征提取网络为ResNet18模型和Conv4模型, 训练数据使用的记忆网络为双向LSTM, 测试数据使用的记忆网络为LSTM, 距离度量网络使用余弦距离度量两个样本间的距离, 注意力分类网络采用softmax损失函数分类。 分类模型结果如表1所示。
| 表1 不同分类模型在4-way 1-shot和4-way 5-shot情况下的检测结果 Table 1 Detection results of different classification models in the cases of 4-way 1-shot and 4-way 5-shot |
结果表示, 基于度量的小样本学习模型表现得比较好, 其中3D-RN模型表现最好, 5-shot比1-shot表现更好。 3D-RN模型采用卷积网络度量查询集与支撑集中样本的距离, 这种非线性的度量方法能够更好地度量样本之间的距离, 且3D-RN模型不需要过低的学习率就能够得到很好的效果, 说明在少量样本下3D-RN模型需要的算力和迭代更小。 5-shot将支撑集中每类的5个样本的特征结合在一起能够更好地表示样本的特征, 从而使得分类的结果比1-shot更加优异。 本模型训练好后可以用于工业应用, 实际应用中不再需要训练, 直接完成预测任务, 耗时少, 精度高, 能够舍去大量的人工检测成本。
每个任务中每类样本数量也是个重要的超参数, 故分别对每类共含10, 20和30个样本使用3D-RN模型进行4-way 5-shot实验, 实验结果显示在10, 20和30个样本条件下, 准确率分别为69.27%±1.95%, 82.75%±2.50%和73.46%±1.44%。 表明样本数量太小使得模型无法充分学习导致准确率不高, 样本数量太大, 任务复杂度超出模型的表达能力, 导致准确率下降。
目前, 高光谱成像技术已经广泛应用于农业病虫害检测, 但是对于小样本检测仍然面临很大的困难。 利用高光谱图像技术采集正常大豆样本以及被大豆食心虫啃食的不同时期的大豆样本, 建立了基于高光谱成像的大豆虫害三维关系网络小样本元学习检测模型。 在三维关系网络模型中, 三维卷积神经网络充分提取高光谱图像特征, 卷积神经网络非线性距离度量模型, 更有效的对比样本特征, 因此, 与MAML和MN元学习模型对比, 3D-RN模型的检测效果更精确, 最终正确率在5-shot情况下正确率达到82.75%±2.50%。 在样本数量不够, 标记样本数量小的情况下, 本模型表现优异。 综上, 小样本学习模型能够在高光谱成像技术上应用, 解决实际问题, 并且可以通过改进卷积网络模型以及其他参数进一步提高正确率。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|