作者简介: 白雪冰, 1991年生, 中国农业大学信息与电气工程学院博士研究生 e-mail: 464161695@qq.com
随着居民生活水平的提高和对健康饮食结构的重视, 羊肉作为一种高蛋白且低脂肪和胆固醇的畜肉, 需求量逐年上涨。 根据国家统计局统计, 2012年—2019年我国畜肉产业中羊肉产量占比从6.27%上升到9.02%。 研究提出了一种基于二次迭代Monte Carlo(MC)算法剔除异常样本的羊肉硬度定量检测PLSR模型。 采用GaiaSorter高光谱分选仪的Image-λ-V10E-H相机采集羊肉样品400~950 nm的高光谱数据, Image-λ-N17E相机采集羊肉样品900~1 650 nm的高光谱数据。 首先, 对比分析了S-G平滑、 二阶求导、 多元散射校正(MSC)、 标准正态变换(SNV)等光谱预处理方法在消除噪声影响, 提高光谱分别率等方面的能力, 选取最佳光谱预处理方法。 然后, 在第一次MC抽样中, 计算所有样本预测误差均值和标准差的平均值, 以该平均值的2.5~3倍作为可疑样本阈值, 3倍作为异常样本阈值; 剔除异常样本, 保留并标注可疑样本, 进行第二次MC抽样, 以样本预测误差均值和标准差的3倍值为阈值进行异常样本二次剔除; 对第一次MC抽样中标注可疑样本进行二次检测。 最后, 对比分析了基于全波长建立的偏最小二乘回归(PLSR)模型和基于回归系数法(RC)提取的特征波长建立的PLSR模型。 研究结果表明, 所提出的二次迭MC算法可以准确判别可疑样本是否为异常样本, 有效优化样本集, 为建模提供良好的数据基础。 以MSC作为光谱预处理算法基于400~950和900~1 650 nm两段高光谱数据建立PLSR模型的RP2分别为0.947 2和0.978 3, RMSEP分别为47.789 9和30.590 1 g, 优于其他三种光谱预处理算法。 另外, 基于900~1 650 nm建立的PLSR模型明显优于基于400~950 nm波长样本集建立的模型。 通过RC算法选取出羊肉硬度在400~950和900~1 650 nm波长范围的特征波长分别为14个(410, 438, 450, 464, 539, 558, 612, 684, 701, 734, 778, 866, 884和935 nm)和10个(915, 949, 1 085, 1 156, 1 206, 1 262, 1 318, 1 384, 1 542和1 580 nm)。 其中, 基于900~1 650 nm波长建立的PLSR模型的RP2为0.985 0, RMSEP为24.397 0 g, 为羊肉硬度预测的最佳模型。 结果表明, 所提出的融合二次迭代MC算法的PLSR模型可以有效预测羊肉冷藏过程中硬度特性变化趋势, 为羊肉品质无损检测相关研究提供参考。
Mutton, as a kind of meat with high protein content and low fat and cholesterol content, is becoming more and more popular with consumers. The demand for mutton is on the rise. According to the National Bureau of Statistics, China's mutton production rose from 6.27% to 9.02% from 2012 to 2019. This study proposed a quantitative detection PLSR model of mutton hardness based on the twice iterative Monte Carlo (MC) method. In this study, the Image-λ-V10E-H camera of the GaiaSorter hyperspectral sorter was used to collect the hyperspectral data of mutton samples at 400~950 nm, and the Image-λ-N17E camera was used to collect the hyperspectral data of mutton samples at 900~1 650 nm. Firstly, the study compared and analyzed four spectral pretreatment methods (S-G smoothing, 2 derivations, MSC and SNV) in eliminating interference factors, such as noise and baseline drift. Then, in the first MC sampling, the samples were divided into normal samples, suspicious samples and abnormal samples according to the 2.5 and 3 times the means of the prediction error means and standard deviations of each sample. The second MC sampling was performed based on rejecting abnormal samples, retaining and labeling suspicious samples. The new abnormal samples were eliminated by 3 times of the means of the prediction error means and standard deviations of each sample. Finally, the PLSR model based on the full wavelengths and the characteristic wavelengths extracted by the regression coefficient method (RC) were established and analyzed. The experiment results show that the twice iterative Monte Carlo method proposed in the study could abnormal samples, optimize the sample set, and provide a good foundation for modeling. With MSC as the spectral preprocessing algorithm, the PLSR model based on 400~950 and 900~1 650 nm hyperspectral data was superior to the other three spectral preprocessing algorithms RP2=0.947 2 and 0.978 3, RMSEP=47.789 9 g and 30.590 1 g. And, the accuracy and stability of the PLSR model based on 900~1 650 nm were significantly better than that based on 400~950 nm. 14 characteristic wavelengths (410, 438, 450, 464, 539, 558, 612, 684, 701, 734, 778, 866, 884, 935 nm) and 10 characteristic wavelengths (915, 949, 1 085, 1 156, 1 206, 1 262, 1 318, 1 384, 1 542 and 1 580 nm) of mutton hardness were selected by RC algorithm from 900~1 650 and 400~950 nm. The PLSR model based on 900~1 650 nm was the optimal model for predicting the hardness of mutton with RP2=0.985 0 and RMSEP=24.397 0 g. In conclusion, the PLSR model based on the twice iterative MC algorithm can effectively predict the changing trend of mutton hardness during cold storage and provide a reference for related research on non-destructive detection of mutton quality.
随着居民生活水平的提高和对健康饮食结构的重视, 羊肉作为一种高蛋白且低脂肪和胆固醇的畜肉, 需求量逐年上涨。 根据国家统计局对2012年至2019年我国肉类参量的统计, 八年间猪肉在畜肉产量中的占比从84.22%下降到78.65%, 而羊肉从6.27%上升到9.02%。 2019年我国羊肉产量达到488万吨。 羊肉的感官特性主要包括颜色、 硬度等, 能够很大程度上反映冷鲜肉的外观属性和质地特征, 直接影响消费者的购买决策[1]。 其中, 羊肉的硬度信息与肌肉纤维密度、 脂肪含量与分布均匀性等密切相关, 可以反映出活羊养殖年龄、 屠宰与贮藏时间等羊肉品质状况[2]。 羊肉的硬度可通过质地剖面分析(TPA)准确测量, 但该方法对样本的破坏性较强, 不适于羊肉生产在线检测的需求[3]。 目前, 在羊肉产业中常由人工采用“ 指压法” 测定, 存在较大的主观性[4]。
高光谱成像技术可以获得羊肉样本图像上每个像素的光谱信息, 通过计算感兴趣区域(ROI)的平均光谱可以具有代表性地表述羊肉样本的硬度属性, 为羊肉硬度的无损快速检测提供了技术支持。 Cheng等[5, 6]选出了450, 530, 550, 616, 720, 955和980 nm的7个最佳波长, 建立了鱼片硬度的LS-SVM预测模型。 Tao等[7]基于400~1 100 nm波段的猪肉高光谱散射图像, 采用Lorentzian分布函数拟合散射剖面, 建立了猪肉硬度和弹性的无损预测模型。 Kamruzzaman等[8]基于900~1 700 nm高光谱成像系统建立了羊肉硬度和剪切力的预测模型, 研究表明, 900~1 700 nm光谱数据在羊肉硬度的无损检测研究中更具潜力。 刘贵珊[9]等采集了羊肉400~950和900~1 650 nm两段高光谱数据, 区间变量迭代空间收缩法提取了123个特征波长, 提高了羊肉嫩度预测模型的准确性和稳定性。 现有相关研究一般建立在相对理想的实验数据基础上, 对于由仪器误差、 操作误差等客观因素造成的异常样本的检测方法研究相对较少。
本工作旨在研究400~950和900~1 650 nm两段高光谱数据对羊肉贮藏过程中硬度变化趋势预测的可行性。 首先, 提出了二次迭代Monte Carlo(MC)法对异常样本进行有效的检测、 验证和剔除; 然后, 基于PLSR算法建立了羊肉硬度定量预测模型; 最后, 采用回归系数法(RC)从400~950和900~1 650 nm两段高光谱数据中提取了羊肉硬度特征波长, 并建立基于特征波长的PLSR模型。
实验样品选定为绵羊的背部最长肌, 是一次性从北京某超市购置并通过恒温箱送至实验室。 将羊肉里脊横切处理为厚度约1cm的肉片, 分装入无菌袋中作为待测样本。 共制备140个样本, 对其中5个样本立即进行高光谱数据采集和硬度测定, 作为样品的初始标定值。 其余135个样本分为三组贮藏于0, 4和8 ℃的恒温箱中。 每隔24h从各温度组随机选取5个样本, 进行高光谱数据采集和硬度测定。 实验中选用GaiaSorter高光谱分选仪的Image-λ-V10E-H相机采集样品400~1 000 nm的高光谱数据, 设置电控平台移动速度为0.35 cm·s-1, 曝光时间调整为19.3 ms; Image-λ-N17E相机采集样品900~1 700 nm的高光谱数据, 设置电控平台移动速度为0.65 cm·s-1, 曝光时间调整为25 ms; TA.XTPlus物性测试仪测量样本的硬度值, 设置探头测试速度为1 mm·s-1, 样品压缩程度为50%。
MC抽样法对异常样本判定阈值具有较强的敏感性, 阈值设定过大将无法检测出部分异常样本, 而阈值设定过小则导致部分正常样本被误判[10]。 此外, 当异常样本被随机分配到校正集次数过多时, 会使正常样本的预测均值产生偏差, 从而无法对该样本做出异常认定。 本研究提出一种基于二次迭代理论的MC抽样改进方法, 以提高异常样本检测的准确能力。 具体步骤如下:
Step 1: 以所有样本为校正集建立主成分预测模型, 得到最优主成分数;
Step 2: 采用MC方法从样本集中抽取3/4或以上的样本作为校正集, 根据Step 1中所得的最优主成分数建立预测模型, 并对剩余样本进行预测, 获得剩余样本的预测误差;
Step 3: 将Step 2重复执行N次(N需足够大), 确定每个样本都作为剩余样本得到相应的预测误差;
Step 4: 基于上一步每个样本所得的多个预测误差, 计算各样本的均值μ和标准差σ , 计算方法见式(1)和式(2)
式(1)和式(2)中, k为第j个样本作为剩余样本得到预测误差的次数, ε i为该样本在第i次作为剩余样本所得的预测误差。
Step 5: 分别计算各样本作为剩余样本所得的预测误差均值和标准差; 再求的所有样本预测误差均值和标准差的平均值, 以该平均值的2.5~3倍作为可疑样本阈值, 3倍作为异常样本阈值。 剔除异常样本, 保留并标记可疑样本, 对剩余样本进行第二次MC抽样法。
Step 6: 对剔除异常样本后的样本集进行第二次MC抽样, 再次计算所有样本预测误差均值和标准差的平均值, 以该平均值3倍作为异常样本阈值。
Step 7: 对Step 5标记的可疑样本进行逐一剔除判定, 确定异常样本集。
光谱预处理可以有效消除原始光谱中噪声、 基线漂移等干扰, 是基于光谱数据建立预测模型中不可或缺的步骤。 光谱预处理方法可分为两类: 基线校正[11]和散射校正[12]。 基线校正主要以S-G多项式的一阶求导和二阶求导为代表, 散射校正主要以多元散射校正(MSC)、 标准正态变换(SNV)等算法为代表。 本研究选取S-G平滑、 二阶求导、 MSC、 SNV等四种光谱预处理方法。
PLSR是一种有效克服变量间存在多重线性关系的线性回归分析方法。 PLSR的主要原理是采用主成分分析样本的光谱数据矩阵和测量值矩阵, 探索测量值与光谱信息之间的关联性, 建立基于光谱信息预测测量值的最佳关联函数[13]。 本研究采用PLSR算法建立羊肉硬度的定量预测模型。
通过对比分析模型的决定系数(R2)、 均方根误差(RMSE)和剩余预测偏差(RPD)评估模型的性能。 R2反映了样本测量值与预测值的相关性, 取值范围为[0, 1], 计算公式见式(3)
式(3)中, yi为第i个样本的测量值,
RMSE可以用于评估样本测量值和预测值之间的偏差, 计算方法如式(4)
RPD可以通过衡量预测集的验证结果, 对模型性能进行评价, 计算方法如式(5)
式(5)中, SD为预测集样本测量值的标准偏差。 当RPD小于1.5, 说明模型的预测的性能较差; 当RPD在1.5~2之间, 说明模型可判待测量值的高低; 当RPD在2~2.5之间, 说明模型可作近似定量计算; 当模型的PRD大于2.5, 说明模型预测能力较高。
羊肉硬度随温度和时间变化的平均值与标准偏差如图1所示。 在0, 4和8 ℃环境下贮藏期间, 羊肉的硬度呈现明显的下降趋势。 羊肉样本硬度初始值为: 硬度1 291.128 g, 随着自溶现象的加深, 不同贮藏温度的样本硬度持续下降至初始值的65.09%, 47.90%和41.43%。 冷藏过程中羊肉硬度下降的原因是微生物和酶的作用下肌细胞中的蛋白纤维网架体系被破坏, 导致细胞承受外力、 保持内部结构的能力下降[14]。
 | 图1 不同冷藏温度下羊肉硬度的变化情况Fig.1 The development trends of muttons' hardness under different storage temperatures |
图2(a, b)分别为羊肉样本在380~1 020和860~1 700 nm范围的反射光谱。 从图中可知, 所有羊肉样本的反射光谱变化趋势基本相同, 在每个波长处的反射强度存在一定差异; 另外, 靠近两端的光谱信号由于干扰噪声的影响产生了明显的基线漂移。 为避免光谱曲线抖动、 基线漂移等干扰噪声对数据分析和预测模型的影响, 选取400~950和900~1 650 nm范围的反射光谱信息作为后续研究的基础数据。
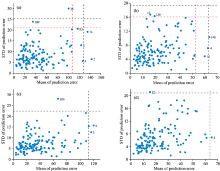
采用二次迭代MC抽样法对羊肉硬度的异常样本检测结果如图3所示。 抽样次数为5 000次, 校正集和预测集的划分比例为3:1, 红色虚线和黑色虚线分别表示可疑样本检测阈值和异常样本检测阈值。
图3(a)和(c)分别为基于400~950 nm光谱数据的羊肉硬度异常样本检测中第一次MC抽样和第二次MC抽样的结果。 由图3(a)可知, 在第一次MC抽样中, 样本3, 4, 83和109分布于2.5~3倍阈值线之间, 为可疑样本, 样本2, 35和74分布于与3倍阈值线之外, 为异常样本。 由表1可知, 在剔除3个异常样本, 并保留样本3, 4, 83和109的情况下, 剩余样本集PLSR的交叉验证参数为
| 表1 基于二次迭代MC抽样的羊肉硬度异常样本检测结果 Table 1 Detection results of mutton hardness outliers based on 2 iteration MC sampling |
图3(b)和(d)分别为基于900~1 650 nm光谱数据的羊肉硬度异常样本检测中第一次MC抽样和第二次MC抽样的结果。 同理分析, 采用二次迭代MC抽样法检测出样本4, 5, 12和140等4个异常样本。
最佳预处理方法通过对比分析上述四种方法对PLSR模型的影响进而选出, 如表2所示。 相比基于原始光谱建立的模型, 采用二阶求导、 MSC和SNV预处理后的模型性能都有所提升, 其中经过MSN预处理的预测模型
| 表2 光谱预处理结果 Table 2 Results of spectral data preprocessing |
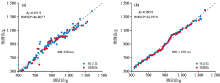
基于400~950和900~1 650 nm两组羊肉高光谱数据, 采用留一交叉验证法建立PLSR模型, 结果如表3所示。 基于400~950 nm波长建立的羊肉硬度PLSR预测模型的性能较好(RPD> 2.5), 模型的
| 表3 羊肉硬度PLSR模型的预测结果 Table 3 Mutton hardness prediction results of PLSR models |
高光谱数据的高维度导致预测模型的复杂度较高, 限制了其在实际应用中的时效性[15]。 基于最佳特征波长建立的简化预测模型, 可以在保证模型的预测精度不受较大影响的同时最大限度的降低高光谱数据的复杂度。 本研究采用RC方法提取400~950和900~1 650 nm波长范围内羊肉硬度的特征波长, 提取结果分别如图4(a, b)所示。 通过RC算法选取出羊肉硬度在400~950和900~1 650 nm波长范围的特征波长分别为14个和10个。 基于两组特征波长分别建立羊肉硬度的PLSR模型, 结果如表3所示。 与基于全波长建立的羊肉硬度模型相比, 基于特征波长建立的PLSR模型
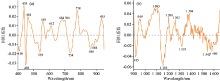 | 图4 采用RC法提取羊肉硬度的特征波长 (a): 400~900 nm; (b): 900~1 650 nmFig.4 Optimal wavelengths of mutton's hardness by RC (a): 400~900 nm; (b): 900~1 650 nm |
探讨了采用400~950和900~1 650 nm两段高光谱数据预测羊肉冷藏期间硬度变化的可行性, 具体结论如下:
(1)本研究提出的二次迭代MC方法, 将羊肉样本划分为正常样本、 可疑样本和异常样本。 通过第一次MC抽样剔除异常样本, 然后通过调整异常样本阈值进行第二次MC抽样, 进一步剔除异常样本, 最后对可疑样本进行二次判定。 实验结果证明该方法能够准确的辨别异常样本, 有助于提高建模的准确性和稳定性。
(2)研究发现, MSC预处理方法能有效消除噪声的影响, 提高模型的性能。 基于400~950和900~1 650 nm两段高光谱数据, 采用留一交叉验证法分别建立了羊肉硬度PLSR模型, 模型的
(3)采用RC方法分别从400~950和900~1 650 nm两段高光谱数据内提取出羊肉硬度特征波长为14个和10个。 根据对比分析, 基于特征波长建立的PLSR模型的预测能力更高。 其中, 基于900~1 650 nm波长建立的PLSR模型的
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|