作者简介: 郑开逸, 1983年生, 江苏大学食品与生物工程学院副研究员 e-mail: kaiyizhengjsu@126.com
近红外光谱(NIR)具有快速、 无损、 操作方便的特点, 故广泛用于食品分析。 作为一种间接的分析技术, NIR需要建立光谱与待测浓度之间的统计模型来实现检测。 故模型的维护有助于保证NIR的预测准确性。 在外界条件发生变化的情况下, 诸如样品性状的改变、 仪器对理化指标函数关系的变化、 湿度和温度等环境因素的改变, 会导致相同样品的光谱信号发生偏移, 进而使得原有模型的预测精度下降。 此时, 如果重新建模, 虽然可以解决光谱偏移对建模的影响, 但是重新建模将耗费大量的人力物力。 对此, 模型转移可以在避免重新建模的情况下, 校正光谱的偏移, 进而提高模型预测精度。 通常模型转移算法多用全光谱进行模型转移, 这种方法计算量较大, 且不能找到合适的有化学意义的波段。 故提出一种基于模型转移中的变量选择方法: 向后迭代区间选择法(IIBS), 通过计算主光谱(用于建模的那组光谱)和从光谱(发生偏移, 需要通过模型转移算法将其校正的光谱)中, 变量区间的重要性信息(回归系数( β)、 残差向量(Res)以及变量重要性投影(VIP))。 进而通过计算该区间变量重要性信息的几何平均数, 并以此作为该区间的区间重要性指标。 接着根据区间的重要性, 删除重要性信息较小的变量区间。 然后对主光谱和从光谱重复迭代上述过程: 计算变量的重要性信息, 计算区间的重要性信息, 删除重要性信息较小的区间。 最后, 比较不同的主光谱和从光谱区间组合的验证均方根误差(RMSEV), 选择RMSEV最小的主光谱和从光谱区间作为最优区间。 玉米、 小麦两套NIR数据测试了该算法。 结果显示, 与全波段相比, β, Res以及VIP均可以从主光谱和从光谱中选择较少的, 有化学意义的区间, 提高模型转移的精度。 在比较不同变量重要性向量方面, 基于 β的变量选择算法, 模型转移的计算误差较小。
The near-infrared spectra (NIR) with advantages of fastness, non-destructiveness and easy operation have been widely used in food analysis. As an indirect analysis method, NIR should calibrate the model between spectra and concentrations for analysis. Thus, the maintenance of the model can ensure high accuracy. The changes of external conditions, including the changes of samples characters, the variations of functions between physical and chemical indicators and the changes of the environment such as humidity and temperature, can diverge the spectra of the same samples and then decrease the prediction accuracy of the original model. To solve this problem, recalibration can eliminate the chances of spectra butcost huge laborious and economic expense. Thus, calibration transfer can correct the spectral divergence and improve model prediction accuracy without the expense of recalibration. In previous work, the calibration transfer algorithms usually use full spectra variables to transfer, which increase computation burden and not find spectra intervals with chemical information. Thus, this paper proposed a variable selection method called iterative interval backward selection (IIBS) for calibration transfer. IIBS firstly calculates the importance vectors of variable intervals in spectra, including regression coefficients ( β), residual errors (Res) and VIP (VIP) vectors. Then set the geometric mean of the important values of variables in each interval as the corresponding interval’s importance. Moreover, based on the importance values of intervals, remove the smallest one. After that, repeat the above procedure iteratively for both primary and secondary spectra, including computing the importance and values of variables and intervals and remove the intervals with minimal importance value. Finally, compute the root mean squared error of validation (RMSEV) for each interval subsets combination of both primary and secondary spectra and choose the intervals combination with minimal RMSEV as the best one. Two datasets, including corn and wheat datasets, were executed to test this algorithm. The results show that compared with the spectra with full intervals, the β, Res and VIP can select fewer but more important variable intervals from whole spectra to improve the calibration transfer accuracy. In contrast with different variable importance vectors, the β can select variables intervals with low prediction errors.
近红外光谱(near infrared spectra, NIR)是一种位于760~2 500 nm的电磁波。 NIR分析方法具有检测快速、 不破坏样品的特点, 故广泛用于食品[1, 2]、 石油[3]、 制药[4]等领域。 但是, NIR光谱产生机理复杂, 干扰较多, 因此它不可能像紫外-可见光谱(ultraviolet-visible spectra, UV-Vis)那样遵循严格的Lambert-Beer定律, 也就难以建立理论模型来描述光谱和浓度的关系。 对此, 可以用统计模型建立NIR光谱(X)和理化指标(y)之间的关系。 故在NIR分析中, 模型的建立和维护对分析结果的正确性至关重要[5, 6]。
然而, 当某一测量条件发生了变化, 就算是测量具有相同理化指标的样品, 得到的光谱也是有很大区别的, 这就导致建立好的模型预测不了发生变化的测量条件下的样品。 常见导致相同理化指标的光谱发生变化的原因有如下几点: (1)样品性状的改变, 即样品中与理化指标无关的成分发生变化。 (2)仪器对理化指标函数关系的变化。 (3)诸如湿度和温度等环境因素的改变。 为了解决这一矛盾, 人们提出了模型转移。 模型转移, 是指在不重新建模的情况下, 通过一定算法校正新光谱的偏移, 进而使得校正后的光谱能被原有的模型准确预测。
在模型转移中, 主光谱(A)为用于建模的那组光谱, 其在模型转移中起主导作用。 而发生偏移, 需要通过模型转移算法将其校正成类似于主光谱的光谱被称为从光谱(B)[7, 8, 9]。 在模型转移过程中, 光谱的变量数往往远大于样本数。 这些过多的变量会增加计算的负担, 降低预测精度。 故必须要对模型转移中的光谱做变量选择。 以往模型转移使用的变量选择算法, 大多是对主光谱进行变量选择, 然后从主光谱和从光谱中选择相同的波段实现模型转移。 这种方法只考虑了主光谱的有信息区段而未考虑从光谱的区段。 在实际应用中, 由于主光谱和从光谱的差异性, 主光谱的有信息区段并非从光谱的有信息区段。 此外, 有时候主光谱和从光谱并非具有相同的波段(例如主光谱为1 100~2 500 nm, 从光谱为800~1 100 nm), 甚至主从光谱并非同一种类型的的光谱(例如主光谱为NIR区段, 从光谱为可见光谱区段)。 此时, 我们无法从主光谱和从光谱中选择相同的波段。 为此提出采用向后迭代区间选择法(iterative interval backward selection, IIBS), 基于主光谱和从光谱的重要性信息, 对主光谱和从主光谱同时进行变量选择, 进而获得建模能力较强的波段。
基于光谱校正的模型转移主要是通过建立主光谱与从光谱之间的一个转移矩阵T来实现模型转移。 主要的操作步骤是: 在主光谱和从光谱中分别找到一组浓度相同的样本(转移集), 设为At(m× n1)和Bt(m× n2), 然后通过矩阵运算, 获得T。 获得T之后, 将要预测的从光谱数据乘以T, 这样就可以得到一个类似主光谱的光谱。 这样, 校正后的从光谱就可以通过由主光谱建立的模型来预测。
通常, 矩阵T通过直接校正法(direct satandardization, DS)[10, 11, 12, 13]实现, 故本工作就用DS算法实现模型转移, 具体的算法是:
①直接用At对Bt进行多元线性回归, 进而获得转移矩阵T
有时, 式(1)中的矩阵为
②对于Bp, 可以按照式(2)直接地校正成Bnew
此时, Bnew就可以直接地用主光谱的模型预测。 DS算法的优势是, 其用光谱矩阵的整体信息进行模型转移, 计算较为方便。 同时, 其矩阵乘法可以用于校正变量数不同的两个光谱矩阵。
IIBS算法的主要步骤如下:
①构造主光谱和从光谱的变量重要性信息向量:
主光谱和从光谱均可构造重要性信息向量, 在此用β , Res以及VIP数值分别构造有信息向量[14, 15]。 从光谱可以通过对其转移集的PLS建模获得相应的变量重要性指标, 诸如β , Res, VIP数值等。 其有信息向量简介如下:
β 为回归系数向量, 主光谱和从光谱的回归系数可以通过PLS拟合获得
如式(3)和式(4)所示。 在PLS模型中, 其β 的绝对值大小可以作为变量选择的指标。 如果β 的绝对值较大, 其建模能力较强, 故这些变量需要被选取。 因此, 通过比较主光谱和从光谱β 绝对值的大小, 选择绝对值较大的变量, 建立模型, 即可实现变量选择, 降低预测误差。
Res为残差, 在PLS建模时, 可以用PLS的主成分重构出主光谱和从光谱的近似矩阵
在式(5)和式(6)中, 由于矩阵
其中ej表示E矩阵中第j个列向量。 在式(7)和式(8)中可以看出, 如果第j列的残差平方和越小, qj值就越大, 该变量有信息成分占的比例就越大, 因此该变量也就越重要。 可以选择一些qj值较大的变量, 然后将这些变量组成一个集合, 这样就可以提高模型的准确度。 这样, 各个变量的的q值便构成了一条残差向量(Res)。
VIP为变量重要性投影, 它也是通过PLS成分计算得到的向量, 其长度表示的就是变量数。 一般通过设定一个阈值, 然后VIP大于这个阈值的变量就可以视为重要的变量, 进而被选择并建立模型。 也可以将变量按照VIP值大小排序, 选择具有较大的VIP值的变量并组成集合, 进而建模, 以便提高模型的精度。
②构造光谱区间的重要性向量:
考虑到主光谱和从光谱变量数均较多, 如果直接模仿主光谱校正集的基于单个变量的变量选择算法, 其主光谱和从光谱的变量子集合的组合将会非常多。 所以采用变量区间代替单个变量选择的方法来提高运算速度。 此外, 相对于离散的光谱数据点, 光谱波段更能反映光谱的化学信息[16]。 因此, 我们将整个光谱集就分成多个区间, 每个区间的重要性以该区间每个变量的重要性的平均数来表征。
考虑到每个变量的重要性指标均大于零, 而且有时候区间中某个重要性较大变量可能会掩盖重要性较小的变量。 故我们选择几何平均数而非算术平均数, 因为几何平均数既可以总体反映变量区间中各个变量的重要性信息, 也可以保证变量重要性信息受到异常的大值的影响较小。
(3)选择重要性较大的光谱区间:
按照重要性顺序排列, 将主光谱和从光谱的区间, 按照其区间的重要性排序, 选择重要性较大的区间。 考虑到变量区间的重要性指标会随着变量区间数的缩小发生细微的变化, 故我们计划用逐步删除的办法, 每一次迭代, 删除一个重要性最差的区间, 最后将重要性数值较大的区间保留下来, 同时重新计算每个区段的重要性并进行新的一轮迭代, 直到剩下最后一个区间。
考虑到光谱信息的复杂性, 有的化学信息往往在多个波段中均有体现。 故在区间优化的过程中, 如果单纯优选出变量重要性最大的区间, 其建模能力也可能不是最优, 因为有时若干个建模能力较弱的区间, 其信息具有互补性, 其组合建模的预测效果好。 故为了提高区间选择的效果, 用验证均方根误差(root mean squared error of validation, RMSEV)来评价区间组合的建模能力。
IIBS算法的详细流程如图1所示。
在图1中, IIBS先以区间重要性指标, 通过向后筛选法构造主光谱的一系列区间子集合。 然后对主光谱的每个集合, 计算从光谱的区间重要性指标, 以向后筛选的方法建立从光谱的一系列区间子集合。 最后比较这些主从光谱子集合组合后模型转移的RMSEV值, 选择RMSEV最小的主光谱和从光谱的子集合组合。
数据集被分为四部分, 转移集, 校正集, 验证集, 独立测试集。 转移集用于模型转移; 校正集用于建立模型; 验证集用于计算验证误差, 优化参数, 进而获得最佳的变量集; 独立测试集不参与模型优化, 只用于检验变量优选后模型的预测最终结果。 主光谱的转移集用Kennard-Stone方法从主光谱的校正集中选出, 然后从光谱中和主光谱相同浓度的样品作为转移集的从光谱。
1.2.1 玉米数据集
玉米数据下载于: http://www.eigenvector.com/data/Corn/index.html。 这套光谱里有三组数据集m5, mp5, mp6; 波长范围均是1 100~2 498 nm (700个波长点)。 选择mp6作为主光谱, m5作为从光谱, 取水分数据作为y值。 将y浓度从小到大排序, 每4个连续的样本中取出第一个样本, 这样20个样本就被取出, 剩下60个样本为校正集。 取出的20个样本中, 按照浓度排序, 每两个样本中第一个为验证集, 第二个为独立测试集。 因此验证集与独立测试集的样本数均为10。
1.2.2 小麦数据集
小麦的数据取自: http://www.wiley.com/legacy/wileychi/chemometrics/datasets.html, 这套数据有775个样本, 1 050个波数点, 波长范围是400~2 498 nm。 其中蛋白质含量作为待测指标。 为了研究IIBS算法处理不同波数点数据集的能力, 我们将该数据集分为两个部分: 可见-短波NIR和长波NIR。 其中可见-短波NIR的数据点包括350个波长点(400~1 098 nm), 长波NIR包括700个波长点(1 100~2 498 nm)。 主光谱选择长波NIR, 从光谱选择可见-短波NIR。 其中400条光谱作为校正集, 50条光谱作为验证集, 325条光谱作为独立测试集合。
IIBS算法中, 两个参数对建模非常重要: 转移集的样本数(m)以及区间的长度(n)。 选择不同的m和n组合, 可以获得不同组合下RMSEV的值, 同时计算不同m值下, 全光谱的RMSEV值。 以β 做为变量重要性指标, 经过搜索, 发现在m=30, n=14时, 所选择的变量可以取得较小的RMSEV值。 故选择m=30, n=14。
在通过验证集确定参数后, 需要用独立测试集检测相应参数下, 模型的计算结果, 结果如表1所示。
| 表1 玉米数据不同重要性指标变量选择结果 Table 1 The results of different variable selection methods for corn dataset |
在表1中, 与全光谱变量选择的结果相比, 基于β 的IIBS算法不仅可以使得验证集获得较小的RMSEV值, 而且可以使独立测试集获得较小的RMSEP值。 和β 一样, VIP值也可以选择合适的变量获得较低的RMSEV和RMSEP值。 Res虽然也可以降低RMSEV数值, 但是Res的RMSEP数值反而大于全光谱的RMSEP值。 其原因可能是Res虽然选择了较少的变量, 而这些变量只利于校正集, 导致了过拟合, 反而增大了独立测试集的RMSEP值。 故β , Res以及VIP可以被看作模型转移中的变量重要性向量, 用于变量选择。
为了研究变量选择结果的化学意义, 基于β , Res, VIP的IIBS算法选择的变量如图2所示。
从图2中可以看出, 虽然β , Res, VIP三种指标选择的变量互不相同, 但是β 和VIP选择的变量位置相似性较高, 诸如二者的主光谱均选择了1 450和2 300 nm附近的吸收峰, 且二者的从光谱也选择了1 450, 1 950以及2 300 nm附近的吸收峰。 这些吸收峰都与水的吸收密切相关。 1 950 nm附近的吸收可以称为水的吸收Ⅰ 区1 450 nm附近的吸收可以称为水的吸收Ⅱ 区, 均与O— H的伸缩振动有关[17, 18]。 此外, 2 300 nm附近的吸收也与水的吸收有关[18, 19]。 而基于Res的变量选择算法则与β 以及VIP有较大的区别, 首先, Res从主光谱中选择了280个变量, 从从光谱选择了462个变量。 其次, 它选择了一些与水相关性较小的区段, 诸如1 150~1 350 nm。 此外, 它没有选中一些与水相关性较强的区段, 例如: 主光谱没有选择2 300 nm附近的吸收峰, 从光谱没有选择1 950 nm附近的吸收峰。 这可能是导致Res选择的变量具有较高的误差的原因。
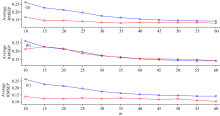
为了更加深入研究变量选择的结果, 我们将数据进行随机分类, 随机生成校正集(60个样本), 验证集(20个样本), 独立测试集(20个样本)。 然后用β (n=14), Res(n=14)以及VIP(n=20)进行变量选择, 利用验证集筛选出好的变量, 然后将其代入独立测试集中或的预测误差。 重复上述步骤100次, 获得的误差均值如图3所示。
从图3中可以看出, 基于β 以及VIP的IIBS选择的变量, 其RMSEP均值明显地小于全波段的RMSEP均值, 这证明了上述算法的有效性。 选择Res的IIBS, 其计算结果的RMSEP均值和全光谱的均值相近, 甚至在一些m值下, 其误差反而大于全波段的RMSEP均值。 故对于玉米数据, IIBS结合β 以及VIP可以选择出较好的变量, 并获得较低的RMSEP值。
与前者类似, 小麦数据也被随机分成三部分: 校正集400条光谱, 验证集50条光谱, 独立测试集325条光谱。 在IIBS算法中, β , Res以及VIP算法的n值均为20。 将上述方法重复运行100次, 其计算结果如图4所示。
从图4中可以看出, 与全波段建模比较, 基于β , Res, VIP的IIBS算法均可以降低独立测试集的RMSEP值。 这证明了变量选择的有效性。 同时, 在图4中可以得出基于β 以及Res的计算误差要显著小于基于VIP的计算误差。 故对于小麦数据, β , Res可以获得较好的变量集合。
向后迭代区间选择法(iterative interval backward selection, IIBS)通过多次迭代, 每次迭代删去重要性最小的区间, 最终获得主光谱和从光谱模型转移误差最小的区间。 玉米、 小麦NIR数据测试了IIBS算法。 结果显示, 相对于全波段, IIBS算法可以有效地从主光谱以及从光谱中同时筛选出各自有意义的波段, 实现降低误差, 提高预测精度。 同时, 在选择不同的变量重要性向量方面, 基于回归系数的IIBS算法可以获得较小的预测误差。 因此, IIBS可以用于模型转移中的变量选择, 进而获得较小的误差。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|