作者简介: 路皓翔, 1991年生, 桂林电子科技大学计算机与信息安全学院博士研究生 e-mail: 646510477@qq.com
近红外光谱分析技术对检测样品无损伤且检测速度快、 精度高, 因此被广泛应用在了药品检测、 石油化工等领域, 尤其近年来机器学习和深度学习建模方法的深入应用使其具备了更准确的检测性能。 然而, 样品的近红外光谱数据具有比较高的维度且存在谱间重合、 共线性和噪声等问题, 对近红外光谱模型的性能产生消极影响, 此时样品有效特征波长的筛选极为重要。 为了提高近红外光谱定量和定性分析模型的准确性和可靠性, 提出了一种近红外光谱变量选择方法, 其结合了最小角回归(LAR)和竞争性自适应重加权采样(CARS)的优点, 具有更优的性能。 该方法利用LAR初步筛选样品全谱区的特征波长, 接着利用CARS对筛选出来的特征波长进一步选择, 从而有效去除无关特征波长。 为验证该方法的有效性, 从定量和定性分析两个方面评价该方法。 在定量分析实验中, 以FULL, LAR, CARS, SPA和UVE作为对比方法, 以药品样品数据集为实例建立PLS回归分析模型, 经LAR-CARS筛选出的变量建立的PLS模型在药品数据集表现出较高的预测决定系数和较低的预测标准偏差。 在定性分析实验中, 以SVM, ELM, SWELM和BP作为对比方法、 不同比例训练集的药品数据集为实例建立分类模型, 经LAR-CARS筛选出的变量建立的SVM分类模型精度最高达100%。 从实验结果可见, LAR-CARS可有效的筛选出表征样品特征的波长, 利用其筛选出的波长建立的定量、 定性分析模型具有更好的鲁棒性, 可用于样品光谱的特征波长筛选。
Near-infrared spectroscopy is widely used in drug detection, petrochemical industry, etc., because it has no damage to the samples, and the detection speed is fast, and the accuracy is high. In particular, it has more accurate detection performance with the in-depth application of machine learning and deep learning modeling methods in recent years makes. However, the NIR spectral data of the sample has relatively high dimensions and has problems such as spectral overlap, collinearity and noise, which will negatively impact the performance of the NIR spectral model. In this case, the selection of effective characteristic wavelength points of the sample is extremely important. In order to improve the accuracy and reliability of the quantitative and qualitative analysis models of NIR spectra, a variable selection method for NIR spectra is proposed, which combines the advantages of Least Angle Regression and Competitive Adaptive Re-weighted Sampling, and has better performance. In this method, LAR was used to preliminarily screen the characteristic wavelengths in the whole spectrum of the sample, and then CARS was used to further select the selected characteristic wavelengths to effectively remove the irrelevant characteristic wavelengths. In order to verify the effectiveness of the method, the method was evaluated from two aspects of quantitative and qualitative analysis. In the quantitative analysis experiment, PLS regression analysis model was established using FULL, LAR, CARS, SPA and UVE as comparison methods and drug sample data set as example. PLS model established by variables screened by LAR-CARS showed higher predictive determination coefficient and lower predictive standard deviation in drug data set. In the qualitative analysis experiment, the classification model was established with SVM, ELM, SWELM and BP as comparison methods and drug data sets with different proportions of training sets. The accuracy of the SVM classification model established by the variables screened by LAR-CARS reached the highest 100%. From the experimental results, it can be seen that LAR-CARS can effectively select the wavelength points that the characteristics of the sample, and the quantitative and qualitative analysis model established by using the selected wavelength points has better robustness and can be used for the characteristic wavelength screening of the sample spectrum.
近红外光谱分析凭借其便捷且不存在污染的特点自20世纪80年代以来, 被广泛应用在各个领域[1, 2]。 样品光谱数据的重叠以及特征吸收区域不明显, 导致全谱区建立的分析模型准确度和可靠性较差[3, 4, 5]。 故在利用样品光谱数据建立稳定性较强的分析模型时, 首要做的是筛选出能够表征样品特征的特征波长[6]。 在国外, Wang等[7]以样品光谱数据建立的偏最小二乘回归模型回归系数为依据的无信息变量消除法实现了样品光谱数据中特征波长的筛选。 Tsakiridis等[8]采用堆叠的遗传算法选择波长解决光谱数据共线性问题。 在国内, 王坤等[9]采用蒙特卡罗-无信息变量消除-连续投影算法对样品光谱数据进行特征波长选择。 李鑫星等[10]采用主成分分析法结合连续投影算法对样品的特征光谱进行筛选。 赵环[11]等对样品光谱数据二进制矩阵采样法进行采样, 然后根据变量的频率及偏最小二乘法(partial least squares, PLS)的回归系数求解出每个光谱波长的贡献率, 进而对样品光谱数据的全波长进行筛选。 传统的变量筛选方法一般采用回归系数作为变量筛选的依据, 会导致筛选出来的变量不能完全表征样本特征, 从而影响分析模型的性能。
近些年来, 机器学习凭借其较好的分析能力在各个领域也有了极为广泛的应用[12, 13]。 国内外的一些专家学者尝试将机器学习算法用于样品光谱最佳波长的筛选从而提高近红外光谱分析模型的准确性和可靠性[14, 15, 16]。 文中结合竞争性自适应重加权采样(competitive adaptive reweighted sampling, CARS)和最小角回归(least angle regression, LAR)提出了一种自适应最小角回归波长筛选方法, 用于解决样品光谱数据谱区重叠及共线性问题。 首先采用LAR对样品光谱数据进行筛选; 然后利用CARS进一步优选样品光谱波长; 最后采用药品的近红外光谱数据进行相应的定量定性分析, 实验结果表明经过波长筛选后的药品近红外光谱分析模型预测更为准确。
LAR通过将变量中无关变量的系数设为0实现有用变量的选择[17]。 其模型为
其中, (xi1, xi2, …, xip)为样本i的波长, yi为样本的响应, β i为样本i第j个波长的系数, t≥ 0为约束值。 在t→ 0, LAR通过将对样本i影响较小的波长的系数β j置为0, 筛选出最能表征样本i属性的波长。
CARS利用自适应重采样依据求解的回归系数绝对值的大小来进行变量的选择[17]。 假设样本光谱数据为Xm× p, 其得分矩阵记为T, 则
其中W为组合系数, 样本的目标响应变量Ym× 1与样品光谱Xm× p之间满足
式(3)中, c为Y对T进行回归分析的回归系数, e为模型的预测残差。 将式(2)代入式(3)中得
式(4)中, b=Wc=[b1, b2, …, bp]表示相对于原始变量的回归系数向量。 |bi|为b中第i个波长对目标响应变量Y的贡献能力, 其值越大表示相应的波长越重要。 采用经过标准化处理的回归系数对各波长的重要性进行评价, 定义样品波长的权重为
根据ω i的值对样品变量进行筛选, 其值越大说明该波长的贡献越大。
假设样本的光谱数据为X={xi1, xi2, …, xil}, LAR-CARS筛选光谱波长的具体过程为:
Stage Ⅰ : LAR变量初筛
(1) 求解回归变量矩阵β j。 将样品的光谱数据X={xi, yi}送入LAR模型中, 遵循最小化AIC原则, 对回归系数进行求解进而构造出回归变量矩阵。 该矩阵中1表示该波长与样本i相关性较高, 0表示该波长与样本i相关性较低。
(2) 变量初筛。 LAR模型利用变量矩阵求解样品光谱的特征波长, 即,
式中,
Stage Ⅱ : CARS变量再筛
(3) 变量再筛。 利用筛选出来的变量
贡献力矩阵bi可以表示为
进而通过该矩阵求解各个波长相应的权重ω i, 式(8)代入式(4)中得,
根据ω i的值, 对LAR提取的样品光谱波长进一步优选, 最终得出对响应变量贡献最大的波长记为
为了评价LAR-CARS波长选择方法的有效性, 分别采用不同样品的光谱数据进行定量定性分析, 并采用全波长(FULL), LAR, CARS, SPA和UVE作为对比方法。
2.1.1 实验数据
用由国际漫反射会议公开的药品近红外光谱数据为例评价LAR-CARS模型。 其中, 药品样品包含655个药品的近红外光谱信息(155条作为模型校正集, 460条作为测试集, 40条作为验证集), 这些光谱由Foss NIR Systems光谱仪以2 nm为间隔在600~1 898 nm范围内测得。 具体信息如表1所示。
| 表1 实验样品详细信息 Table 1 Detailed information of experimental samples |
2.1.2 数据预处理
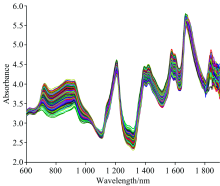
药品样品的近红外光谱信息如图1所示。 从图中可以看出受环境及仪器的影响通常采集的样品和药品光谱数据中包含电噪声、 杂散光等, 这些信息会对模型的建立产生消极的影响。 故采用S-G卷积平滑法(窗口大小17, 多项式导数为3)对样品光谱数据进行平滑化处理, 以消除噪声、 杂散光等信息。
2.1.3 变量筛选结果
LAR, CARS, UVE, SPA和LAR-CARDS在药品数据集上筛选出来的变量分布为152, 76, 198, 103和51。 图2直观的表示出了不同的变量筛选方法在药品数据集上筛选出来特征变量在全谱区的分布。
实验中不同变量选择方法对样本的波长筛选存在随机性。 以药品数据集一次实验为例, 由图2可看出LAR筛选出来的波长主要分布在1 150~1 200, 1 450~1 550和1 750~1 850 nm范围内, CARS筛选出来的波长主要分布在1 200~1 250, 1 450~1 500和1 650~1 750 nm范围内, LAR-CARS筛选出来的波长主要分布在1 150~1 250, 1 450~1 550和1 650~1 700 nm范围内, 基本与LAR和CARS筛选出来的变量重合, 但其筛选出的变量更少。
2.1.4 PLS回归结果分析
以表1中药品光谱数据为例建立药品近红外光谱定量分析模型对LAR-CARS的性能进行分析评估, 并与全波长(记为FULL), LAR, CARS, UVE和SPA进行对比。 在利用PLS建立预测模型时采用RMSEC最小来确定样品主成分数, 并从校正决定系数、 校正均方根误差、 预测决定系数和预测均方根误差四个方面评估各模型。 预测决定系数越小、 预测均方根误差越大表明近红外光谱分析模型的性能越稳定。 PLS回归模型在药品数据集上经过不同波长选择方法筛选的样品波长数、 PLS回归分析的主成分数以及训练集和测试集的决定系数和均方根误差结果如表2所示。 从表2中可看出, 采用各模型筛选的波长建立的药品近红外光谱定量分析模型较全波长建立的模型具有更高的预测精度且用于建立模型的波长数较少。
| 表2 PLS回归模型用不同波长选择方法的预测效果 Table 2 Predictive effects of PLS regression model by different wavelength selection methods |
在药品样品的分析模型中, 经过LAR-CARS筛选后建立的PLS分析模型所需波长数最少为51个, 同时PLS模型具有最高的预测标准偏差和预测校正系数分别为0.929 1和4.667 4。 LAR和UVE所需要的波长数最多分别为152和198个, 但其预测标准偏差较全波长较低较LAR-CARS较高, 预测决定系数较全波长较高但是较LAR-CARS较低, 说明该模型的性能较LAR-CARS较差。
2.2.1 实验数据
采用中国食品药品检定研究院利用Bruker Matrix光谱仪测定的不同生产厂家生产的头孢克肟片光谱数据验证LAR-CARS方法的有效性。 其中光谱仪的采样区间设置为4 000~11 995 cm-1, 采样间隔为4 cm-1, 每个头孢克肟片光谱包含2 074个特征波长。 头孢克肟片样品的详细信息如表3所示。
| 表3 头孢克肟片近红外光谱数据集数据统计 Table 3 Data statistics of cefxime tablets near infrared spectral data set |
2.2.2 数据预处理
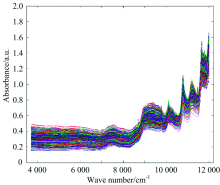
由于头孢克肟片药品的光谱数据存在偏移, 为了增大头孢克肟光谱样品数据的识别度、 减少样品谱图之间的信息重叠, 采用归一化、 平滑求导及标准正态变量多阶段数据预处理方法对其进行处理, 预处理后的光谱信息如图3所示。 可以看出经过预处理后的头孢克肟片的光谱的辨识度增加, 更有利于药品的鉴别。
2.2.3 定量结果分析
将江苏正大生产的药品光谱数据作为正类药品, 其他三个厂商生产药品的作为负类药品。 采用分类精度、 预测时间以及预测标准偏差作为各个模型性能的衡量指标。
(1) 与其他模型比较
为了进一步验证LAR-CARS在近红外光谱定性分析模型中的性能, 采用SVM, ELM, SWELM和BP作为对比方法进行鉴别实验, 将测试集按0.1~0.9的比例从药品近红外光谱数据集中随机抽取数据构建不同规模的训练集, 并对SVM, ELM, SWELM和BP各模型进行配置。 结果如表4所示。 从表中可看出无论训练集的大小如何, 经过LAR-CARS筛选波长并采用SVM对药品鉴别较全波长建立SVM模型的预测准确度有了较大的提高, 尤其当比例越大LAR-CARS-SVM的预测精度越高, 最高达100%。 之所以LAR-CARS-SVM较SVM的鉴别精度有提高, 是因为LAR-CARS方法滤除了样品光谱数据中无关的波长。 ELM和SWELM表现出的分类能力一致, 表明激活函数对模型的分类精度影响不大。 BP神经网络较ELM和SWELM具有较高的分类准确度, 这说明BP神经网络较ELM和SWELM具有较强的建模能力。
| 表4 不同比例下各模型的分类准确度 Table 4 Classification accuracies of each model under different scales |
在运行时间方面, 从表4中可看出ELM, SVM, SWELM和LAR-CARS-SVM模型较BP神经网络训练时间均更短, 这是因为BP神经网络需要通过多次迭代对网络参数进行反向微调从而求解出最优模型, 然而ELM和SWELM属于浅层神经网络无需参数的反向微调、 SVM则无需对参数的迭代更新。 此外经LAR-CARS筛选后用于建立SVM模型的时间较采用全波长建立模型的时间较短, 这主要是LAR-CARS筛选后用建立模型的波长数较少, 减少了运算量使得LAR-CARS-SVM较SVM模型的运行时间更短。
以预测标准偏差作为模型稳定性的评价指标, ELM, SVM, SWELM, LAR-CARS-SVM模型在不同规模训练集的预测标准偏差如图4所示。 从图中可看出, 经过LAR-CARS变量筛选后建立的SVM药品鉴别模型较全波长建立的SVM模型具有更强的稳定性, 说明LAR-CARS可有效删除不利于模型稳定性的样品波长。 BP神经网络的稳定性次之、 SWELM和ELM表现出最差的稳定性。
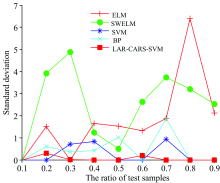 | 图4 不同比例训练集下各模型的预测标准偏差(2)变量筛选结果Fig.4 Prediction standard deviations of each model with different proportion training sets |
为了更加直观的表示出LAR, CARS, LAR-CARS, SPA和UVE变量筛选方法筛选出来的变量分布, 以比例为0.7训练集为实例进行实验。 LAR, CARS, LAR-CARS, SPA和UVE在训练集上筛选出来的变量数分别为70, 46, 40, 100和130, 其在全谱区的分布如图5所示。 由于波长在选择过程中会存在些许误差。 以头孢克肟片光谱数据的一次实验为例, 由图5可以看出LAR筛选出来的波长主要分布在8 050~9 050和10 800~11 300 nm范围内, CARS筛选出来的波长主要分布在8 050~9 060和10 500~11 200 nm范围内, LAR-CARS筛选出来的波长主要分布在8 050~9 050和11 000~11 500 nm范围内, 可看出LAR-CARS筛选出来的波长变量基本和上述LAR和CARS筛选出来的波长变量吻合。
 | 图5 各方法在头孢克肟片数据集中筛选出来的变量分布Fig.5 Distribution of variables selected by each method for cefixime tablet dataset |
(3)与已有波长选择方法比较
为了说明LAR-CARS波长筛选方法的有效性, 分别采用FULL, LAR, CARS, SPA和UVE变量选择方法作为对比方法, 然后将不同变量筛选法筛选出来的样品的特征波长点送入ELM, SWELM和SVM进行真假药品的鉴别。 在训练集占头孢克肟片光谱数据的比例为0.7时的实验结果如表5所示。
| 表5 不同变量选择方法下各模型的分类精度 Table 5 Classification accuracy of each model using different variable selection methods |
从表5中可看出采用全波长建立的ELM, SWELM和SVM模型的分类精度较采用变量筛选选择的变量建立的模型低, 这是由于样品的近红外光谱数据包含了随机噪声和对表征样品特征相关性较差的波长, 这些波长对于鉴别模型的建立起着消极的影响, 而变量筛选能够筛选出更能表征样品特征的波长并将无关的波长删除。 与SPA, UVE相比, 用本方法筛选的样品光谱数据的波长建立的ELM, SWELM和SVM模型具有更优良的性能。
近红外光谱分析应用广泛, 但样品光谱数据中通常包含较多的共线性或低信噪比波长, 严重影响了近红外光谱分析模型的准确性。 针对这一问题, 采用LAR结合CARS提出了一种新的变量选择方法, 并以药品数据集的定量和定性分析为例对该方法进行了评价。 首先, 利用PLS模型建立了药品近红外光谱定量回归分析模型, 实验结果表明经过LAR-CARS筛选出的变量建立的PLS模型具有更优的性能。 接着, 以四个不同厂家生产的头孢克肟片光谱数据为例构建了不同厂商药品近红外光谱的定性分析模型, 采用不同比例的训练验证LAR-CARS方法的有效性, 结果表明, 采用LAR-CARS筛选出来的变量建立的分类模型具有更高的分类准确度。 综上, LAR-CARS方法能够很好的筛选出样品的有效波长, 建立的近红外光谱分析模型具有更强的应用性和鲁棒性。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|