作者简介: 阮 真, 1989年生, 重庆医科大学检验医学院主管技师 e-mail: 224751928@qq.com
非结核分枝杆菌(NTM)是除结核分枝杆菌复合群(MTC)和麻风分支杆菌以外的分枝杆菌总称。 近年来NTM导致人类感染的发病率不断上升, 其感染的临床症状与MTC感染极为相似, 但两者治疗方案却存在差异, 临床亟须快速、 准确的鉴定方法用于诊断NTM感染。 单细胞拉曼光谱技术(SCRS)具有非标记、 免培养、 快速、 准确、 低成本等优势。 据此, 我们提出了一种基于显微共聚焦单细胞拉曼光谱技术鉴定NTM的方法。 通过对临床常见的六种NTM(脓肿分枝杆菌、 戈登分枝杆菌、 偶发分枝杆菌、 土分枝杆菌、 鸟分枝杆菌以及堪萨斯分枝杆菌)的拉曼光谱进行处理比较, 并结合峰位注释进行分析。 采用无监督低维可视化的 t-分布式随机邻域嵌入方法展示六种NTM的拉曼数据结构, 证明其数据在低维空间上的可分性后, 比较分类中常用的六种分类器[支持向量机分析(SVM)、 K最近邻分类算法(KNN)、 偏最小二乘判别分析(PLS-DA)、 随机森林(RF)、 线性判别分析(LDA)、 XG Boost]的效果。 SVM和LDA在NTM分类中效果最好, 分别达到了99.4%和98.8%的测试准确率; SVM仅对于堪萨斯分枝杆菌(97.96%, 48/49)的分类准确性略低, 其余均为100%; LDA对于脓肿分枝杆菌(95.65%, 22/23)和戈登分枝杆菌(96.30%, 26/27), 其余也均为100%。 因此, 单细胞拉曼检测结合SVM分类器为NTM快速准确鉴定提供了富有潜力的新工具。
Non-tuberculosis mycobacteria (NTM) are the collection of mycobacteria other than Mycobacterium tuberculosis complex (MTC) and Mycobacterium leprosy. The clinical symptoms of NTM are very similar to MTC infection, yet their treatments are different, thus rapid and accurate identification methods of NTM are urgently needed. Single-cell Raman Spectroscopy (SCRS) is label-free, and independent of cultivation, thus it is deemed a rapid and efficient technology with low cost. Here we propose an SCRS based method to identify NTM based on confocal SCRS. We selected six common NTM species in the clinic, Mycobacterium abscessus, Mycobacterium gordonae, Mycobacterium fortuitum, Mycobacterium fortuitum, Mycobacterium avium and Mycobacterium kansasii. The unsupervised low-dimensional visualization t-distribution random neighborhood embedding method for the data structures proved the separability of data in the low-dimensional space. Performance of six commonly classifiers, including Support Vector Machine (SVM), K-Nearest Neighbor method (KNN), Partial Least Square-Discriminate Analysis (PLS-DA), Random Forests (RF), Linear Discriminant Analysis (LDA) and XG Boost was compared, with SVM and LDA achieving an accuracy of 99.4% and 98.8% respectively in NTMs classification. SVM offers 100% classification accuracy for every species, except Mycobacterium kansasii which is slightly lower (97.96%, 48/49), while LDA offers 100% accuracy for each species except Mycobacterium abscessus (95.65%; 22/23) and Mycobacterium gordonae(96.30%, 26/27). Therefore, SCRS combined with SVM can accurately classify NTMs and thus provide a new tool for the rapid diagnosis of NTM.
非结核分枝杆菌(non-tuberculosis mycobacteria, NTM)作为分枝杆菌属中除结核分枝杆菌复合群(Mycobacterium tuberculosis complex, MTC)和麻风分支杆菌以外的分枝杆菌, 截至目前共发现近200种, 致病能力各不相同[1]。 分枝杆菌具有相似的生物学性状: 多生长周期长(1~8周)、 有抗酸性。 NTM导致人类感染无论是临床症状还是其培养后的菌落常与MTC难以区分, 导致其易被误认为MTC进行诊治, 但常规的MTC治疗方案对NTM可能没有效果, 还会诱导耐药菌的产生[2, 3]。 近年来NTM的分离率不断增加, 许多国家和地区在结核病的发病率不断下降的同时NTM的感染率和耐药率却在不断上升[4, 5]。 NTM感染的诊断和治疗依赖于病原学依据[6, 7]。 由于存在生物安全风险, 常规实验室多未对分枝杆菌进行鉴定。 目前, 基因测序是NTM鉴定的金标准, 但操作相对繁琐, 需在专业实验室进行检测。 商品化试剂盒用于鉴别NTM其敏感性受标本质量影响较大, 仅能用于常见NTM鉴定, 无法对新物种检测[8, 9]。 当下亟须一种能快速、 准确区分和鉴定NTM的新方法来帮助临床诊断NTM感染。
单细胞拉曼光谱技术(single-cell Raman spectroscopy, SCRS)是基于拉曼光谱分析原理实现检测物质结构的新方法, 通过收集检测物的生物学信息(包括蛋白质、 核酸、 脂质等)组成生物体特有的“ 指纹图谱” , 并结合不同的分析方法实现对检测物的快速、 准确区分[10, 11]。 由于SCRS检测样本无需特殊处理、 操作简便, 20世纪初SCRS开始应用于病原微生物研究在微生物鉴定方面展现了强大的应用前景[12, 13]。
本次实验选择了NTM菌群中六种最常导致临床感染的NTM菌株, 通过直接单个细胞拉曼检测、 原始拉曼光谱比较、 峰位注释分析、 比较常用的六种分类器效果, 来寻找最适合NTM区分鉴定的分类方法, 为临床快速、 准确鉴定NTM提供新思路。
实验涉及NTM菌株共六种(表1), 由重庆市公共卫生医疗救治中心实验室提供。 经灭活后送至北京睿博兴科生物技术有限公司(RuiBio BioTech)进行16S rRNA 测序鉴定, 确定细菌种类。
| 表1 菌株列表及获得单细胞拉曼光谱数 Table 1 List of species and the number of Raman spectra from single cells |
在Ⅱ 级生物安全柜内将NTM菌株接种在Loewenstein-Jensen(L-J)培养基上, 置于细菌培养箱中, 37 ℃培养14 d。 培养后的NTM菌落于生理盐水中制备菌悬液。 放入恒温金属浴中100 ℃ 15 min, 对NTM菌株灭活。 已有研究证实高温灭活微生物细胞对后续拉曼检测结果影响不大[14]。
检测单细胞的拉曼光谱由临床单细胞拉曼耐药性快检仪(CAST-R, 青岛星赛)收集。 具体配备含有532 nm Nd:YAG激光器(cobolt, Sweden), CCD图像传感器(Andor, UK), 100倍长焦镜头(NA=0.95, Olympus, Japan)等。
取1 mL菌液, 10 000 g离心2 min收集菌体后用超纯水洗涤3次, 重悬于1 mL超纯水中。 取重悬后的菌液1 μL于干净的氟化钙玻片上, 将样品置于拉曼光谱仪的显微镜下, 进行拉曼测量。 每个样品随机选取 100个单细胞和4个无细胞区域, 逐一收集拉曼光谱。 单细胞拉曼光谱测量参数为: 输出激光为532 nm, 输出强度约为100 mW, 50% filter下收集时间为5 s。
采用R软件对6种单细胞拉曼光谱进行减背景、 一阶导数变换和Savitzky-Golay卷积平滑处理, 将处理后的数据按照7:3的比例划分成训练集和测试集, 采用训练集数据构建各类预测模型, 应用该模型对测试集进行预测, 测试模型性能。 处理后的光谱进行t-分布式随机邻域嵌入, 测试支持向量机分析(support vector machine, SVM)、 K最近邻分类算法(K-nearest neighbor method, KNN)、 偏最小二乘判别分析(partial least square-discriminate analysis, PLS-DA)、 随机森林(random forests, RF)、 线性判别分析(linear discriminant analysis, LDA)、 XG Boost等的分类效果。
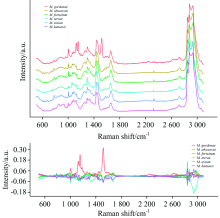
本研究共获得6种NTM菌株细胞拉曼特征光谱553 例, 表1记录了具体光谱分布。 经基线扣除及归一化处理后, 各样品的绝对峰值差异已经消除, 各样品已基本处于分析的同一水平线。 从不同类别细胞光谱的平均光谱可以看出, 不同分枝杆菌的单细胞拉曼光谱的大致形态相似, 但根据平均光谱的差异峰谱可以看出, 不同分枝杆菌的单细胞拉曼光谱在局部波数范围内存在不同程度的差异(图1)。
其中, 戈登分枝杆菌与其他分枝杆菌的差异较大, 主要表现在类胡萝卜素(1 153.82和1 518.22 cm-1)含量高于其他细胞, 代表戈登分枝杆菌单细胞相较于其他分枝杆菌而言存在细胞色素的累积(表2)。 土分枝杆菌的单细胞拉曼光谱显示其苯丙氨酸(1 004.49 cm-1等)位置上与其他分枝杆菌的光谱相比强度较低, 代表土分枝杆菌中苯丙氨酸的含量相对较少。 分枝杆菌细胞壁中复合脂质含量较高, 其主要成分为分枝菌酸(碳原子含量70~90个), 分枝菌酸被认为是分枝杆菌不同种属间特异性的化合物, 不同种的分枝杆菌菌酸含量存在差异[15]。 从NTM细胞的拉曼光谱图中也可观察到这种差异性, 在1 449.47 cm-1位置上, 六种分枝杆菌的拉曼信号强度呈现阶梯式差异, 代表六种NTM单细胞内菌酸含量呈现不同程度的差异。
| 表2 细胞拉曼光谱的峰位归属 Table 2 Molecular structure and composition of Raman peaks |
为验证光谱数据结构是否具有可分性, 在训练分类器之前, 采用低维投影可视化的方法测试数据在低维空间上是否可分。 将原始光谱进行减背景, 一阶导数变换及Savitzky-Golay卷积平滑处理(图2), 然后采用t-SNE的方法展示数据在二维空间上的区分程度。
将高维的光谱数据在二维空间中进行展示(图3)。 根据结果可以看出, 六种NTM可以较好的相互区分开, 代表虽然属于同一菌属的分枝杆菌在生物学性状上不易区分, 但它们的拉曼光谱数据结构存在很好的可分性。 其中土分枝杆菌和堪萨斯分枝杆菌相对其他种类的分枝杆菌的区分程度更显著。
Fig.3t-SNE results of six NTMs SCRS
为获得更好的分类效果, 本研究测试了六种常用的机器学习的方法构建分类器, 分别为SVM, KNN, PLS-DA, RF, LDA和XG Boost。 六种分析方法对分枝杆菌单细胞拉曼光谱进行建模分析, 采用相同的训练及测试过程, 通过比较测试集的分类准确率选择最合适的分类方法。
首先按照7:3的比例将预处理后的数据随机划分成训练集和测试集, 采用相同的训练集数据对不同方法的模型进行预测, 应用模型对相同的测试集进行验证, 测试模型性能。 根据测试的结果显示SVM 和LDA的预测准确率较高, 对六种分枝杆菌的分类效果较好, 预测准确率分别为99.4%和98.8%, 而且几乎所有分类器的分类效果都能达到80%以上(表3)。
| 表3 六种分类器的分类测试结果 Table 3 Results of six identification models |
提取两个模型的混淆矩阵后可以发现, SVM模型仅有一个单细胞预测错误, 将堪萨斯分枝杆菌分类成土分枝杆菌, 预测准确率为97.96%(48/49); 其他种类的分类效果达到了100%(表4)。 而LDA模型有两个单细胞预测错误: 将1个脓肿分枝杆菌细胞分类成偶发分枝杆菌, 预测准确率为95.65%(22/23); 将1个戈登分枝杆菌细胞分类成土分枝杆菌, 预测准确率为96.30%(26/27), 对其他分枝杆菌的分类效果可以达到100%(表5)。
| 表4 SVM的混淆矩阵 Table 4 Prediction results of SVM confusion matrix |
| 表5 LDA的混淆矩阵 Table 5 Prediction results of LDA confusion matrix |
通过对六种不同的分类器的效果比较, 提出一种基于特征拉曼光谱训练SVM模型的方法来鉴定六种非结核分枝杆菌。 在利用已知种类的单细胞拉曼光谱样本构建数据库的基础上, 可以实现对未知非结核分枝杆菌样本的鉴定。 单细胞拉曼技术有快速、 简易、 成本低等优势, 未来通过大量临床样本的检验及数据库的扩充, 具备为临床医生提供更快速、 便捷、 低成本的临床辅助诊断方案的巨大潜力。
此外, 还可以结合拉曼分选与单细胞测序(RACS-Seq), 验证拉曼光谱分类和鉴定的结果, 并揭示全基因组水平、 单碱基精度的遗传信息[16]。 针对大肠杆菌, 我们前期已经证明, 利用RAGE-Seq能从临床尿液样本中利用单细胞拉曼光谱直接识别和分选出耐受特定抗生素的临床大肠杆菌, 并进行了精确到一个细胞的全基因组测序, 覆盖度可达99.5%[17]。 这一高覆盖保证了基因组上所有耐药基因突变均得以全面、 精确地揭示。 因此, 本研究基于拉曼光谱的NTM分类和鉴别, 为一个针对NTM的 RACS-Seq奠定了基础。 这种在单个病原微生物细胞精度的临床分析, 代表着生命单元和进化单位水平的病原识别、 基因组追踪与药敏机制研究, 因此对于新一代的疾病防控平台具有重要的意义。
综上, 显微共聚焦单细胞拉曼光谱技术与各种机器学习的方法可以对生物样本进行种类鉴定, 从生物大分子的结构、 组成及相对含量等方面给出依据, 可为临床诊断及微生物学研究提供有力的技术支撑。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|