作者简介: 王楚彪, 1982年生, 国家林业和草原局桉树研究开发中心博士研究生 e-mail: scauwcb@163.com
摸清粗皮桉( Eucalyptus pellita)群体的遗传亲缘关系, 对研究桉树杂交育种理论, 开发优良新品种具有非常重要的意义。 研究意在通过对比粗皮桉种源遗传差异与其光谱差异间的关系, 探索近红外光谱(NIRs)技术用于粗皮桉遗传亲缘关系分析的可行性。 以粗皮桉天然种源材料为对象, 每个种源采集8~12个家系叶样。 通过全基因组重测序, 基于核苷酸序列差异计算种源间的遗传距离。 同时, 每个家系选择4~6片健康叶片烘至绝干后, 将其粉碎装于透明自封口塑料袋。 用手持式近红外仪Phazir Rx (1624)采集样品的NIRs信息。 以簇类独立软模式(SIMCA)判别分析统计对比种源到目标种源的光谱距离, 并基于NIRs欧氏距离对种源进行层级聚类。 以NIRs的PCA因子得分图分析种源间的遗传亲缘关系及其遗传变异。 结果显示, 粗皮桉新几内亚岛种源间的平均遗传距离为0.186, 昆士兰州种源间的平均为0.157, 新几内亚岛种源与昆士兰州种源间的平均遗传距离为0.295, 明显大于区域内种源间的遗传距离。 粗皮桉2大区域种源间的NIRs光谱距离与其种源间遗传距离基本呈正相关关系。 基于NIRs的层级聚类在一定程度上印证了种源遗传距离、 光谱距离的大小关系, 但与其地理距离非完全对应, 说明基因交流对粗皮桉群体的遗传亲缘关系有较大的影响。 PCA聚类显示, 遗传或光谱距离大的种源样本因子得分图存在严重重叠, 而遗传或光谱距离小的种源样本因子得分反而会清晰聚类, 这表明NIRs信息区分异质样本的敏感性, 同时也反映了粗皮桉种源内家系间遗传变异的大小。 研究结果表明, NIRs技术能够真实反映粗皮桉种源间的遗传差异, 可用于桉树群体遗传亲缘关系及群体内的遗传变异分析, 可辅助桉树群体的世代改良研究。
Clarifying the pedigree on Eucalyptus pellita populations is of great significance for studying rules of interspecific hybridization of eucalypt and the development of excellent new eucalypt genotypes. The purpose of the present study was to assess the accuracy and reliability of near infrared spectroscopy (NIRs) used in the analysis of the pedigree of E. pellita populations by comparing the relationship between genetic variations and NIRs differences that. The genetic materials involved natural provenances from the E. Pellita population, fresh leaves of 8~12 families were collected from each provenance. The DNA information of materials was obtained through whole-genome resequencing. Firstly, the genetic distances among provenances were evaluated with the DNA nucleotide sequence differences between samples. Meanwhile, four to six healthy leaves of each sample were placed in a drying ovenuntil completely dry. The dried leaves were milled and then put into a transparent self-sealing plastic bag. A portable NIR device, phazir RX (1 624), was used to take the NIRs information of samples. The NIRs spectral distance between validating provenance and calibrating provenance was estimated with the soft independent modeling of class analogy (SIMCA). Hierarchical clustering was performed for all provenances with NIRs Euclidean distance. PCA scores plots of provenances NIRs demonstrated the pedigree and the genetic variations of provenances. The results showed that the total mean of the genetic distance of provenances from New Guinea Island and Queensland were 0.186 and 0.157 respectively, the total mean of genetic distance between New Guinea Island and Queensland was 0.295, which was higher than that within each separate district significantly. There was a positive correlation between NIRs spectral distance and genetic distance between provenances in two separate districts, but a negative correlation was also found between some provenances of E. pellita. The correlation between genetic distance and NIRs spectral distance was also proved by the NIRs Hierarchical clustering of all provenances. However, the clustering did not completely correspond with their geographical distance of provenances, suggesting that gene flow of some forms greatly affects the genetic relationship among separate districts of E. pellita populations. The PCA score plots demonstrated that PCs plots of some provenances with large genetic distance or NIRs spectral distance would overlap seriously, and PCs plots of some provenances with close genetic distance or NIRs spectral distance would be clustered, which verified the sensitivity of NIRs in the distinguishing of heterogeneous samples, also showed the genetic variation among families inprovenance of E. pellita. All the current study results proposed that NIRs could genuinely reflect the genetic differences among provenances of E. pellita, and could be used to analyze the genetic relationship and genetic variation within eucalypt populations, and could be used to assist the improvement of eucalypts breeding populations in a generation.
粗皮桉天然分布于新几内亚岛南部和澳大利亚昆士兰州北部, 其不同种源的生物学特征和生长适应性均具有很大的遗传差异[1]。 在炎热潮湿的热带地区, 粗皮桉良好的抗病虫害能力使其逐渐取代金合欢属(Acacia)树种, 成为地区重要的造林树种[2]。 巴西、 东南亚等国对粗皮桉进行了大量的引种和高世代改良[3]。 国内系统的粗皮桉引种改良始于20世纪80年代, 并于2011年在两广多地建立了其高世代育种群体[4]。 粗皮桉在国内主要作为杂交亲本用于开发桉树优良无性系, 并针对其开展速生、 抗病虫害等杂交育种理论研究。
摸清育种群体的遗传亲缘关系是推动粗皮桉群体世代改良的基础工作。 目前常用的表型数据分析方法很难精确估算家系的遗传和环境分量。 DNA生物信息学分析专业要求高, 程序复杂且成本高。 鉴于此, 本研究计划通过对比基于DNA分析的遗传距离和基于NIRs分析的光谱距离间的关系, 探索近红外光谱技术用于粗皮桉群体遗传基础分析的精度和可行性。
研究对象为粗皮桉育种群体试验中的23个天然种源(表1)。 遗传材料基本涵盖了粗皮桉的天然原生地, 即澳大利亚昆士兰州(QLD, AUS)北部和新几内亚岛(New Guinea Island)南部(印度尼西亚(Indonesia)和巴布亚新几内亚(Papua New Guinea, PNG))。 两地被大洋分隔, 最近的海岸线距离约为150 km。 在粗皮桉大田群体试验中, 每家系采集10~15片新鲜叶样, 每个种源采样8~12个家系代表该种源。 叶样尽快置于-30 ℃冰箱, 用于后续遗传分析及NIRs信息采集。
| 表1 粗皮桉23个天然种源信息 Table 1 Details of 23 natural provenances of E.pellita studied |
手持式近红外仪Phazir Rx (1624) (Polychromix, Thermo Scientific, USA)用于叶样NIRs的采集。 Phazir Rx波长范围为1 600~2 400 nm, 分辨率12 nm, 单次扫描获得100维向量数据, 自带背景校正, 内置可编程的MEMS技术微衍射光栅。
1.3.1 光谱采集
光谱采集样本与DNA分析样本完全对应, 每个家系选择4~6片外形健康的叶片, 置于ClassicLine干燥箱(BINDER, 德国), 设置60 ℃, 72 h, 以至绝干。 用中药打粉机粉碎后装入透明自封口塑料袋。 用Phazir Rx (1624)隔袋扫描5次, 其均值代表该样品的近红外NIRs信息[5], 每个种源共获得8~12条NIRs。
1.3.2 NIRs数据的预处理和分析
经不同的预处理效果对比后, 本研究对NIRs原始数据进行Savitzky-Golay平滑的二阶导数预处理[6, 7]。 采用全交互式内部交叉验证算法, 建立目标种源的主成分分析(PCA)模型, 通过簇类独立软模式(SIMCA)判别分析, 统计对比种源到目标种源模型间的NIRs光谱距离。 基于NIRs欧氏距离(Euclidean distance)按全链接算法(Complete linkage), 对测试种源进行层级聚类(Hierarchical clustering)。 根据样本NIRs数据PCA的因子得分图分析种源间的遗传亲缘关系及其遗传变异。 NIRs数据的预处理和分析均由The Unscrambler x10.4 (CAMO, Oslo, Norway)实现。
1.3.3 样本遗传距离计算
通过全基因组重测序(Whole Genome Resequencing)方法获得粗皮桉种源各家系的DNA信息, 根据Kimura提出的核苷酸序列差异原理, 使用Mega-X软件估算粗皮桉种源间的遗传距离[8, 9]。
粗皮桉新几内亚岛7个种源间的平均遗传距离为0.186, 昆士兰州16个种源间的平均为0.157(数据冗余, 未列出)。 表2为新几内亚岛与昆士兰州2大区域种源间的遗传距离。 数据显示, 2大区域种源间的最小、 最大及平均遗传距离分别为0.229, 0.370和0.295, 明显大于区域内种源间的遗传距离。 新几内亚岛种源A20659, S16120, S16121和S16122与昆士兰州各种源间的遗传距离均超过了0.300。 2大区域内和区域间种源间遗传距离的差异, 说明区域分隔(海洋)明显拉大了粗皮桉种源间的遗传亲缘关系。
| 表2 粗皮桉新几内亚岛种源与昆士兰州种源间的遗传距离 Table 2 The genetic distance of provenances between New Guinea Island and Queensland of E. pellita |
利用NIRs光谱距离可以从不同角度解析、 印证样本间的内在遗传差异。 表3为粗皮桉新几内亚岛种源与昆士兰州种源间的NIRs光谱欧式距离。 数据显示, 2大区域种源间NIRs平均光谱距离大小与其遗传距离的大小趋势基本一致, 如种源S16120与昆士兰州种源间的平均遗传距离最大(0.370), 两者之间的平均光谱距离(0.220)也明显大于其他种源间的光谱距离。 偶有例外, S16122与昆士兰州种源间的平均光谱距离就与其遗传距离的大小关系相悖。 种源A17854与昆士兰州种源间的NIRs光谱距离偏大可能与其有效样本偏少有关。
| 表3 粗皮桉新几内亚岛种源与昆士兰州种源间的NIRs欧氏距离 Table 3 NIRs Euclidean distance of provenances between New Guinea Island and Queensland of E. pellita |
因实际值太小, 表中所列光谱距离为实际数值放大100倍后的结果。
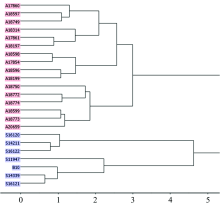
图1中粗皮桉天然种源的NIRs层级聚类显示, 23个种源的聚类效果在一定程度上印证了其遗传距离、 光谱距离的差异。 巴布亚新几内亚岛的S16120, S16121和S16122与昆士兰州种源间的遗传距离和光谱距离均大于其他种源间的两类距离, 也基本显示了与昆士兰州种源的分别聚类。 但23个种源并没有完全按照巴布亚新几内亚岛和昆士兰州2大地域分别聚类, 来自巴布亚新几内亚的种源A18197, A17854, A18199及A20659与多数来自昆士兰州的种源聚在了一起。 这或与几个种源的海拔较低(< 50 m), 且两大地域间的地理距离比较接近, 地域间存在花粉、 种子或传粉媒介等某种形式的基因交流有关。
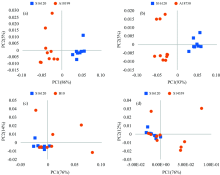
由于巴布亚新几内亚岛种源与昆士兰种源间的遗传距离普遍较大, 因此, 主要列举了2大区域种源间的PCA主因子得分聚类结果。 图2中巴布亚新几内亚岛内种源S16120与A18199间(a), 及S16120与昆士兰州种源A18750间(b)相同主因子的因子得分显示, 不仅不同地理区域的种源可以清晰聚类(S16120与A18750), 同一地理来源的种源也可以清晰地区分(S16120与A18199)。 对比种源间的遗传距离大小及其PCA主因子的得分图, 遗传距离小的种源间其因子得分有可能将其清晰地区分; 而遗传距离大的种源间, 其因子得分有可能存在严重的重叠, 这可能与种源内家系间的遗传变异较大有关。 研究中大多数种源间的PCA主因子得分都显示出清晰的聚类, 为免冗余, 未一一展示。
图2显示种源S16120与B10(c)和S14339(d)不能清晰区分, 这与它们之间具有较大的遗传距离不符, 可能是种源B10和S14339内家系间存在较大的遗传变异, 从其非常分散的因子得分图也能看出。 另外, 图2各种源因子得分的聚集度各不相同, B10和S14339的得分比较分散, 而S16120, A18199和A18750的得分则相对集中, 影响了种源间的相互区分。 从遗传角度也反映了种源内家系间的遗传变异信息, 分散可能表明其遗传变异大, 集中则表示遗传变异小。
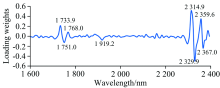
图3是种源S16120—A17861间PCA聚类分析时第一主因子在不同波段的因子载荷。 图中标出的8处特征峰代表的化学键、 化合物或是导致样本差异的来源。 参阅文献[10, 11]可知, 波长1 731~1 768 nm表征C—H伸缩、 C—H和S—H一级倍频吸收峰; 1 919 nm表征水的吸收峰及C=O的伸缩吸收峰; 2 310和2 328~2 332 nm表征C—H伸缩、 C—H变形吸收峰, 对应化合物为半纤维素; 2 361 nm左右表征C—H伸缩、 C—H变形及C—H2一级倍频弯曲振动吸收峰, 对应化合物为纤维素。 特征峰分析的意义在于直接建立其与目标物质间的模型, 以优化模型结构和精度。 另外, 对于林木遗传育种研究, 特征峰也为利用NIRs更好地辅助分析材料的遗传差异或性状变异来源提供了方向。
对比粗皮桉天然种源间的遗传距离与其NIRs光谱距离间的关系发现, 种源遗传距离与其光谱距离间普遍存在正相关关系, 但个别种源间的遗传距离与其光谱距离呈负相关。 种源NIRs聚类结果与地理距离间的关系也非完全对应, 这印证了粗皮桉群体不同地域间的基因交流对其遗传亲缘关系有较大的影响。 NIRs数据的PCA聚类显示, 遗传或光谱距离大的种源间存在严重重叠, 而遗传或光谱距离小的种源样本反而会清晰聚类, 这既表明了NIRs信息的敏感性, 也在一定程度反映了粗皮桉各种源内存在不同水平的遗传变异。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|