作者简介: 冯 帅, 1992年生, 沈阳农业大学信息与电气工程学院博士研究生 e-mail: int_crazy@163.com
为提供一种高效、 快速和无损的粳稻叶片氮素含量反演方法, 以粳稻小区试验为基础, 利用高光谱技术和室内化学实验, 获取粳稻分蘖期、 拔节期和抽穗期三个生育期共280组叶片高光谱数据以及相对应的水稻叶片氮素含量数据, 分析不同施氮水平的粳稻叶片光谱特征, 采用随机青蛙算法(random_frog)与迭代和保留信息变量算法(IRIV)相结合的方式筛选特征波段, 并将任意两个光谱波段随机组合构建差值植被指数DSI( Ri, Rj)、 比值植被指数RSI( Ri, Rj)和归一化植被指数NDSI( Ri, Rj), 分别将较优的特征波段组合和植被指数组合作为模型输入, 构建BP神经网络、 支持向量机(SVR)和非支配的精英策略遗传算法优化极限学习机(NSGA2-ELM)粳稻叶片氮素含量反演模型, 并对模型进行验证分析。 结果表明: 随着施氮水平的增加, 粳稻叶片近红外波段范围反射率逐渐升高, 在可见光波段范围反射率逐渐降低。 采用random_frog与IRIV相结合的方式筛选特征波段共得到8个特征波段, 其中可见光波段7个, 分别为414.2, 430.9, 439.6, 447.9, 682.7, 685.4和686.3 nm, 近红外波段仅有1个为999.1 nm, 该方法较好地剔除了干扰信息, 大大降低了波段间的共线性。 同时从三种植被指数(DSI( Ri, Rj), RSI( Ri, Rj), NDSI( Ri, Rj))与粳稻叶片氮素含量的决定系数等势图中可知, DSI( R648 .1, R738 .1), RSI( R532 .8, R677.3)和NDSI( R654 .8, R532 .9)与叶片氮素含量相关性最好, R2分别为0.811 4, 0.829 7和0.816 9。 在输入参量不同的建模效果对比分析中, 以特征波段组合作为模型输入所构建的模型反演效果略优于植被指数组合, R2均大于0.7, RMSE均小于0.57。 而在反演模型间的对比分析中, 提出的NSGA2-ELM反演模型的估测效果要优于BP神经网络模型和SVR模型, 训练集决定系数 R2为0.817 2, 均方根误差RMSE为0.355 5, 验证集 R2为0.849 7, RMSE为0.301 1。 鉴于此, random_frog-IRIV筛选特征波段方法结合NSGA2-ELM建模方法在快速检测粳稻叶片氮素含量中具有显著优势, 可为粳稻田间精准施肥提供了参考。
In order to provide an efficient, rapid and non-destructive Inversion method for the nitrogen content of japonica rice leaves, based on the japonica rice plot experiment, using high spectrum technology and laboratory chemistry experiments to obtain the effective data for three growth periods of japonica rice in the tillering stage, joining stage and heading stage. A total of 280 sets of leaf high spectrum data and corresponding rice leaf nitrogen content data were obtained to analyze the spectral characteristics of japonica rice leaves with different nitrogen treatment levels. The random frog algorithm (Random_frog) is combined with the iteratively retaining informative variables algorithm (IRIV) to screen the feature bands, and any two spectral bands are randomly combined to construct the difference vegetation index DSI ( Ri, Rj), ratio vegetation index. RSI ( Ri, Rj) and normalized vegetation index NDSI ( Ri, Rj), respectively, combine the superior feature band combination and vegetation index as model inputs. Therefore, BP neural network, support vector machine (SVR) and non-dominated elite strategy genetic algorithm optimization limit learning machine (NSGA2-ELM) japonica rice leaf nitrogen content Inversion model were constructed, and the model was verified and analyzed. The results showed that with the increasing level of nitrogen fertilizer treatment, the reflectivity of the japonica rice leaves in the near-infrared range gradually increased, while the reflectance decreased gradually in the visible range. A total of 8 characteristic bands were obtained by combining Random_frog and IRIV. Among them, there are 7 visible light bands, which are 414.2, 430.9, 439.6, 447.9, 682.7, 685.4 and 686.3 nm, respectively. Only one of the near-infrared bands is 999.1 nm. This method effectively eliminates interference information and greatly reduces the collinearity between the bands. At the same time, it can be analyzed from the three vegetation index (DSI ( Ri, Rj), RSI ( Ri, Rj), NDSI ( Ri, Rj)) and the determination coefficient of the nitrogen content of the japonica rice leaves. DSI ( R648 .1, R738 .1), RSI ( R532 .8, R677 .3) and NDSI ( R654 .8, R532 .9) have the best correlation with leaf nitrogen content, R2 are 0.811 4, 0.829 7 and 0.816 9, respectively. In the comparative analysis of the input parameters with different input parameters, the model Inversion with the feature band combination as the model input is slightly better than the vegetation index combination, R2 is greater than 0.7, and the RMSE is less than 0.57. In the comparative analysis between the Inversion models, the estimation effect of the NSGA2-ELMInversion model proposed in this paper is significantly better than the BP neural network model and the SVR model. The training set determination coefficient R2 is 0.817 2, and the root means square error RMSE is 0.355 5. The set R2 is 0.849 7 and the RMSE is 0.301 1. According to the results of this study, the Random_frog-IRIV screening characteristic band method combined with NSGA2-ELM modeling method has a significant advantage in rapidly detecting the nitrogen content of japonica rice leaves. The research results can provide a theoretical reference for field precision fertilization of japonica rice.
氮素是农作物生长发育过程中重要的营养成分, 实时监测和评估农作物的氮素含量对于农作物田间精准管理和长势预测等均具有十分重要的意义[1]。
目前, 采用高光谱检测法对果蔬[2, 3]和粮食作物[4, 5, 6]的氮素营养诊断已成为国内外学者研究的主要内容。 王树文[7]等研究表明基于主成分分析和相关分析结合多元回归分析模型的差值指数、 多变量单波段指数等模型反演效果较好, 预测集R2为0.869, RMSE为0.085。 刘明博[8]等采用连续投影法(SPA)筛选的有效波段、 光谱指数RVI、 NDVI以及全光谱波段构建多种水稻叶片氮素含量反演模型。 对比发现, 基于SPA有效波段构建的模型的估测效果明显优于光谱指数所建, 但略差于全光谱波段所建模型。 Tian[9]等通过分析多种高光谱植被指数与水稻叶片氮素含量的定量关系, 得出采用绿色比率指数SR(R553, R537)反演叶片氮素含量具有最佳估测精度。 Du[10]等采用高光谱激光雷达(HSL)技术构建两种积分指数NOAC和RII反演水稻叶片全氮含量(LNC)。 方美红[11]等采用小波系数构建水稻叶片氮含量反演模型, 研究表明该模型有较高估测精度, 预测值与估测值的复相关系数高达0.99, 显著优于传统光谱指数反演模型。
粳稻生长过程中受氮肥影响较为明显, 不同施氮水平下的粳稻生长趋势有一定的差异, 叶片对自然光的吸收和反射也表现出不同的变化, 进而对粳稻产量产生较大影响。 鉴于此, 本研究为实现粳稻叶片氮素含量的高效、 快速和精准反演, 分别采用特征波段组合和光谱植被指数组合作为输入, 构建3种氮素含量反演模型并对比分析, 进而提出基于高光谱的粳稻叶片氮素含量反演方法, 以期为粳稻氮素营养诊断和田间精准管理提供科学依据和理论支持。
粳稻小区试验于2018年6月至8月在辽宁省沈阳市沈河区沈阳农业大学南区水稻试验基地(118° 53'E, 38° 43'N, 平均海拔40 m)开展, 供试品种为沈稻9816。 共划分12个小区, 每个试验小区面积为30 m2(7.61 m× 4.20 m), 如图1所示。 人为设置小区试验无肥、 低氮、 正常和高氮4种情况, 共设4个施氮梯度, 分别为N0(不含氮)、 N1(150 kg· hm-2)、 N2(240 kg· hm-2)、 N3(330 kg· hm-2), 每个水平3次重复。 同时各试验小区之间采取隔离措施, 保证小区之间水肥不互相渗透, 其他田间管理水平均按当地正常水平进行。
采用美国海洋光学公司生产的光纤光谱仪HR2000+, 其光谱范围为400~1 000 nm, 光谱分辨率为0.45 nm。 按标号将磨碎的叶片粉末放置于操作平台, 光谱探头紧压在叶片粉末上; 测量前通过黑白板校正。 通过自带OceanView软件完成粳稻叶片高光谱数据的采集。
从每个试验小区采样点获取粳稻不同部位叶片20片左右, 分别装入自封袋中并标注小区名称和编号, 立即带回实验室。 在室内, 首先对叶片进行洗涤, 去除叶片表面灰尘等无用物质, 然后在105 ℃条件下杀青30 min, 并在70 ℃的烘箱中烘干至恒量, 称量粉碎。 最后采用凯氏定氮法测定粳稻叶片氮素含量。 所采集的数据样本中去除粗大误差, 最终共得到粳稻叶片氮素含量有效数据280组, 其概率密度函数如图2所示。
 | 图2 粳稻280组叶片氮素含量的概率密度函数Fig.2 Probabilistic density function of nitrogen content in 280 groups of japonica rice leaves |
由图2可知, 280组粳稻叶片氮素含量数据呈正态分布, 均值为2.860 mg· g-1, 最大值为4.530 mg· g-1, 最小值为1.060 mg· g-1, 标准差为0.825 mg· g-1, 变异系数为28.846%, 满足氮素含量反演要求。 同时采用Kennard-Stone算法(KS)将样本按照训练集与验证集3:1的比例进行划分, 其氮素含量统计表如表1所示。
| 表1 训练集与验证集粳稻叶片氮素含量数据统计表 Table 1 Statistical table of nitrogen content in leaves of japonica rice in training set and validation set |
1.4.1 光谱特征波段筛选方法
高光谱数据具有较高的分辨率, 因此在光谱数据中包含了大量的数据信息, 但在庞大的数据信息中仍存在一定的冗余信息, 给后续的研究带来了一定困扰。 筛选特征波段进可减少数据冗余, 获得对模型起关键性作用的特征波段是不可或缺的。 分别采用随机青蛙法(random frog)与迭代和保留信息变量算法(iteratively retaining informative variables, IRIV)两种方法筛选特征波段。 随机青蛙法(random frog)采用蒙特卡洛法通过维度转换进行采样, 以每个波段变量的选择频率作为剔除冗余变量的依据, 最终得到最佳特征波段。 迭代和保留信息变量算法是基于二进制矩阵重排过滤器提出的特征变量选择算法, 通过多次迭代, 计算包含某一波段和不包含该波段变量时的RMSECV平均值, 得到两个均值之差DMEAN和曼-惠特尼检验的P值, 从而判断波段的重要性, 其判断规则如表2所示[12], 并依据该规则消除无信息和干扰波段变量, 保留强和弱波段变量, 最后通过反向消除获得最佳特征波段。
| 表2 迭代和保留信息变量算法判断规则 Table 2 The judgment rule of iteratively retaining informative variables |
1.4.2 光谱植被指数的构建
植被指数常常被用来精准监测植被生长状况和生物量信息等植被信息, 同时具有增强植被信息, 减弱非植被信息的作用, 因此选取3种对粳稻叶片氮素含量具有较好反演效果的高光谱植被指数, 即比值植被指数(ratio spectral index, RSI)、 差值植被指数(difference spectral index, DSI)以及归一化植被指数(normalized difference spectral index, NDSI)。 其计算公式如式(1)— 式(3)
式中, Ri与Rj分别表示在400~1 000 nm范围内的光谱反射率值。
分别将特征波段组合和植被指数作为模型输入, 实测粳稻叶片氮素含量作为输出, 构建BP神经网络、 支持向量机和NSGA2-ELM三种反演模型。 由于ELM模型采用随机生成输入权重和隐含层偏向值, 这将导致ELM输出波动大, 模型不稳定。 因此提出一种将NSGA2算法与ELM模型相结合的混合模型算法。 具体NSGA2优化过程如下:
(1)根据样本数据集确定ELM的网络拓扑结构, 将神经元之间的权重和偏向值构成实数向量, 用以表示种群M中的个体。 同时随机生成实数向量的初始值构成大小为N的第一代父代种群P。
(2)对父代种群进行非支配排序, 并采用传统的遗传算法对父代种群进行选择、 交叉和变异操作产生大小为N的子代种群P1。 将种群P和P1合并为大小为2N的种群B。
(3)对种群进行非支配排序, 获得非支配解的前端Ft, 即为非支配面的F1, F2和F3, 并计算拥挤度。 之后采用精英保留策略筛选最优个体, 即由于子代和父代种群个体均包含在种群B中, 则采用非支配排序后的F1中的个体为种群B中最佳的, 因此现将F1全部个体放入新父代种群P3中。 若P3小于N, 则继续将F2中的个体加入种群P3中。 若P3仍小于N, 则对F3进行拥挤度排序, 取N-|P3|个种群个体添加至P3, 直至P3种群大小为N。 然后采用遗传算法对种群P3进行选择、 交叉和变异产生新的种群P4。
(4)重复n次上述计算过程, 达到设定的最大迭代次数则停止迭代, 得到最佳ELM的最佳权重和偏向值, 完成优化。
同时为了检测粳稻叶片氮素含量反演模型的准确性和可靠性, 采用决定系数R2和均方根误差RMSE两个评价指标检测模型的拟合效果和估测能力。 其计算公式如式(4)和式(5)
式中, yi为真实值;
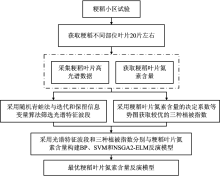
整体技术路线如图3所示。
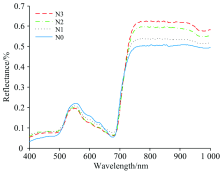
由图4可知, 在不同施氮水平下的粳稻叶片光谱反射率曲线变化趋势大致相同。 在可见光波段范围(400~770 nm)光谱反射率均呈现出“ 先升后降” 的变化规律。 同时, 在绿波段(550 nm附近)出现明显的反射峰, 在红波段(680 nm附近)出现明显的吸收谷。 在680~770 nm范围内不同施氮水平的光谱反射率曲线变化基本一致, 反射率急剧升高。 在近红外波段范围(> 770 nm)形成较高的反射平台。 然而, 在可见光和近红外波段范围, 各个施氮水平的叶片光谱反射率表现出相反的变化规律。 在可见光波段范围, 光谱反射率随着施氮水平的升高而减小, 尤其在“ 红峰” 处极其明显, 在近红外波段范围, 光谱反射率随着施氮水平的升高而增大, 尤其在“ 近红外反射平台” 处较为明显。
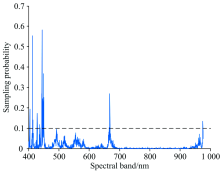
鉴于高光谱的波段数较多, 且存在无用和干扰波段, 对建模的效率和精度产生较大影响。 因此首先采用随机青蛙算法(random-frog)初选特征波段, 去除冗余信息。 确定random-frog算法的潜在变量的最大数为6, 初始采样变量数为1 000, 但由于random-frog算法以蒙特卡洛法为筛选原理, 每次筛选出的特征波段略有差异。 因此共运行random-frog算法50次, 最后取结果的平均值作为特征波段的判断依据, 其结果如图5所示。 由图5可知, random-frog算法筛选的结果较好, 大部分光谱波段的选样概率较小, 小部分光谱波段的选样概率较大。 同时, 设定筛选阈值为0.1, 选样概率大于0.1的波段被认定为特征波段, 最终筛选出23个波段作为特征波段。
random-frog方法筛选得到23个特征波段, 波段数量较大, 且random-frog方法在筛选过程具有随机性, 每次的筛选结果略有差异, 可能仍存在无信息波段。 因此, 为提高模型的稳健性和准确性, 采用迭代和保留信息变量算法(IRIV)对波段进行进一步筛选, 消除冗余信息和无用信息。
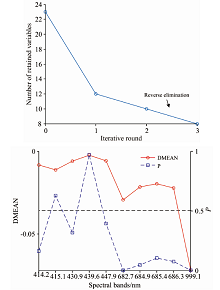
通过多次测试确定IRIV算法的最大主因子数为10, 交叉验证次数为5。 IRIV算法筛选特征波段共进行3轮, 如图6(a)所示。 前2轮波段变量个数逐渐减少, 从23个波段减少到10个, 其中在第2轮迭代过程中基本剔除了无用信息波段和干扰波段, 余下波段的DMEAN和P值如图6(b)所示, 在第3轮采用反向消除, 最终得到8个特征波段, 其中弱波段变量仅有1个为439.6 nm, 强波段变量仅有7个, 分别为414.2, 430.9, 447.9, 682.7, 685.4, 686.3和999.1 nm。 此时, 大部分波段集中于400~700 nm之间, 即在可见光波段区域之间有7个特征波段, 仅有1个特征波段位于近红外波段区域。 从筛选结果来看, 虽然各个波段间仍存在少量的共线性, 但相比于初次筛选, 已较好地降低了波段间的共线性和冗余信息。
图7为400~1 000 nm中任意两个光谱波段随机组合构成的DSI(Ri, Rj), RSI(Ri, Rj)和NDSI(Ri, Rj)与粳稻叶片氮素含量的决定系数等势图。 从决定系数等势图中可知与叶片氮素含量相关性最优的光谱植被指数和相关性最好的波段范围。 在图7(a)中, 对于DSI(Ri, Rj)而言, 在707.3~852.5与625.5~692 nm的光谱波段组合与叶片氮素含量较好, 决定系数R2均大于0.7, 其中DSI(R648.1, R738.1)与叶片氮素含量的相关性最好, R2为0.811 4。 相比DSI(Ri, Rj)而言, RSI(Ri, Rj)与叶片氮素含量相关性较好的波段范围较窄, 多集中于可见光波段范围[图7(b)]。 其中在596.1~686.2和507.2~607.4 nm的波段组合相关性最佳, R2均大于0.7, 最好的波段组合构建的RSI植被指数为RSI(R532.8, R677.3), R2为0.829 7。
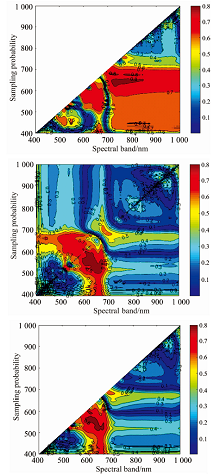 | 图7 任意两波段组合构成的DSI (a), RSI (b)和NDSI (c)与叶片氮素含量的决定系数等势图Fig.7 Contour of R2 between DSI (a), RSI (b), NDSI (c) and leaf nitrogen content in any two bands |
图7(c)为任意两个波段组合构建的NDSI(Ri, Rj)与叶片氮素含量的决定系数等势图。 在601.5~682.7与514.1~609.2 nm的光谱波段组合与氮素含量的相关性较好, R2大于0.7, 其中NDSI(R654.8, R532.9)与叶片氮素含量相关性最好, R2为0.816 9。
2.4.1 BP神经网络反演建模
采用random-frog和IRIV算法筛选的8个特征波段和全生长期的3种较好的植被指数(DSI(R648.1, R738.1), RSI(R532.8, R677.3)和NDSI(R654.8, R532.9))分别作为模型输入, 粳稻叶片氮素含量作为模型输出, 构建BP神经网络反演模型。 将Tansig和Purelin法分别设置为隐含层和输出层的传递函数, 将Trainlm法设置为训练函数, 训练最大迭代次数为1 000次, 学习速率lr和训练精度goal分别均为0.1和0.01。 同时, 通过逐步试验得出隐含层神经节点个数为6时, 模型估测效果最佳。 建模结果如图8所示。
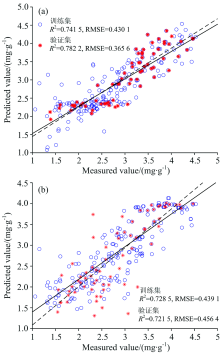 | 图8 BP神经网络建模结果 (a): 特征波段组合; (b): 植被指数组合Fig.8 Modeling results of BP neural network (a): Characteristic band combination; (b): Vegetation index combination |
由图8可知, 以8个特征波段组合作为输入构建的BP神经网络反演模型, 其建模效果要优于植被指数组合。 模型训练集的决定系数R2和均方根误差RMSE分别为0.741 5和0.430 1, 验证集的R2和RMSE分别为0.782 2和0.365 6。 由此可见, 采用8个特征波段组合作为BP神经网络的输入进行建模, 提高了反演粳稻叶片氮素含量的预测能力。
2.4.2 支持向量机反演建模
分别采用特征波段组合和植被指数组合作为支持向量机(SVR)模型的输入, 构建粳稻叶片氮素含量反演模型。 将SVR的核函数设置为Sigmoid, 特征波段组合和植被指数组合的惩罚因子c分别为1.3和2, Sigmoid核函数的系数g分别为10.1和9, 损失函数值分别为0.17和0.1, SVR建模结果如图9所示。
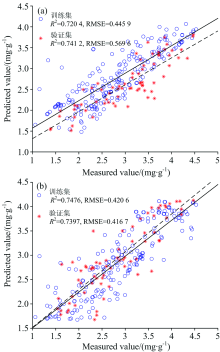 | 图9 支持向量机建模结果 (a): 特征波段组合; (b): 植被指数组合Fig.9 Modeling results of SVR (a): Characteristic band combination; (b): Vegetation index combination |
由图9比较可知, 无论是以特征波段组合还是以植被指数组合作为输入所构建的SVR模型, 可靠性和估测能力具有一致性。 从验证集的建模结果来看, 特征波段组合的SVR模型的R2略高于植被指数组合, 但其RMSE略低于植被指数组合。 特征波段组合和植被指数组合的训练集R2和RMSE分别为: 0.720 4, 0.445 9和0.747 6和0.420 6, 验证集R2和RMSE分别为: 0.741 2, 0.569 6和0.739 7和0.416 7。
2.4.3 非支配的精英策略遗传算法优化极限学习机反演建模
采用random-frog和IRIV算法筛选的8个特征波段和全生长期的3种较好的植被指数(DSI(R648.1, R738.1), RSI(R532.8, R677.3)和NDSI(R654.8, R532.9))分别作为非支配的精英策略遗传算法优化极限学习机(NSGA2-ELM)模型的输入, 构建粳稻叶片氮素含量反演模型。 将特征波段组合和植被指数组合的NSGA2-ELM模型的种群个数均设置为30, 交叉概率和变异概率分别设置为0.96, 0.97和0.001, 0.001, 最大拥挤距离均为10 000, 保留最优个体比例均为0.1, 迭代次数均为50次。 建模结果如图10所示。
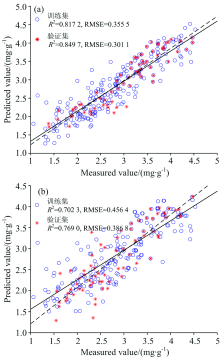 | 图10 NSGA2-ELM建模结果 (a): 特征波段组合; (b): 植被指数组合Fig.10 Modeling results of NSGA2-ELM==(a): Characteristic band combination; (b): Vegetation index combination |
由图10可知, 以特征波段组合作为NSGA2-ELM模型的输入构建模型的估测效果明显优于植被指数组合, 模型训练集的决定系数R2和均方根误差RMSE分别为0.817 2和0.355 5, 验证集的R2和RMSE分别为0.849 7和0.301 1。 相比BP神经网络模型和支持向量机模型, NSGA2-ELM模型无论是以特征波段组合还是以植被指数组合作为输入, 在模型精度和估测能力上都有显著提高, 说明采用NSGA2算法优化ELM模型对粳稻叶片氮素含量的预测具有较大的优势。 分析原因在于NSGA2算法能够保持种群的多样性, 并引入精英策略, 不仅能增大样本采集空间, 而且能较好地防止最优个体丢失, 也避免了类似BP神经网络等模型陷入过拟合和局部最优现象, 从而使模型具有更好的非线性映射能力和鲁棒性。 同时从不同输入类型来看, 3种反演模型多以特征波段组合作为输入, 模型估测效果较好。 说明使用random-frog和IRIV筛选出的特征波段具有较好的代表性, 包含了粳稻叶片高光谱的大部分特征。 因此采用random-frog和IRIV方法获取的特征波段所建立的反演模型, 具有更好的稳定性和估测能力。
以粳稻叶片高光谱数据为数据源, 先后采用随机青蛙算法与迭代和保留信息变量算法筛选特征波段, 并构建3种较优植被指数, 分别以特征波段组合和植被指数组合作为模型输入, 构建BP神经网络、 支持向量机和非支配的精英策略遗传算法优化极限学习机粳稻叶片氮素含量反演模型并比较其优劣。 主要结论如下:
(1)针对高光谱较多的光谱波段, 通过采用random-frog和IRIV算法相结合的方式筛选光谱特征波段, 共得到8个特征波段, 其中可见光波段有7个, 分别为414.2, 430.9, 439.6, 447.9, 682.7, 685.4和686.3 nm, 近红外波段仅有1个为999.1 nm。 结果表明能够较好地剔除无用和干扰光谱波段, 大大降低了波段间的共线性和冗余信息, 提高了建模效率和精度。
(2)分别采用特征波段组合和植被指数组合作为模型输入, 构建3种粳稻叶片氮素含量反演模型。 对比分析得出, NSGA2-ELM模型的估测效果要优于BP神经网络模型和SVR模型, 这是由于NSGA2算法能保持种群的多样性, 增大了样本空间, 防止最优个体丢失, 提高了ELM模型的估测精度和鲁棒性。 同时, 对比不同输入类型建模可知, 以特征波段组合作为输入, 模型估测效果较好, 验证集决定系数R2为0.849 7, 均方根误差RMSE为0.301 1。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|