作者简介: 于叶霞, 1995年生, 吉首大学生物资源与环境科学学院硕士研究生 e-mail: yyx921583014@163.com
青叶胆( Swertia leducii)为獐牙菜属( Swertia)一年生草本植物, 在肝炎病治疗方面效果显著。 其与同属近似种外观极其相似, 加之常以干燥全草入药, 仅从形态难以正确鉴别。 不同物种有效成分存在明显差异, 其药效也有所不同。 基于光谱和色谱数据融合建立青叶胆及近似种的鉴别方法, 为青叶胆药用真实性与安全性提供科学依据。 采集青叶胆及其近似种植物共102份样品的傅里叶变换红外光谱(FTIR)和超高效液相色谱(UPLC)指纹图谱; 利用标准正态变量(SNV)、 多元散射校正(MSC)、 Savitzky-Golay平滑(SG)、 一阶导数(1D)、 二阶导数(2D)等方法对原始红外光谱数据进行预处理, 通过系统聚类分析(HCA)探讨獐牙菜属不同种类样品化学信息相似性与差异性; Kennard-Stone算法将所有样品按2:1比例划分为训练集和预测集, 训练集基于FTIR, UPLC, 低级与中级数据融合建立随机森林(RF)判别模型, 预测集用于验证模型预测能力, 其中灵敏性(sensitivity)、 特异性(specificity)、 精密度(precision)和正确率(accuracy)用来评价模型性能。 结果显示: (1)采用SNV+SG+2D组合对FTIR数据进行预处理, R2 Y和 Q2最大, 分别为91.2%和84.1%, 所有类别被正确区分, 为最佳预处理。 (2)HCA反映了5种獐牙菜属植物样品分类情况与亲缘关系, 除紫红獐牙菜外, 其余4种獐牙菜植物均分类正确, 准确率为93.1%; 青叶胆、 川东獐牙菜、 紫红獐牙菜与西南獐牙菜亲缘关系较近。 (3)基于FTIR、 UPLC、 低级和中级数据融合策略建立RF判别模型, 样品错判总数分别为1, 5, 1和0, 中级数据融合效果最佳, 所有样品均正确分类, 所建模型性能良好。 FTIR与UPLC通过中级数据融合策略结合RF判别分析能正确鉴别不同种类獐牙菜属植物, 结合HCA分析能够明确青叶胆及其近似种之间的亲缘关系, 为獐牙菜属植物资源开发与质量控制提供理论基础。
Swertia leducii is an annual herbaceous plant of the genus Swertia. It has a remarkable high effective in treating liver inflammation. The appearance of S. leducii and the species of the same genus is very similar, and the whole dry herb of Swertia plants is often used as medicine. It is very difficult to correctly identify different species from the morphology. Nevertheless, It is different in treating effective due to different species with different chemical components. In this study, based on data fusion of spectra and chromatography, an effective identification method of S. leducii and its closest relative species was established to provide the scientific basis for authenticity and security of S. leducii medication. Fourier transform infrared (FTIR) and ultra performance liquid chromatography (UPLC) of 102 samples of Swertia were collected from 5 species. Standard normal variate (SNV), multiplicative signal correction (MSC), Savitzky-Golay smoothing (SG), first derivative (1D) and second derivative (2D) were used to treat raw spectral data. Then, the optimal spectral data was utilized to process Hierarchical cluster analysis (HCA) for analyzing the similarity and dissimilarity of genus Swertia with different species. Kennard-Stone algorithm was applied to divide 102 samples into the calibration set and validation set in accordance with 2:1 ratio. The calibration set was established the random forest (RF) discriminant model basing on FTIR, UPLC, low-level and mid-level data fusion, and the validation set was used to test the predictive ability of these models. In addition, the model performance was evaluated by sensitivity, specificity, precision and accuracy. The results indicated that: (1) SNV+SG+2D was the optimal pretreatment that all samples were correctly classified with the highest R2 Y (91.2%) and Q2 (84.1%). (2) HCA could reflect the classification and genetic relationship of S. leducii and its wild relatives. The other 4 species excepting S. punicea were correctly classified and its total accuracy rate reached 93.1%. S. punicea, S. cincta and S. davidii had closed relationship with S. leducii while S. angustifolia was relative far. (3) Comparing the FTIR, UPLC, low-level data fusion and mid-level data fusion, the number of error samples in the classification of RF analysis were 1, 5, 1 and 0, respectively. In the RF models, the best classification of mid-level data fusion with none error samples was better than other data matrices. Mid-level data fusion combined with RF methods can identify different species of genus Swertia and display the genetic relationship between S. leducii and its wild relatives. Besides, it could provide a theoretical basis for the development of plant resources and quality control of genus Swertia.
青叶胆(Swertia leducii)又名蒙自獐牙菜、 青鱼胆、 肝炎草等, 为龙胆科(Gentianaceae)獐牙菜属(Swertia)一年生草本植物, 集中分布在云南红河州地区[1]。 青叶胆化学成分主要有黄酮类、 环烯醚萜类、 三萜类和生物碱类等, 有保肝、 降血糖、 抗菌、 抗病毒等作用[2], 并被收录于2015年版《中华人民共和国药典》[3]。 獐牙菜属植物种类众多, 仅我国就有75个种的分布。 由于青叶胆与同属近似种十分相似, 且常以干燥全草在市场流通, 故仅从外观难以准确鉴别, 易被混淆使用。 目前, 青叶胆临床上广泛用于治疗急性肝炎, 为黄疸肝炎丸、 青叶胆片、 肝复康片等保肝药物的主要成分之一。 由于不同物种的化学组成和含量存在一定差异, 混淆用药可能导致药用疗效发生改变[4], 因此探索青叶胆及其近似种的快速有效鉴别方法有利于保证青叶胆用药的准确性和有效性。
目前, 常用植物鉴别方法包括光谱鉴别、 色谱鉴别和电化学鉴别等。 吴喆等[5]利用傅里叶变换红外光谱(Fourier transform infrared spectroscopy, FTIR)对云南重楼及4个近缘种进行偏最小二乘判别分析(partial least squares discrimination analysis, PLS-DA)、 主成分分析和系统聚类分析(hierarchical cluster analysis, HCA), 结果显示FTIR可用于重楼属植物鉴别与亲缘关系分析。 施崇精等[6]采集川牛膝、 混淆品头花杯苋和掺混川牛膝液相色谱指纹图谱, 结合相似度分析、 聚类分析和主成分分析能够区分3种川牛膝, 结果表明3种川牛膝化学成分差异较大, 不可混淆用药。 Fu等[7]通过电化学方法采集石蒜属植物花瓣指纹图谱, 能鉴别14种石蒜属植物。 可见, 单一仪器数据来源信息可有效完成中草药近缘种种类鉴别研究。 但药用植物化学组分复杂, 其药用功效常与多种化学成分有关, 单一仪器提取的信息无法全面反映整体化学信息。
近年来, 研究发现将多仪器来源指纹图谱数据进行融合并建立分类模型, 可对样品进行更全面的评价[8]。 数据融合分为低级、 中级和高级三个层次[9]。 其中, 最常用的是低级融合和中级融合, 前者直接将多源数据简单串联后建模, 后者通过对原始数据提取特征变量, 再将特征变量串联, 进而建立分类模型。 Wu[10]等将中红外数据与液相色谱数据进行低级融合与中级融合, 成功鉴别5种重楼属植物, 中级融合正确率达到100%。 Sun等[11]通过融合近红外与中红外光谱数据, 建立偏最小二乘和支持向量机判别模型, 准确对大黄真伪品进行了区分, 其数据融合分类效果更佳。 上述研究表明, 数据融合可使不同仪器信息互补, 弥补单一仪器数据信息不全的缺陷, 从不同层面反映样品间的差异, 更加全面地描述样品信息, 提高分类准确率。
迄今为止, 獐牙菜属植物种类鉴别研究以单一仪器分析为主[12, 13], 基于数据融合策略鉴别不同物种的研究未见系统报道。 本研究采集青叶胆(S. leducii)及其近似种植物共102份样品FTIR光谱与超高效液相色谱指纹图谱(ultra-performance liquid chromatography, UPLC)数据, 光谱数据预处理后通过HCA对青叶胆及其近似种之间亲缘关系进行分析, 同时, 通过FTIR、 UPLC、 低级融合与中级融合数据建立随机森林(random forest, RF)判别模型, 以期为獐牙菜属植物资源利用提供科学依据。
102份獐牙菜属植物样品信息详情见表1, 所有样品经由吉首大学李鹂教授鉴定为狭叶獐牙菜(S. angustifolia Buch. -Ham. ex D. Don.)、 西南獐牙菜(S. cincta Burk.)、 川东獐牙菜(S. davidii Franch.)、 青叶胆(S. leducii Franch.)和紫红獐牙菜(S. punicea Hemsl.)。 样品采集后洗净根茎部杂质, 分装于信封, 45 ℃恒温下烘干至恒重, 粉碎后过100目筛, 置于自封袋保存, 备用。
| 表1 獐牙菜属不同种类样品信息 Table 1 Information of Swertia samples with different species |
LC-8030超高效液相色谱仪(日本岛津公司); Frontier型傅里叶变换红外光谱仪(配备DTGS检测器和ATR附件, 美国珀金埃尔默公司); CP214型万分之一电子分析天平(上海奥豪斯仪器有限公司); Inertsil ODS-HL色谱柱(3.0× 150 mm, 3 μ m); SY-3200-T型超声仪(上海声源超声波仪器设备有限公司); DFT-50A型高速粉碎机(温岭市林大机械有限公司); 100目标准筛盘(浙江上虞市道墟五四仪器厂)。
分析纯甲醇(四川西陇化工有限公司), 色谱纯甲醇和乙腈(美国Thermo Fisher Scientific公司)。 色谱纯甲酸(美国Dikmapure公司)。 纯水由屈臣氏集团有限公司提供。
样品粉末置于ATR附件ZnSe晶体材料上(室温25 ℃), 分辨率4 cm-1, 扫描范围设为4 000~550 cm-1, 累积扫描16次, 采集红外光谱, 保存。
色谱条件: Inertsil ODS-HL色谱柱; 流动相: 0.1%甲酸(A)-乙腈(B)梯度洗脱; 流速: 0.5 mL· min-1; 进样体积: 3 μ L; 检测波长: 237和246 nm, 进样前对流动相超声10 min(功率80%), 排除气泡干扰。 梯度洗脱程序: 0~2.55 min, 8% B; 2.55~13.27 min, 8%~12.6% B; 13.27~14.00 min, 12.6%~12.9% B; 14.00~14.01 min, 12.9%~100% B; 14.01~16.99 min, 100% B; 16.99~17 min, 100%~8% B; 17~20.4 min, 8% B。
精密称取样品粉末(0.025 0± 0.000 1) g于5 mL具塞试管, 加入1.5 mL 70%甲醇, 称定重量, 保鲜膜封住试管口超声提取30 min(功率100%), 冷却至室温, 用70%甲醇补足重量, 摇匀, 过0.22 μ m微孔滤膜于进样瓶, 进行UPLC分析。
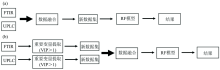
基于低级数据融合策略, 将FTIR数据与UPLC数据简单串联, 得到新的数据矩阵用于建立判别模型。 变量投影重要性(variable importance in the projection, VIP)是常用的特征变量提取方法之一, 它反映了自变量在解释因变量作用时的重要性, VIP> 1的变量被认为是重要变量[14]。 基于中级数据融合策略, FTIR和UPLC数据通过VIP> 1提取特征变量, 筛选的特征变量串联后建立模型, 具体过程见图1(a, b)。
为了消除随机抽样带来的随机性影响, 102份样品通过Kennard-Stone(KS)算法按2:1的比例划分训练集与预测集。 其中68份样品作为训练集用于建立模型, 其余34份为预测集对模型预测能力进行验证。 基于真阳性(ture positive, TP)、 假阳性(false positive, FP)、 真阴性(ture negative, TN)和假阴性(false negative, FN)4个参数, 计算灵敏性(sensitivity)、 特异性(specificity)、 精密度(precision)和正确率(accuracy), 用于评价模型性能[15]。 其中, TP为分类正确的阳性样本, FP为分类错误的阳性样本, TN为分类正确的阴性样本, FN为分类错误的阴性样本。 计算方法如式(1)— 式(4)
SIMCA 13.0软件对FTIR数据进行标准正态变量(standard normal variate, SNV)、 多元散射校正(multiplicative signal correction, MSC)、 平滑(savitzky-Golay smoothing, SG)、 一阶导数(first derivative, 1D)、 二阶导数(second derivative, 2D)等预处理。 SIMCA 13.0软件通过PLS-DA中的VIP提取特征变量; 通过R包(3.5.2版)建立RF判别模型; MATLAB R2017a软件进行KS算法划分训练集与预测集; ORIGIN 2017软件作图。
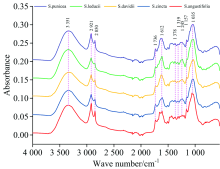
图2为青叶胆及4种近似种平均光谱图, 如图2所示, 在4 000~550 cm-1波段范围内不同獐牙菜属植物红外光谱图相似度较高, 3 351, 2 921, 2 850, 1 736, 1 612, 1 378, 1 319, 1 245, 1 157和1 035 cm-1等特征峰波数大致相同。 3 351 cm-1附近吸收峰主要归属为O— H与N— H伸缩振动; 2 921 cm-1附近吸收峰主要归属为C— H反对称伸缩振动; 2 850 cm-1附近吸收峰主要归属为C— H对称伸缩振动。 1 736 cm-1附近主要归属为酯类羰基C=O伸缩振动[16], 1 612 cm-1附近吸收峰主要归属为C=C骨架振动, 1 378 cm-1附近吸收峰主要归属为C— H面内弯曲振动, 1 319 cm-1附近吸收峰主要归属为C— H面内弯曲振动, 1 245 cm-1附近吸收峰主要归属为C— C伸缩振动和C— H面内弯曲振动, 1 157 cm-1附近吸收峰主要归属为C— C伸缩振动, 1 035 cm-1附近吸收峰主要归属为C— H面外弯曲振动, 主要与糖类物质有关。 综合分析, 青叶胆及4种近似种含有酯类、 醇类、 酮类、 萜类等物质, 其红外光谱特征峰峰形、 峰位基本一致, 但各官能团所引起的振动吸收强度差异较大, 表明青叶胆与其近似种整体化学组成相似, 但各化学成分累积量有所不同。 仅通过红外光谱的比较难以鉴别不同獐牙菜属植物, 故将借助化学计量学对样品做进一步鉴别分析。
选取指纹特征区1 800~550 cm-1波段(删减682~653 c
PLS-DA是最常用的判别分析方法之一, 通过自变量X(光谱波数)与因变量Y(类别数)建立的判别模型。 R2Y为PLS-DA模型主成分累积贡献率, Q2为交叉验证所得的一项拟合参数, R2Y与Q2的值越接近与1, 模型越可靠。 表2为青叶胆及其近似种FTIR数据经不同预处理后所建PLS-DA模型的主要参数。 由表可知, SNV+SG+2D对FTIR数据进行预处理, R2Y与Q2最大, 分别为91.2%和84.1%, 样品分类正确率达到100%。 表明SNV+SG+2D能减少干扰信息产生的影响, 有效区分重叠峰并放大其所包含的化学信息, 为最佳预处理方法。
| 表2 FTIR光谱经不同预处理后PLS-DA模型参数R2Y与Q2 Table 2 R2Y and Q2 of PLS-DA models with different pretreatment methods for FTIR spectra |
HCA是一种无监督的分析方法, 根据样品间化学信息相似程度的不同将其分为若干组。 图3为青叶胆与近似种基于FTIR数据的HCA树状图。 图中横坐标代表样品编号, 纵坐标为不同獐牙菜属植物间临界值距离, 距离越小, 样品相似度越高, 标红色样品代表被错分样品。 图中显示仅7个紫红獐牙菜(Sp)样品被错分, 其余4种獐牙菜属植物样品均分类正确, 正确率为93.1%。 聚类距离为25时, 獐牙菜属植物样品被分为两组, 狭叶獐牙菜(Sa)单独成一组, 表明狭叶獐牙菜与其他4种獐牙菜属植物样品化学成分差异最大; 距离为15时, 剩余4种獐牙菜属植物样品被分为3组, 第一组为青叶胆(Sl), 第二组包括川东獐牙菜(Sd)、 紫红獐牙菜和西南獐牙菜(Sc), 第三组仅包括一个紫红獐牙菜样品(Sp-1), 可能是由于个体变异导致Sp-1样品化学成分发生变化; 距离为10时, 仅包括紫红獐牙菜和西南獐牙菜, 表明紫红獐牙菜与西南獐牙菜化学组成相似, 其中小部分紫红獐牙菜与西南獐牙菜聚为一类, 可能是个体差异所致, 也有可能与两个物种亲缘关系较近有关。
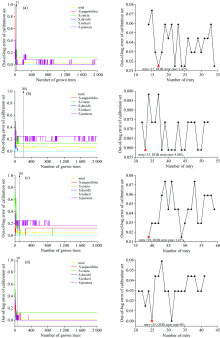
RF是一种利用多个分类树对数据进行分类或预测的分析方法, 因其使用方便、 受噪音干扰小、 能有效减少过拟合等特点, 广泛用于鉴别研究[19]。 为了获得较低误差和较高的分类性能, 在模型训练阶段, 需对RF参数ntree和mtry进行优化。 初始ntree为2000, 基于最小袋外数据(Out-of-bag, OOB)误差, 筛选最佳ntree, 此时, mtry默认为变量数的平方根。 基于最优ntree, 通过最小OOB误差, 在默认值mtry± 10的范围内, 筛选最优mtry。 将最优参数代入训练集建立最终的判别模型, 通过OOB数据验证模型预测能力。 若模型性能较差, 则需重复上述操作进一步优化参数ntree和mtry。
青叶胆及其近似种FTIR、 UPLC、 初级融合和中级融合数据集通过筛选最优ntree和mtry, 建立RF判别模型, 图4(a, b, c, d)左侧显示了OOB分类错误与ntree之间关系, 右侧显示了mtry的优化结果。 通过参数优化, FTIR、 UPLC、 初级融合和中级融合最优ntree值分别为31, 204, 101和50, mtry值分别为17, 33, 39和25, 最低OOB误差分别为1.47%, 5.88%, 1.47%和0%。 参数优化后OOB误差率由7.35%降至0%。
表3为FTIR、 UPLC、 初级融合和中级融合数据集构建RF模型的训练集与预测集参数结果。 灵敏性、 特异性、 精密度和正确率值越接近1, 则说明分类效果越好。 UPLC判别模型对獐牙菜属植物的分类效果最差, 5个样品被错分。 FTIR与初级融合分类效果一样, 仅1个样品分类错误, 表明FTIR和初级融合数据更能揭示不同种类獐牙菜样品间化学信息的差异。 FTIR模型中1个西南獐牙菜样品被错分为紫红獐牙菜, 而初级数据融合模型中1个紫红獐牙菜样品被错分为西南獐牙菜, 两个错判的原因可能是由于西南獐牙菜与紫红獐牙菜在化学组成上相似度较高, 难以区分。 这也表明紫红獐牙菜与西南獐牙菜亲缘关系较近, 与聚类分析结果一致。 与FTIR、 UPLC和初级融合相比, 中级数据融合策略能区分所有样品, 其灵敏性、 特异性和精密度均为1, 鉴别效果最佳, 说明通过筛选特征变量, 能去除一些不重要变量的干扰, 从而有效提高分类正确率。 表明青叶胆及其近似种FTIR数据与UPLC数据进行中级融合, 建立RF模型能鉴别相似度较高的样品, 分类效果最好, 为最佳策略。
| 表3 FTIR, UPLC, 低级融合与中级融合RF模型参数结果 Table 3 Parameters results of RF models for FTIR, UPLC, Low-level and Mid-level data fusion |
采集青叶胆及近似种FTIR光谱与UPLC色谱, 采用MSC, SNV, SG, 1D, 2D等方法对原始光谱进行预处理, 对最佳预处理光谱数据进行HCA分析, 探讨5种獐牙菜属植物间的亲缘关系, 并通过FTIR、 UPLC、 低级融合与中级融合数据结合RF建立物种鉴别模型。 结果显示, SNV+SG+2D为光谱最佳预处理组合; 在此基础上进行HCA分析, 表明除紫红獐牙菜Sp-1样本外, 明显聚为5类, 其中青叶胆与川东獐牙菜、 紫红獐牙菜、 西南獐牙菜亲缘关系最近, 与狭叶獐牙菜亲缘关系最远; 中级数据融合策略结合RF建立判别模型对未知样品种类的分类正确率达到100%, 效果优于FTIR、 UPLC和低级数据融合策略, 表明中级融合利用FTIR和UPLC数据信息的互补性增加了整体化学信息, 通过对数据中有效信息的提取, 提高了青叶胆及近似种分类的正确率。 中级数据融合策略建立RF判别模型能准确区分青叶胆及近似种, 为獐牙菜属植物鉴别提供了一种有效新方法, 进一步完善了獐牙菜种类鉴别体系。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|