作者简介: 姚 燕, 女, 1978年生, 中国计量大学副教授 e-mail: yaoyan@cjlu.edu.cn
厌氧发酵技术是最具发展前景的有机废弃物资源化利用技术之一, 其研发与利用在国内外都已广泛开展。 在有机废弃物厌氧发酵过程中, 通常采用生化甲烷潜力(BMP)表示物料的厌氧降解能力。 传统BMP的测定方法存在成本高、 耗时长等缺点, 因此提出了利用近红外光谱分析技术快速预测有机废弃物的生化甲烷潜力(BMP), 采用遗传算法(GA)结合支持向量机(SVM)建立函数模型, 对有机废弃物生化产甲烷潜力进行预测。 实验收集了64份水生植物和能源藻类生物质, 样品BMP原始数据通过自行搭建的产甲烷潜力实验平台获得, 同时, 利用傅里叶近红外光谱仪获取样品的近红外光谱数据。 首先, 对光谱数据进行预处理后在全谱区范围内分别建立主成分回归(PCR)、 偏最小二乘法(PLS)和递归指数偏最小二乘法(RPLS)模型, 将原始BMP数据与光谱数据建立关联, 从而实现水生植物和能源藻类BMP的快速预测。 结果表明, 在全谱区上, 递归指数偏最小二乘能够解决传统偏最小二乘法的抗粗差效果差, 易受不良数据影响等问题, 该方法可以提高模型的稳定性, 但响应速度慢、 计算效率低, 在此基础上提出遗传算法(GA)结合支持向量机(SVM)的机器学习方法, 该方法具有良好的全局搜索能力, 适用于小样本情况, 避开了从归纳到演绎的传统过程, 剔除了大量冗余样本信息, 算法简单且具有良好的鲁棒性。 结合近红外光谱频带分配可知, 利用遗传算法(GA)筛选出1 404个波长点, 大致可划分为3个代表性波段, 因此在所选取的波段利用支持向量机建立回归模型。 依据模型评价结果可知, 采用遗传算法和支持向量机所建立的预测模型不仅简化了数据规模, 同时还能提高模型预测精度, 其预测均方根误差(RMSEP)为10.32 mL, 相关决定系数( R2)为0.92, RPD为6.56, 与常规的PLS和RPLS算法建模相比, RMSEP分别减少了19.56和14.81 mL, R2分别提高了0.06和0.04, RPD分别提高了4.31和3.85。 结果表明, 采用GA-SVM算法建模预测有机废弃物生化甲烷潜力的模型准确度较高, 可以代替传统的BMP测定方法, 满足快速检测的需要。
Anaerobic fermentation technology is one of the most promising technologies for the utilization of organic waste resources. Its research and utilization have been widely carried out at home and abroad. Usually, biochemical methane potential (BMP) is used to represent the anaerobic degradation of the material in the anaerobic degradation technology of organic waste. The traditional measuring methods of BMP, are usually expensive and time-consuming. Therefore, near-infrared spectroscopy is proposed to rapid predict the biochemical methane potential (BMP) of organic waste in this paper. And genetic algorithm (GA) combined with support vector machine (SVM) is applied to establish a functional model to predict the biochemical methane potential of organic waste. 64 samples of aquatic plants and algae are collected from the south and east of China. The original BMP data of samples were obtained from the experimental scale digesbers. At the same time, near-infrared spectral data are obtained by Fourier transform near-infrared spectrometer. First of all, the prediction models were developed by the principal component regression, partial least squares, recursive exponential partial least squares (RPLS) on the pre-processed data, respectively. The aim is to connect the original BMP date with the spectral data and realize the rapid prediction of aquatic plants and algae BMP. The results show that the RPLS method on the full spectral can solve the problem of poor robustness and the poor data interference caused by the traditional PLS method. Although this method improves the robustness of the model, it has slow response speed and low computational efficiency. Therefore, we proposed a genetic algorithm (GA) combined with support vector machine (SVM) method, which is suitable for small sample cases, has good global search ability, and also avoids the traditional process from induction to deduction, and eliminates a lot of redundant sample information. In summary, the GA-SVM method is simple, and it has good stability. Combined with the band assignment of the near-infrared spectrum, it could know that the 1 404 characteristic wavelength points were selected, and roughly divided into 3 representative bands by genetic algorithm (GA), so we built the regression model by support vector machines on the selected characteristic bands. According to the results of model evaluation, it is known that the prediction model based on GA-SVM not only simplifies the date scale, but also improves the prediction accuracy. The root mean square error of prediction (RMSEP) is 10.32 mL, the coefficient of determination ( R2) is 0.92; the residual prediction deviation (RPD) is 6.56. Compared with the models PLS and RPLS, the RMSEP was decreased by 19.56 and 14.81 mL respectively; the R2 increased by 0.06 and 0.04, the RPD increased by 4.31, 3.85 respectively. The results show that the NIRS model based on GA-SVM can predict the biochemical methane potential of organic waste rapidly and has higher accuracy, it can replace the traditional BMP determination method to meet the needs of rapid detection.
厌氧发酵工艺中, 生化产甲烷潜力(biochemical methane potential, BMP)是一项重要的测试指标。 BMP是指单位有机物料在厌氧条件下发酵产生甲烷气体的能力, 通过BMP测试可以了解有机废弃物的生物降解性能和产气潜力, 从而衡量发酵效率和过程稳定性、 评估厌氧发酵工程投资收益[1]。 传统测量有机废弃物BMP的方法是在BMP测试仪器上将原料厌氧发酵一定时间, 得到发酵期间的产气量[2]。 目前得到商化仪的BMP自动测试设备有瑞典碧普公司AMPTS全自动甲烷潜力测试设备、 德国WTW公司生产的OxiTop测试设备等。 这些测试设备具有良好的准确性, 但测试周期长、 成本高、 仅适用于事前分析, 不适用于大批量实验。 近红外光谱技术预测有机废弃物BMP的方法[3], 可以实现快速、 准确的测定, 这在监控厌氧发酵状态以及指导厌氧发酵系统运行具有重要的意义。 利用近红外光谱法测定有机废弃物BMP[4]的主要思想是通过近红外光谱仪扫描样本, 将样本内部信息以光谱图的形式表现出来, 通过建立近红外光谱定量分析模型预测样本BMP。 还对光谱进行波段筛选以及算法优化, 有效提高了有机废弃物生化甲烷潜力预测模型的准确度。
实验样本选用中国东部、 南部等地收集的水生植物及能源藻类植物, 主要来源于公园、 沟渠、 海洋等地。 样品制备: 将采集到的水生植物和能源藻类样本放置于数显式101A-2工业电热恒温鼓风干燥箱, 干燥温度设置为60 ℃, 干燥时间为6 h。 通过YB-600A型粉碎机研磨成粉末状, 通过100目样本筛筛成颗粒大小均匀的粉末样本。 根据Triolo的研究[5], 干燥过程在60 ℃下研磨不会影响BMP和其他沼气生产特性。 将干燥后的样本迅速放入样本袋中进行标号, 放入干燥皿密封避光保存。 实验共制备64个样本, 随机选取其中的54个样本作为校正集, 10个样本作为预测集。
自行搭建实验平台, 该平台及简图如图1所示。 实验中所用的接种物来自杭州市七格污水处理厂, 底物为64种已制备的粉末样本。 将接种物和底物按5:1的比率加至500 mL发酵瓶, 在中温条件(37 ℃)条件下进行发酵, 不再产气时视为发酵终止。 实验每批为期30 d, 每隔两天记录一次排水量, 发酵总历时4个月。 实验设置实验组和空白对照组(无底物)。
利用美国Thermo Fisher Scientific公司生产的Nicolet系列NEXUS670型号的傅里叶变换近红外光谱仪扫描样本得到近红外光谱, 用样品勺取出少量粉末状样本使其均匀的覆盖在光谱仪操作台的石英片上, 光谱仪的扫描方式为漫反射, 测量范围806~2 500 nm, 分辨率设为16 cm-1, 扫描次数32次。 每个样本采集光谱5次, 取5次平均值作为最终实验数据, 以减少随机误差造成的影响。 实验全程在室温下进行, 环境湿度为50%。
1.4.1 遗传算法
遗传算法(GA)模拟了遗传选择和自然淘汰的生物进化过程计算模型, 是一种具有“生存+检测”的迭代过程的搜索算法, 可用于建立校正模型前的波长优选[6], 减少建模波长数据, 提高预测精度, 主要包括编码、 初始群体生成、 适应性函数设定、 复制、 交叉、 变异等六个主要步骤[7]。
1.4.2 支持向量回归
支持向量机是建立在统计学习理论的VC维理论和最小化结构风险基础上的一种数据挖掘方法[8], 它根据有限的样本信息在模型的复杂性和学习能力之间找到一个最佳平衡点, 以获得模型最好的推广能力和适应能力[9]。 研究中利用遗传算法选取的特征波长作为输入向量, 建立支持向量回归预测模型。
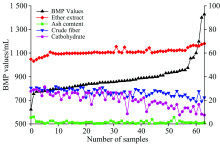
64个样品产气量如图2所示。 从图2中可以看出, 64个水生植物和能源藻类样品在30天产气周期里, 产气量范围为615~1 428 mL, 多数样本产气量在800 mL上下浮动。 其中以64号样本红藻和63号样本马尾藻产气量最多, 1号样本羊栖菜产气量最少。 根据Bryant提出的厌氧降解过程的四阶段原理, 碳水化合物经过4个阶段的化学反应, 产出甲烷和二氧化碳, 样本的生化产甲烷潜力与碳水化合物的含量成正比。 实验测得的样本碳水化合物含量与BMP产气量关系如图3所示, BMP产气量大体上随碳水化合物含量减少而减少, 本研究所得到的实验数据基本与理论重合。 实验中存在个别样本如8号、 32号、 35号、 44号等碳水化合物含量相对较高, BMP相对较低的情况, 经验证得知, 该情况的出现可能与这几种样本内在的特殊成分有关。
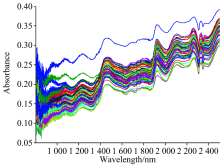
图4为64个废弃物原始近红外光谱图。 图中显示, 样本的吸收峰出现的范围900~1 500和1 800~2 300 nm, 其吸光度随波长的增加而增加。 1 000, 1 200和1 500 nm附近的C—H, N—H和O—H的倍频吸收带以及2 000和2 100 nm附近的N—H和O—H倍频吸收带均可见, 这些吸收峰反映了样本中C—H, N—H和O—H等含氢基团的信息, 样本的主要成分如蛋白质、 碳水化合物等均含有这些含氢基团, 选择的样本具有代表性。
由于本实验直接采集样本光谱, 存在信号噪声、 信号干扰等问题, 因此需要对样本原始近红外光谱进行预处理, 减弱或消除各种非目标因素对光谱信号的影响。 有研究表明, 在建立定量分析模型前, 通过正交的方法, 可将与样本无关的信息剔除, 提高模型预测精度; 同时还可以减少建模所需要的主因子数, 进一步简化模型[10]。 在众多消噪的方法中, 选择利用正交信号校正(orthogonal signal correction, OSC)进行滤除干扰信号。 实验选择非线性迭代偏最小二乘(NIPALS)、 类主成分分析(类PCA)和直接正交信号校正(DOSC)三种常用的正交信号校正算法分析, 对校正后的光谱建立PLS模型, 其结果如表1所示。
| 表1 光谱预处理校正后的模型预测结果 Table 1 Model prediction results after spectral preprocessing correction |
在表1中可以看出, 与未经预处理的模型预测结果相比, 经过预处理后的模型预测结果显著提高, 说明预处理能够有效提高模型预测效果和稳定性。 在正交信号校正的三种算法中, NIPALS算法最佳, 与不经过消噪处理的结果相比, 预测均方根误差RMSEP减少了16.33 mL, 相关系数提高了0.15。
为了简化模型数据, 降低近红外光谱区域内的冗余信息, 提高模型预测精度, 采用特征波长选取方法GA-SVM波长筛选算法选取近红外原始光谱特征波段, 与全波段范围内建立的PCR、 PLS及RPLS模型进行比较, 通过比较交互验证均方根差(RMSECV)、 预测均方根误差(RMSEP)、 相关系数(R2)、 相对分析误差(RPD)等模型评价参数来探讨GA-SVM方法的性质特点。
按照遗传算法波长筛选步骤, 将原始光谱谱区806~2 500 nm包含的2 179个光谱数据分为30个子区间, 即染色体长度为30。 遗传算法的各参数设定如下: 种群大小为54个, 最大繁殖代数为200, 交叉概率为0.85, 变异概率为0.05, 适应度函数为f=RMSECV。
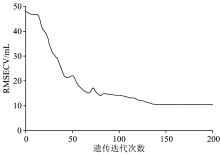
当前RMSECV最小值随遗传代数变化趋势如图5所示, 当遗传代数达到140后, RMSECV基本不再减小, 曲线趋于平坦, 这时已经搜索到最优解。 由此挑选出了1 404个波长点以及三个特征波段, 如表2所示, 与原始2 179个波长点相比简化了数据规模。
| 表2 遗传算法筛选波段及波长点 Table 2 The characteristic bands and characteristic wavelength points selected by GA |
从表2中可以看出, 遗传算法挑选出了1 404个波长点, 约占原波长点数的64%, 与原始原始2 179个波长点相比大大简化了数据规模。 结合近红外光谱频带分配[11]的分析, 利用GA-SVM挑选的806~1 362.5 nm波段包含了C—H基团的信息, 在水生植物和能源藻类样本中, 6种主要有机物均含有大量的C—H基团; 1 415.4~1 751.6 nm波段主要包含了O—H基团和C—H基团的信息, 这些基团主要存在于样本的纤维素、 半纤维素和木质素中; 1 896.2~2 500 nm波段主要包含的样本信息较为复杂, 包括O—H基团、 C—H基团、 亚甲基、 C—C单键、 C=O双键和C=C双键。 这些特征波段包含了样本的大量信息, 可见, 选取的波长点和波段具有代表性。
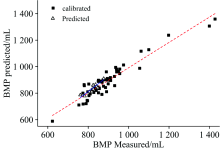
在选取的特征波段上建立支持向量机回归模型, 利用遗传算法选取的1 404个波长点作为SVM建模的输入量, 采用RBF核函数, 选择惩罚系数C=1 000, 核函数的宽度参量γ =0.5的条件下, GA-SVM模型的预测结果如图6所示。 将该GA-SVM建模实验结果与原始波长下的PCR, PLS和RPLS三种建模方法进行比较, 如表3所示。
| 表3 PCR, PLS, RPLS和GA-SVM预测结果比较分析 Table 3 The prediction results of PCR, PLS, RPLS and GA-SVM |
结合图6和表3分析发现, 在全波谱范围内, PCR和PLS模型的预测精度较低, 且这两种预测模型的RPD均小于2.5, 说明模型的预测效果较差, 难以进行定量分析。
RPLS的模型预测的准确性有所提高, R2为0.88, RPD为2.71, 但是提高精度有限, 仍需进一步改善。
相比在全谱区范围建模, 运用GA-SVM选择特征波长建模后, 模型的预测精度得到很大提高, RMSEP从RPLS的43.04 mL下降到16.61 mL, R2由RPLS的0.88提高到0.93, 同时模型的波长点数由2 179下降到1 404, 模型数据得到简化。 虽然GA-SVM预测模型的复杂程度有所加深, 但是模型的RPD值达到6.56, 表明GA-SVM模型预测精度有明显提高, 模型预测效果良好, 可用于实际检测。
综合评价四种方法所建模型的预测能力, 在提取的特征波段上建立预测模型可以有效地提高模型预测精度, GA-SVM所建模型各评价指标均优于RPLS, 可见遗传算法对于提取水生植物和能源藻类有效的BMP近红外光谱信息具有良好的效果, 利用支持向量机建模大幅提高了预测精度和模型稳定性, 预测效果良好。
通过遗传算法(GA)和支持向量机(SVM)对水生植物和能源藻类生物质的近红外光谱进行特征谱区和特征波长的选取。 结果发现, 与全谱区建立模型相比, GA-SVM算法能够提取具有代表性的特征波段从而简化了模型数据, 较全谱区建模具有较高的BMP模型预测精度, 两者的结合有效提高了运算效率和模型精度, 最终建立的水生植物和能源藻类生物质的近红外光谱模型预测生化产甲烷潜力(BMP)的能力和精度更高。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|