作者简介: 王彩霞, 1994年生, 宁夏大学农学院硕士研究生 e-mail: 18295671639@163.com
为探究高光谱成像(400~1 000 nm)对羊肉中饱和脂肪酸(SFA)含量检测的可行性, 提出一种基于特征光谱信息和图像纹理特征融合的SFA含量预测模型, 实现对羊肉中SFA含量的快速检测及分布可视化。 利用分段阈值法构建掩膜图像, 获取羊肉样本感兴趣区域(ROI), 结合SPXY法对样本集进行划分并对相关光谱信息进行预处理, 分别采用连续投影算法(SPA)、 变量组合集群分析法(VCPA)和 β权重系数法提取特征光谱; 通过获取羊肉样本主成分图像, 结合灰度共生矩阵(GLCM)算法提取图像纹理信息; 分别对特征光谱、 图像信息及图谱融合信息建立的偏最小二乘回归(PLSR)与最小二乘支持向量机(LS-SVM)预测模型进行对比分析。 利用5种不同对原始光谱数据进行预处理, 经SNV法预处理后的光谱其校正集与预测集相关系数分别为0.921和0.875, 较原始光谱分别增加了0.001和0.04, 均方根误差模型分别为0.244和0.268, 较原始光谱模型分别减少了0.003和0.06; 对SNV法预处理后的光谱数据进行特征波长提取, SPA法、 VCPA法及 β权重系数法分别提取出12, 10和9个特征波长; 获取羊肉样本的前5个主成分图像, 选择所含信息量最多的第一主成分图像进行纹理特征提取, 依次提取0, 45°, 90°和135°方向下的能量、 熵、 同质性和相关性共4个主要纹理特征。 利用SPA法提取的特征波长建立的PLSR与LS-SVM模型性能较好, PLSR模型校正集与预测集相关系数分别为0.884 9和0.880 7, 均方根误差分别为0.300 1和0.260 6; LS-SVM模型校正集与预测集相关系数分别为0.898 7和0.892 6, 均方根误差分别为0.276 7和0.247 6; 图谱信息融合模型中, PLSR模型校正集与预测集相关系数分别为0.907 1和0.907 8, 较特征光谱模型分别增加了0.02和0.03, 均方根误差分别为0.326 9和0.299 2, 较特征光谱模型分别增加了0.03和0.04; LS-SVM模型校正集与预测集相关系数分别为0.920 6和0.894 6, 较特征光谱模型分别增加了0.02和0.002, 均方根误差分别为0.251 9和0.245 8, 较特征光谱模型分别减少了0.02和0.002。 光谱预处理中经SNV法处理后的光谱所建模型性能优于其他预处理方法; 采用SPA法提取的12个特征波长简化了光谱模型, 提高了模型性能, 特征光谱建模的最优方法为SPA-LS-SVM; 图谱信息融合模型较特征光谱模型, 模型相关系数增加较少, 表明图像纹理信息虽携带了部分有效信息, 但这些信息与羊肉中SFA含量之间的相关性有待进一步研究。 基于图谱信息融合模型的预测性能最优, 其次为光谱信息模型。 择优选取SPA-PLSR模型计算羊肉样本中每个像素点的SFA含量, 利用伪彩色图直观表示了羊肉样本中SFA的含量分布。 实现对羊肉样本SFA含量的无损检测及分布可视化表达。
In order to explore the feasibility of detection of saturated fatty acids (SFA) in muttons by using hyperspectral imaging techniques (400-1000 nm), this paper proposed a prediction model based on the fusion of characteristic spectral information and image texture features, realizing the rapid detection and distribution visualization of SFA content in mutton. Firstly, the binary mask image was successfully determined by the segmentation of a certain threshold, and Region of Interest (ROI) in the sample of mutton was determined by binary mask image. SPXY methods were used for dividing the sample set, preprocessing of correlation spectral information. And continuous projection algorithm SPA,VCPA and β weight were used to select wavelength of the spectrum. The image textural information was described by taking the principal component image and the gray level co-occurrence matrix (GLCM) algorithm of the mutton samples. The partial least squares regression (PLSR) and the least squares support vector machine (LS-SVM) prediction model built based on the characteristic wavelength, textural information, textural combined with characteristic wavelength were compared and analyzed, respectively. Preprocessing of original spectral data using five methods without pretreatment. The correlation coefficients of calibration set and prediction set were 0.921 and 0.875, respectively. Compared with the original spectrum, the correlation coefficients of calibration set and prediction set were increased by 0.001 and 0.04, and the root mean square errors were 0.244 and 0.268, respectively. Compared with the original spectrum, the correlation coefficients of calibration set and prediction set were reduced by 0.003 and 0.06 respectively. This paper extracted characteristic wavelengths of the spectral from the pre-processed date using SNV, SPA, VCPA and β coefficient methods extracted 12, 10 and 9 characteristic wavelengths, respectively. Five principal component images were selected based on PCA, and four textural feature variables (energy, entropy, homogeneity and correlation) were extracted by the first principal component image, with which the most information in the 0, 45°, 90°, and 135° directions, respectively. The performance of PLSR and LS-SVM models based on characteristic wavelengths extracted by SPA method was better. The correlation coefficients of PLSR model correction set and prediction set were 0.8849 and 0.8807, and the root mean square errors were 0.300 1 and 0.260 6, respectively. The correlation coefficients of LS-SVM model correction set and prediction set were 0.898 7 and 0.892 6, and the root mean square errors were 0.276 7 and 0.247 6, respectively. In the atlas information fusion model, the correlation coefficients of correction set and prediction set of PLSR model were 0.907 1 and 0.907 8 respectively, which were 0.02 and 0.03 higher than that of characteristic spectral model, and the root mean square errors were 0.3269 and 0.2992, respectively, which were 0.03 and 0.04 higher than that of characteristic spectral model; The correlation coefficients of LS-SVM model calibration set and prediction set were 0.920 6 and 0.894 6, respectively, which were 0.02 and 0.002 higher than that of characteristic spectral model, and the root mean square errors were 0.251 9 and 0.245 8, respectively, which were 0.02 and 0.002 less than that of characteristic spectral model. Compared with other pretreatment methods, the performance of the model constructed by the SNV was better than others; The 12 characteristic wavelengths were extracted by SPA method to simplify the spectral dimension and improve the performance of the model. The optimal method of characteristic spectral modeling was SPA-LS-SVM. Compared with the characteristic spectral model, the correlation coefficient of the model increased less, which indicated that the image texture information carried less effective information, and the correlation between these information and saturated fatty acid content in Mutton needed to be further studied. The prediction performance based on the textural combined with characteristic wavelength information fusion model was the best, and the texture information model was the worst. Thus, the SFA content of could be calculated by SPA-PLSR model, and the visual distribution map of SFA content in mutton samples was plotted by using pseudo-color drawing.
脂肪酸是影响羊肉品质的重要因素之一[1]。 羊肉中的脂肪酸主要为饱和脂肪酸(SFA), 其含量与人体血液中的胆固醇水平有密切关系[2]。 传统的脂肪酸检测方法主要为气相色谱法, 虽检测精度较高, 但操作复杂, 耗时耗力[3]。 因此, 对羊肉中脂肪酸含量进行快速无损检测具有重要意义。
高光谱成像技术具有快速无损、 图谱合一的优点, 可以同时实现光谱分析和图像处理。 国内外学者利用高光谱图谱融合技术对果蔬类产品品质研究较多[4, 5], 但在肉品领域涉及较少。 赵娟等[6]基于主成分纹理特征建立的牛肉嫩度线性判别模型预测集判别精度达94.44%; 邢素霞等[7]利用高光谱成像图谱合一的特点, 通过训练后的K-means-RBF神经网络对20个鸡肉测试集样品进行分类, 正确率达100%。 关于羊肉中SFA含量的研究尚未见报道。 因此, 我们利用高光谱图谱融合技术对羊肉中SFA含量进行预测, 提出一种基于分段阈值法的感兴趣区域(ROI)提取方法; 通过不同的光谱预处理及特征波长提取方法建立不同的预测模型, 优选出最佳光谱信息建模方法。 同时, 探讨了光谱信息和纹理信息对羊肉中SFA含量预测性能的影响, 通过绘制SFA含量分布的伪彩色图, 实现对羊肉中SFA含量的无损检测及分布可视化表达。
羊肉样本采自宁夏盐池县鑫海清真食品有限公司及甘肃临夏农贸市场。 经屠宰后置于4 ℃下冷藏24 h进行排酸处理。 排酸结束后, 取羊胴体背最长肌、 前腿肉及后腿肉进行分割, 剔除表面多余的油脂和筋膜, 放入保温箱运至实验室, 贮藏在4 ℃冷柜备用。 光谱扫描前将肉样整形切块(大小约为40 mm× 20 mm× 10 mm), 共获得羊肉样本141个(其中, 背最长肌、 前腿肉及后腿肉样本数分别为53, 41和47个)。
高光谱成像仪为美国Headwall Photonics公司生产的HyperSpec Vis-NIR。 该仪器包括: 成像光谱仪(Imspector N系列, Golden Way Scientific Co., Ltd., US)、 G4-232增强型EMCCD相机(Golden Way Scientific Co., Ltd., US)、 光源系统由2个线光源(90~254 VAC, 47~63 Hz, Golden Way Scientific Co., Lab., EQUIP)、 VT-80精密电控位移平台、 计算机和数据采集软件(Hyperspec-N for AndorLuca Rev A.3.1.4.vi, Headwall Photonics Instruments Co., Ltd., Beijing, China)等组成。 成像光谱仪共有125个波段, 光谱分辨率为2.8 nm, 狭缝宽度25 μ m, 相机像素尺寸8.0 μ m。
光谱扫描前, 高光谱仪器预热30 min。 同时, 将肉样放置于室温, 待肉样中心温度达到室温水平后, 用滤纸吸干样品表面的水分, 进行光谱扫描。 光谱扫描时光源强度、 传感器中暗电流的存在、 曝光时间以及肉样表面纹理形状、 色泽等都会影响光谱信息采集。 因此, 图像采集前需进行黑白校正并设置合理的扫描参数。 经预试验最终确定采集参数为: 物距为360 mm, 扫描速度设置为200 μ m· s-1, 成像光谱仪曝光时间为30 ms, 线扫描实际长度为60 mm, 最终可获得成像光谱分辨率为320 pix× 360 pix× 125 pix立方光谱图像。
羊肉中SFA的测定根据Floch[8]的方法略作修改。 具体方法见文献[9]。
GC条件: HP-5MS色谱柱 (30 m× 0.25 mm, 0.25 μ m), 进样口温度250 ℃, 检测器温度200 ℃, 升温程序: 70 ℃保留1 min, 以5 ℃· min-1升温到210 ℃, 保留96 min。 利用计算机NIST 0.5谱库数据库检索, 对比MS图库中的标准谱图来确认羊肉中的脂肪酸成分, 通过内标十一烷酸定量。
1.4.1 感兴趣区域(ROI)选取
由于羊肉表面不平整, 在采集高光谱图像时肉样与背景交界处会产生阴影区域。 同时, 肉样表面水分分布、 光源照射均一度以及少量筋膜的存在等都会对光谱信息的采集造成影响。 为消除这些影响, 利用ENVI软件中阈值分割法进行ROI选取。 首先对原始光谱图像进行掩膜处理, 得到掩模图像, 再将掩模图像应用于整个高光谱图像上, 得到去除背景干扰的高光谱图像。 然后对亮斑、 筋膜及肌肉部分分别设置阈值进行分割。
1.4.2 光谱特征信息提取
原始光谱数据维度较高, 信息冗余。 为提高预测模型性能, 需对光谱数据进行特征波段提取。 连续投影算法(SPA)[10] 是一种矢量空间共线性最小的前向变量选择算法, 通过从自变量中选取最低限度冗余信息的变量组, 可得到共线性小、 冗余度低的特征波长, 提高了建模效率和模型准确性。 变量组合集群分析法(VCPA)[11]作为一种较新的特征变量筛选方法, 通过二进制矩阵采样法(BMS)进行模型种群分析, 并利用指数衰减函数(EDF)迭代进行变量筛选, 将被选频率较大的剩余变量进行组合作为最终选择出的特征波长变量。 加权β 权重系数是根据建立的PLSR模型得到回归系数曲线图, 基于局部绝对值最大的原则挑选特征波长的方法。
1.4.3 图像纹理信息提取
采用灰度共生矩阵法(GLCM)[12] 提取图像纹理信息。 对高光谱图像进行主成分分析得到前5个主成分图像, 选取表达信息量最多的图像进行纹理信息提取。 利用Matlab中的graycomatrix函数, 设置距离参数值为1, 依次取0, 45° , 90° 和135° 方向下的能量、 熵、 同质性和相关性[13]来描述图像纹理信息。 由于所得信息维度较大, 故以四个纹理信息的平均值和标准差代表最终的纹理信息。 每个样本可以获取8个参数值来表示该样本的纹理信息。
1.4.4 建模方法
比较了偏最小二乘回归模型(PLSR)与最小二乘支持向量机模型(LS-SVM)对羊肉样本SFA含量的预测效果。 PLSR作为一种多变量回归分析方法, 可对光谱数据进行降维, 对数据信息进行综合筛选, 并对两组变量间的相关性进行分析等, 具有较高的建模稳定性。 LS-SVM是在SVM基础上改进的用于解决模式分类和函数估计问题的支持向量机, 它采用最小二乘线性系统作为损失函数, 有较强的非线性处理能力, 有效地简化了计算的复杂性[14]。
模型性能由相关系数(r)和均方根误差(RMSE)综合评价, 性能较好的模型具有较高的r值和较低的RMSEC/RMSEP。 模型的r越大, 稳定性越好; RMSE越小, 模型预测能力越好。
数据处理及分析基于ENVI 4.8(ITT Visual Informagtion Solutions, Boudler, USA); Matlab 2016a (The MathWorks Inc. Massachusetts, USA); The Unscramber X 10.1(CAMO AS, Oslo, Norway)等软件实现。
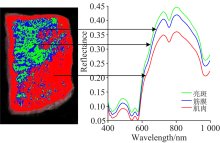
采用分段阈值法进行ROI选择。 筋膜区域阈值设为0.37~0.43, 高于0.43的样本部分定义为亮斑区域, 低于0.37但高于0.1的样本部分为肌肉。 由图1可知, 在670~830 nm波段, 亮斑、 筋膜及肌肉部分的光谱反射率相差较大; 亮斑和筋膜部分的反射率明显高于肌肉部分。 因此, 只选择肌肉部分用于确定ROI。
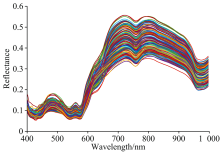
采集的高光谱原始光谱如图2所示。
利用SPXY法对141个羊肉样本按接近3:1的比例划分为校正集105个与预测集36个。 共测得37种脂肪酸。 其中SFA共有17种。 羊肉样本SFA含量化学检测及统计结果如表1所示。
| 表1 羊肉样本饱和脂肪酸含量统计结果 Table 1 Statistics results of saturated fatty acid content in mutton samples |
对划分后羊肉样本的光谱数据进行预处理, 采用卷积平滑(SG)、 区域归一化(Area normalize)、 基线校正(Baseline)、 标准正态变量变换(SNV)及多元散射校正(MSC)等方法对原始光谱进行预处理, 结合PLSR模型建模, 获取校正集、 预测集的相关系数与其均方根误差。 表2表示不同预处理方法建立的PLSR模型比较结果。
| 表2 不同预处理方法的PLSR建模结果 Table 2 The results of PLSR models by different pretreatment methods |
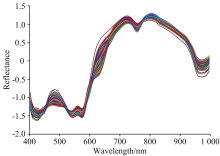
由表2可知, PLSR模型校正集与预测集相关系数差异不显著, 说明校正集样本SFA含量选择范围较好的涵盖了预测集样本。 与原始光谱建模效果比较, 经过预处理后模型的校正集相关系数略有下降, 尤其经Baseline法预处理后的光谱, 其校正集相关系数仅为0.911。 分析其原因, 基线校正法在使测量结果处于同一标准的过程中删除了部分有效信息。 经SNV法预处理后的光谱所建模型的校正集与预测集相关系数均高于原始光谱, 均方根误差小于原始光谱, 表明SNV消除了羊肉样本表面不平整、 表面散射及光程差异造成的影响, 提高模型的稳健性及预测准确性, 因此采用SNV法对光谱数据进行预处理, 处理后的光谱曲线如图3所示。
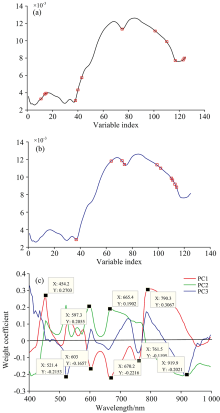
采用SPA, VCPA和β 权重系数法进行光谱数据特征波长提取。 其中, SPA法中设置特征波长选择范围为5~30。 VCPA法中采用5折交叉验证, 采样次数设置为1 000, 迭代次数为50, 变量子集中优秀子集所占的比率0.1。 β 权重系数是反映评价指标重要程度的量化系数, 系数越大, 说明该指标对总目标影响越大。 在PLSR模型建立过程中, 得到前三个主成分累计贡献率为99%, 故选前三个主成分进行特征波长提取。 三种方法提取的信息如图4(a)— (c)所示。
由图4可知, 在400~1 000 nm波段总共有125个波段, 但与SPA含量特征相关的特征波段为数甚少。 从4(a)可以看出, SPA法提取的特征波长在各波段分布较为均匀, 表明通过SPA方法确定特征波长与SFA含量相关性较大; 图4(b)中筛选出的特征波长较分散, 主要集中在880~950 nm的短波近红外波段, 此波段与SFA含量相关的信息较多; 由图4(c)可知, 前3个主成分的波峰波谷位置较为明显, 在603~665及670~760 nm波段内权重系数也出现少量波峰波谷, 但权重值较小, 与原始光谱相比较, 所含信息量不足以确定为特征波长。 特征波长提取结果见表3。
| 表3 特征波长提取结果 Table 3 The results of extracting the characteristic wavelengths |
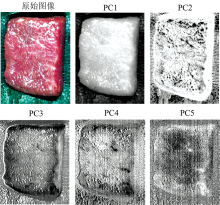
对羊肉样本图像进行主成分分析。 提取前5个主成分(累计贡献率为99%), 主成分图如图5所示。 选择表达信息量最多的PC1进行纹理特征提取。 采用GLCM法依次取0, 45° , 90° 和135° 方向下的能量、 熵、 同质性和相关性进行图像纹理信息提取。 结果以四个纹理信息的平均值和标准差来表示, 数据保存在141× 8的矩阵中。
2.4.1 特征光谱建模分析
对表3提取出的特征变量分别运用PLSR和LS-SVM进行建模分析。 结果见表4。
| 表4 不同模型和特征波长的预测结果 Table 4 Prediction results of different models and characteristic wavelengths |
由表4可知, 除β 权重系数法提取的特征波长所建的PLSR模型效果较差外, 其他模型的校正集与预测集相关系数均达到0.85。 β 权重系数法提取特征波长相对较少, 但有效信息损失较大, 不利于提高模型预测效果。 PLSR模型中SPA波长提取方法所建模型的准确率略优于VPCA。 LS-SVM模型中SPA法校正集的r略低, 但预测集r与RMSEP较VCPA更优。 所以, 选择SPA为最佳特征波长提取方法, 并且非线性的LS-SVM模型效果明显优于线性PLSR模型。 因此, SPA-LS-SVM法建模效果最佳。
2.4.2 图谱特征融合建模分析
根据2.4.1的建模结果, 融合建模方法基于SPA法提取的特征波长进行。 将光谱特征与纹理特征融合优化, 建立基于光谱信息与纹理特征融合的预测模型。 并对建模结果进行比较。 结果如表5所示。
| 表5 特征波长与纹理信息融合模型的预测结果 Table 5 Prediction results of characteristic wavelengths and texture features fusion models |
由表5可知, PLSR模型校正集与预测集相关系数分别为0.907 1和0.907 8, 较特征光谱模型分别增加了0.02和0.03, 均方根误差分别为0.326 9和0.299 2, 较特征光谱模型分别增加0.03和0.04; LS-SVM模型校正集与预测集相关系数分别为0.920 6和0.894 6, 较特征光谱模型分别增加了0.02和0.002, 均方根误差分别为0.251 9和0.245 8, 较特征光谱模型分别减少了0.02和0.002。 光谱信息预测结果与图谱特征融合预测结果相比, 融合建模结果略优, 但总体而言差异不显著。 说明在信息融合的过程中, 纹理特征虽携带了部分有效信息, 但这些信息与羊肉样本中SFA含量的相关性较小。
为了直观表述高光谱模型预测效果, 对羊肉中SFA含量进行可视化分析。 根据2.4中的建模结果分析可知, 虽LS-SVM建模效果优于PLSR法, 但LS-SVM模型结构较复杂, 花费时间较长。 因此样本中SFA含量分布可视化的研究采用SPA法提取出的特征波长所建立的PLSR预测模型进行。 模型方程如下所示
Y=-3.01+2.05X449.4+4.08X463.8+9.90X468.6+13.49X579.0-16.92X588.6+14.86X603.0+13.41X756.7+37.39X881.5-35.22X924.7+23.05X958.3-22.42X987.1+16.88X991.9
将图像中像素点的数据代入到预测模型中, 得到每个像素点的赋值, 然后根据赋值大小对像素点进行显色处理, 显色原则用Jet色度带所示。 主要操作过程为: 采用ENVI软件对高光谱图像进行掩膜处理去除背景, 得到样本掩膜后的二值化图像, 后把此图像应用至125个波段的高光谱图像获取掩膜后图像, 将图像信息用三维数据矩阵表征, 然后利用PLSR模型获取与SFA含量相关的回归系数, 使特征图像每个像素点分别与代表SFA特征的回归系数相乘, 根据矩阵赋值大小对像素点进行显色处理。
可视化处理过程及结果如图6所示。 红色区域表示SFA含量较高, 蓝色为背景色设定参数为0, 颜色随SFA含量降低从红色向蓝色渐变, 样本不同区域颜色差异及深浅表示该像素对应的SFA含量。
由图6可知, 样本边缘部分偏蓝色, 肌间脂肪较多的部分颜色偏红。 分析其原因, 羊肉中的SFA在脂肪中分布较多。 样本边缘部分较为平滑, 大理石花纹不显著, 所以肌间脂肪含量较少颜色偏蓝, 样品中间部分大理石花纹明显, 肌间脂肪含量也较多故颜色偏红。 因此SFA在肌间脂肪部分含量较高。 通过可视化图能对羊肉样本中SFA的含量分布更直观了解。
基于高光谱成像技术对羊肉中SFA含量进行检测分析。 主要结论有:
(1)通过5种不同预处理方法对羊肉样本光谱信息进行预处理, 优选出SNV法为最佳方法。 基于特征波长建立的PLSR与LS-SVM模型预测效果均较好。 其中, SPA法提取的12个特征波长建模结果优于其他两种方法。 LS-SVM模型建模效果均优于PLSR法, 光谱数据的最优建模方法为SPA-LS-LSVM。
(2)基于图谱信息融合建模时, 图谱融合模型较特征光谱模型性能略好, 但优势并不显著。 图谱信息融合建模需各信息均为有效信息, 通过GLCM提取的纹理特征虽携带了部分有效信息, 但这些信息与羊肉样本中SFA含量的相关性较小。 因此, 寻找图像纹理信息与羊肉SFA含量之间的相关性是提高模型性能的重要途径。
(3)通过羊肉样本SFA含量分布可视化图, 可知SFA主要分布在肌间脂肪含量较多的部分, 肉样边缘含量较低。 在后续工作中, 将对其他脂肪酸含量及分布进行进一步研究。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|