作者简介: 沈东旭, 1995年生, 重庆大学大数据与软件学院硕士研究生 e-mail: Dongxu1995@outlook.com
血液鉴别对于检验检疫、 刑侦以及动物保护领域具有非常重要的意义, 传统的的血液鉴别方法在鉴别的过程中存在分析周期长、 对血液样本造成损害等缺点。 而拉曼光谱可以通过分析与入射光频率不同的散射光谱得到分子振动、 转动方面的信息, 进而得到物质的组成成分, 并且具有零污染非接触的特点, 为血液的无损鉴别提供了可能, 但是在拉曼光谱中, 各个波长点之间存在严重的多重共线性, 直接使用全光谱进行建模会增加模型的复杂性和降低模型的稳定性。 针对拉曼光谱的特点, 提出了一种基于神经网络的波长选择方法。 该方法利用神经网络学习到各个波长点对校正模型的贡献权重, 并将权重的均值作为阈值, 去除权重低于阈值的波长点, 以达到波长选择的目的。 为了更容易确定筛选的阈值, 在权重学习的过程中加入了稀疏约束, 极大的减少了用于筛选的波长点。 利用动物与人血清的拉曼光谱数据集对所提方法进行了验证, 实验结果表明, 利用该方法得到的光谱建立的校正模型, 相比于全光谱数据在分类准确率和AUC值都有一定的提升, 人工神经网络(NN)的准确率达到了94.495%, AUC值达到了0.9850, 偏最小二乘(PLS-DA)的准确率达到了92.661%, AUC值达到了0.9760。 与传统的波长选择方法UVE相比, 该方法选择的波长点更少, 仅选择了42个波长点用于建模, 而且得到的校正模型的分类准确率和AUC值更高, 证明该波长选择方法能有效的筛选出对建模有贡献的波长点, 提高了模型的分类准确率和稳定性, 为血液的无损鉴别提供了可能, 具有一定的实用价值。
Blood identification is crucial for the field of inspection and quarantine, criminal investigation and animal protection. Traditional blood identification methods have shortcomings such as long analysis period and damaging to blood samples during the identification process. Raman spectroscopy can obtain molecular vibration and rotation information by analyzing the scattering spectrum different from the incident light frequency, and obtain the composition of the material. Moreover, it has the characteristics of zero pollution and non-contact, which provides the possibility of non-destructive identification of blood. However, there is serious multicollinearity between each wavelength point in Raman spectrum, and directing the use of full-spectrum for modeling will increase the complexity of the model and reduce the stability of the model. According to the characteristics of Raman spectroscopy, this paper proposes a wavelength selection based on neural network. The method uses the neural network to learn the contribution weight of each wavelength point to the correction model, and uses the mean value of the weight as the threshold value to remove the wavelength point whose weight is lower than the threshold value, so as to achieve the purpose of wavelength selection. In order to make it easier to determine the threshold of the screening, sparse constraints are added to the weight learning process, which greatly reduces the wavelength points used for screening. The proposed method was validated by Raman spectroscopy datasets of animal and human serum. The experimental results show that the model established by the wavelength selection using this method has a certain improvement in classification accuracy compared and AUC value with the full spectrum, the accuracy of artificial neural network (NN) reached 94.495% and AUC value reached 0.985 0. The accuracy of PLS-DA reached 92.661%, and AUC value reached 0.976 0. Compared with the traditional wavelength selection method UVE, the method selects fewer wavelength points, and only 42 wavelength points have been selected for modeling, the classification accuracy of the calibration model and AUC value is high, and accuracy reached 92.661%, and AUC value reached 0.976 0. It is proved that the wavelength selection method can effectively screen out the wavelength points contributing to the modeling, which provide a possibility for non-destructive identification of blood, which has certain practical value.
血液中包含了大量的遗传信息和基因信息, 在出入境的检验检疫、 刑侦以及动物保护等领域常常需要对未知的血液样本进行物种鉴别, 所以对于血液鉴别的研究有非常重要的意义。 传统的血液鉴别方法包括沉淀反应、 凝聚反应、 酶免疫分析、 等电聚焦、 高效液相色谱仪等[1]。 这些技术都存在分析周期长、 对血液样品会造成损害等缺点, 但是许多领域要求在检测过程中尽可能的保证样品的完整, 尤其是刑事侦测方面, 需要利用有限的样品做进一步的分析。
拉曼光谱分析法是通过对与入射光频率不同的散射光谱进行分析以得到分子振动、 转动方面的信息, 进而得到物质的组成成分, 它具有零污染、 非接触等特点, 为血液的无损鉴别提供了可能。 到目前为止, 已经有大量实验证明利用拉曼光谱能够区分不同物种的血液[2]。
在拉曼光谱中, 光谱信息和预测成分之间在某些波段不存在特定的相关关系, 除此之外, 各个波长点之间也存在严重的多重共线性[3]。 如果采用全光谱数据进行建模, 不仅会增加校正模型的复杂性, 而且其中一些噪声变量还会降低模型的预测能力。 文献[4, 5, 6, 7, 8, 9]中提到利用选定的波长点或波长区间而不是全光谱数据进行建模可以有效的提高校正模型的性能。 因此, 研究波长选择算法对于简化校正模型和提高校正模型的性能具有重要的实际意义。
现有的波长选择方法大致可以分为三类。 第一类是利用与校正模型相关的统计信息对波长点或波长区间进行评估, 一般是对各个波长点或波长区间逐一考察, 决定该剔除哪一些波长点或波长区间, 典型方法有区间偏最小二乘法(IPLS)[5]、 移动窗口偏最小二乘法(MWPLS)[6]、 SVP(stability and variable permutation)[7]等。 第二类是根据各个波长点的回归系数、 协相关系数等一些指标进行排序, 然后选择剔除靠后的那些波长点, 例如偏最小二乘无信息剔除法(UVE-PLS)[8]、 竞争自适应重加权抽样法(CARS)[4]和变量置换总体分析法[9]等。 第三类是将波长选择看作全局优化问题, 利用智能优化算法对波长点进行选择, 遗传算法(GA)、 粒子群优化算法(PSO)、 模拟退火算法(SA)等算法均属于此类。
本文提出了一种基于神经网络的拉曼光谱波长选择方法。 该方法利用神经网络的自学习能力, 自适应得到各个波长点对校正模型的贡献权重, 然后将权重的均值作为阈值, 去掉权重低于阈值的波长点, 从而达到波长选择的目的。 利用该方法对人与动物血清的拉曼光谱进行波长选择, 将波长选择后的光谱进行分类实验, 取得了较好的实验结果。
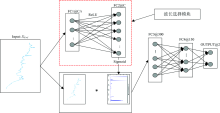
拉曼光谱中包含大量与建模无关的波长点, 本文利用神经网络学习到各个波长点对校正模型的贡献权重, 通过学得的权重筛选出与校正模型相关的波长点用于建模, 有效的提升了校正模型的性能。 波长选择网络的结构如图1所示, 为了方便描述, 将这个网络结构命名为WSNet(wavelength selection network)。 WSNet分为波长选择和校正模型两个部分, 波长选择模块(红色虚线框中的结构)由FC1层和FC2层两个全连接层构成, 校正模型由FC3层、 FC4层两个全连接层和输出层(OUTPUT层)构成。
FC1层的作用是对输入的光谱数据进行降维, 以防止误差反向传播时波长选择模块的网络结构梯度更新过大。 FC1层的变换过程可以表示为
WFC1为FC1层的权值矩阵, bFC1为FC1层的偏置向量, AFC1(· )为FC1层的激活函数, 保证权重大于零, xFC表示FC1层的输出。 FC1层的神经元个数为C/r, C为输入的波长点个数, r(r> 1)为降维倍数。
FC2层利用降维后的数据学习出各个波长点对建模的贡献权重(s)
WFC为FC2层的权值矩阵, bFC2为FC2层的偏置向量, AFC2(· )为FC2层的激活函数。 FC2层神经元个数与输入的波长点个数相同均为C。 FC2层学习到的各个波长点的权重相差较小, 不容易筛选。 考虑到光谱中对建模有贡献的波长点往往较少, 因此对权重增加稀疏约束。 因此构造如下的损失函数J(s)
Loss(· )是原损失函数, λ 是控制稀疏程度的参数。
WSNet网络选择具有以下优点:
(1)对学得的波长点权重增加稀疏约束, 更容易选择与校正模型相关的波长点用于建模: 通过网络学习到的各个波长点的权重差异较小, 不容易选取需要的波长点, 而且在拉曼光谱中对建模有贡献的波长点在全光谱中是稀疏的, 因此对权重增加了稀疏约束, 极大的减少了用于选择的波长点, 更容易确定筛选的阈值, 选出与建模相关的波长点。
(2)波长选择模块计算权值的方式与注意力机制[10]的功能相吻合, 能计算出各个波长点对校正模型的贡献: 注意力机制可以通过扫描全局获取重点关注的目标区域, 在目标区域投入更多的资源, 获取关注目标的更多细节。 波长选择网络利用注意力机制, 能够获取到对模型有贡献的波长点, 能有效的提高模型的性能。
实验选用人、 比格犬、 新西兰兔三种动物的血清作为实验样本, 其中包括采集于重庆市西南医院的人血清样本110例和采集于重庆市中药研究院的动物血清样本216例(其中犬116例、 兔100例), 共计326个样本, 人与动物血清样本清单如表1所示。
| 表1 血清样本清单 Table 1 Serum sample list |
采集的血清样本均通过取沉淀后的血液样本的上层清液获得, 采用乙二胺乙酸二钠(EDTA)抗凝管盛放。 每次实验均采用移液管取2 μ L血清样本置于石英载玻片上, 然后利用光谱仪进行测量, 为了减少实验误差, 每个样本都要测量4次, 取平均值作为最终测量值。
实验仪器主要包括计算机、 光谱仪、 石英载玻片、 移液管、 抗凝管等。 光谱仪是采用小型拉曼光谱仪海洋IDR-Micro-785, 光谱的激发波长为785 mm, 分辨率4 cm-1, 扫描范围为200~2 000 cm-1, 激发功率为18.8 mW, 曝光时间为5 s。
虽然人、 犬、 兔的血液中都包含了蛋白质、 葡萄糖、 色氨酸和血红素等物质[10], 使得这三种动物血清的拉曼光谱具有相似的谱峰(如图2所示), 但是不同物种的遗传基因不同, 导致了血清中的某些成分的浓度和化学特性是不同的[12], 使其在对应的谱峰上存在微弱的差异, 可以利用这些差异对人和动物的血清进行区分。
原始光谱在200~630 cm-1波段受石英载玻片的影响, 在1 710 cm-1波段之后无明显的波峰, 所以实验选择了598~1 718 cm-1波段共800个波长点进行分析。 从图2 中可以看出样本之间的光强差异很大, 为了消除样本之间的光强差异, 每条光谱都除以该光谱中最大的值, 将光强归一化到0~1之间; 然后利用移动平均平滑法去除光谱中的噪声, 滑动窗口大小为9。
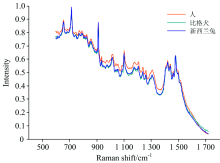 | 图3 人、 犬、 兔血清的拉曼光谱归一化后的对比Fig.3 Raman spectra of human serum, dog serum and rabbit serum after normalization |
波长选择模块中输入的光谱样本波长点个数C=800, 降维倍数r=5, 所以第一个全连接层(FC1层)的神经元个数为160个, 第二个全连接层(FC2层)的神经元个数与输入的波长点个数相同均为C=800, 神经网络校正模型中的两个全连接层(FC3和FC4层)的神经元个数分别为300个和150个, OUTPUT层有两个神经元, 为二分类。 本实验所采用的目标函数是交叉熵, 计算公式如式(4)
式(4)中, yi为第i个样本的测量值,
式(5)中, sij第i个样本的第j个波长点处的权重。
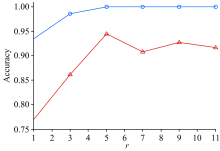
降维倍数r是网络结构中非常重要的一个参数, r过大或过小都不能使校正模型达到最佳性能, 如图5所示, 当r=5时校正模型的分类准确率最高, 在r< 5时, 校正模型在校正集和测试集的的分类准确率都较低; 在r> 5时, 校正集上的分类准确率基本不变, 但是测试集上的分类准确率略有下降, 因此本文选取的降维倍数为5。
在实验的过程中, 将326例光谱样本按比例取出1/3(共计109例, 37例人、 39例犬、 33例兔)作为测试集对校正模型的性能进行测试, 余下的2/3(共计217例, 73例人、 77例犬、 67例兔)作为校正集用于校正模型的训练。 将利用WSNet得到的光谱与全光谱数据分别使用神经网络(neural network, NN)和PLS-DA建模进行对比, 同时将WSNet与经典的波长选择算法UVE(uninformative variables elimination)进行对比, 并使用分类准确率、 ROC曲线(receiver operating characteristic curve)和AUC(area under curve)对这些校正模型的性能进行评价。 ROC曲线的横坐标为假正率(FPR), 纵坐标为真正率(TPR), TPR和FPR的计算公式如式(6)
式(6)中, TP为真正例, FP为假正例, FN为假反例, TN为真反例。 ROC曲线所包围的面积称之为AUC, AUC的值越大, 证明模型的分类精度越高。 AUC的计算公式如式(7)
式(7)中, N为样本的数量。
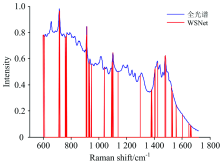
图6(a)给出了WSNet学得的光谱权重, 只有109个波长点的权重不为零。 利用式(5)计算出一个阈值, 将权重小于阈值的波长点也去掉。 最终, WSNet选择了42个波长点用于建模, 如图6(b)所示。
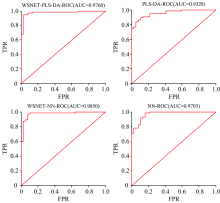
将波长选择前后的光谱利用PLS-DA和NN进行建模, 实验结果表明, 四种校正模型在校正集上都取得了非常好的效果, 如表2所示, NN和WSNet-NN均未错分, 分类精度都达到了100%, PLS-DA和WSNet-PLS-DA分别错分了2例和1例, 分类精度也分别达到了99.539%和99.078%。 在测试集上, WSNet-NN分类精度优于其他三种模型, 仅错分了6例, 分类精度最高为94.495%; PLS-DA错分了10例, 分类精度为90.826%; NN和WSNet-PLS-DA均错分了8例, 分类精度相同为92.661%。 从上述的实验结果可以看出, 利用WSNet对光谱进行波长选择有利于提升校正模型的性能。
| 表2 分类结果对比 Table 2 Comparison of classification results |
PLS-DA, WSNet-PLS-DA, NN和WSNet-NN在测试集上的ROC曲线如图7所示。 通过式(7)计算可以得到这四种校正模型的AUC值, PLS-DA和WSNet-PLS-DA的AUC值分别为0.976 0和0.932 8, NN和WSNet-NN的AUC值分别为0.985 0和0.970 3, 从这四种校正模型的AUC值可以看出, 利用波长选择后的光谱进行建模能提升校正模型的性能, 让其具有更优的鉴别能力。
分别使用WSNet和UVE对光谱进行波长选择, 如图8所示, WSNet选择的波长点个数远少于UVE。 在稀疏约束的作用之下, WSNet去掉了绝大部分的波长点, 只选择了42个波长点, 而UVE却选择了486个波长点。 将WSNet和UVE波长选择后的光谱分别用于建立PLS-DA校正模型, 分类结果如表3所示, WSNet-PLS-DA在校正集和测试集上的分类准确率分别为99.078%和92.661%, UVE-PLS-DA在校正集和测试集上的分类准确率分别为98.156%和91.743%。 WSNet-PLS-DA在校正集和测试集的分类准确率相比于UVE-PLS-DA都要高一些。
| 表3 Raman-WSNet-PLS-DA和UVE-PLS-DA分类结果对比 Table 3 Comparison of Raman-WSNet-PLS-DA and UVE-PLS-DA classification results |
WSNet-PLS-DA和UVE-PLS-DA在测试集上的ROC曲线如图9所示。 根据AUC的计算公式(7)计算可以得到各自的AUC值分别为0.976 0和0.965 8。 从中可以看出, 相比于UVE, WSNet不仅能去掉更多的波长点, 而且对校正模型的性能提升也更为显著。
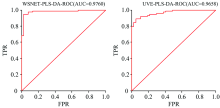 | 图9 Raman-WSNet-PLS-DA和UVE-PLS-DA ROC曲线对比Fig.9 Comparison of Raman-WSNet-PLS-DA and UVE-PLS-DA ROC curves |
拉曼光谱的各个波长点之间存在严重的多重共线性, 采用全拉曼光谱数据用于NN和PLS-DA模型的构建, 得到的校正模型性能不够好。 利用WSNet对拉曼光谱进行处理, 去除了光谱中对建模有影响和无贡献的波长点, 对于校正模型的性能有显著的提升。 WSNet与经典的波长选择算法UVE相比也具有明显的优势, UVE算法的核心思想是在原光谱中引入人工随机变量, 将随机变量和光谱一起进行建模, 将原光谱中各个波长点的回归系数与随机变量回归系数的平均值进行比较, 如果原光谱波长点的回归系数低于人工随机变量回归系数的平均值, 就认为该变量是噪声变量, 应该被剔除。 由于UVE算法引入随机变量, 导致该算法存在一些随机因素, 使得每次筛选的结果都不相同, 但都表现出很强的一致性。 UVE在剔除波长点的过程中参考的是人工随机变量, 即随机噪声, 所以剔除的仅仅是影响建模的噪声波长点, 对建模既没有贡献也没有影响的波长点并不会剔除。 WSNet相比于UVE就要“ 激进” 许多。 WSNet由于是通过神经网络进行波长选择的, 同样也存在一些随机因素, WSNet是通过神经网络自学习能力自适应得到每个波长点的权重, 从而利用学得的权重对波长点进行筛选。 同时在权重学习的过程中加入了稀疏约束, 所以学得权重非常稀疏, 利用稀疏的权重对波长点进行筛选会变得更加容易。 通过上面的实验可以看出, UVE所选择的波长点比较多, 它并没有筛选掉太多的波长点, 是一种比较“ 保守” 的波长选择算法。 相比之下, WSNet所选择的波长点较少, 而且建立的校正模型性能更好, 证明了WSNet是有效的。
利用神经网络的学习能力提出了一种基于神经网络的拉曼光谱稀疏波长选择的新方法— — WSNet。 它能自适应的选择拉曼光谱中包含信息的波长点, 从而建立更加高效的校正模型。 将波长选择前后的拉曼光谱利用同一种方法建模进行对比, 进一步验证了该方法有利于校正模型分类精度的提升, 最高达到了94.495%。 同时还与UVE波长选择算法进行了对比, 实验结果表明WSNet在波长点选择的个数和校正模型的分类精度方面都优于UVE算法, 精度达到了92.661%。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|