作者简介: 张秋兰, 女, 1979年生, 南昌大学化学学院副教授 e-mail: qlzhang@ncu.edu.cn
石墨烯量子点(GQDs)以其优异的性能在生物医学领域引起了广泛的关注, 但其潜在的毒性研究较少。 将荧光和紫外光谱法结合化学计量学研究GQDs对胰蛋白酶结构和功能的影响。 从荧光猝灭实验可知, GQDs可猝灭胰蛋白酶的固有荧光并抑制胰蛋白酶的生物活性。 当加入不同浓度的GQDs, 胰蛋白酶在350 nm处的荧光发射峰的强度随之降低且蓝移(350~344 nm), 表明GQDs可改变胰蛋白酶所处的微环境, 使其疏水性增加; 与此同时, GQDs浓度越高, 胰蛋白酶荧光变化越明显, 说明GQDs对胰蛋白酶可能有潜在毒性。 通过圆二色谱实验可知胰蛋白酶的α-螺旋结构由19.12%下降至16.23%, 说明GQDs加入诱导胰蛋白酶的二级结构发生改变, 使胰蛋白酶骨架松弛; 三维荧光光谱实验进一步说明GQDs的存在不仅改变了胰蛋白酶所处的微环境并使胰蛋白酶的构象发生变化。 蛋白质氨基酸残基的微环境由蛋白质分子的构象所决定, 当蛋白质的生色团所处的微环境发生变化时, 其紫外-可见吸收光谱也随之发生变化。 由于生命作用体系都比较复杂, 测量所得到的波谱数据中大部分信息是隐含和重叠的, 因此需要利用和发展有效的生物信号采集、 转导、 数据处理和解析方法, 把能对生命现象做出解释的有用信息尽可能多地从测量数据中挖掘出来。 为获取足够而有效的生命化学信息, 该研究用连续滴定技术采集多维光谱数据, 运用多元曲线分辨-交替最小二乘法(MCR-ALS)解析光谱数据矩阵, 从重叠严重的光谱中同时得到定性(各组分光谱及作用过程中复合物的真实存在)和定量(各组分的浓度变化趋势)信息, 从而认识GQDs与胰蛋白酶在作用中达到平衡时各组分的状态和整个动态变化过程。 MCR-ALS的解析结果为进一步了解GQDs与胰蛋白酶相互作用的动力学过程提供了依据, 说明GQDs可以与胰蛋白酶相互作用, 并形成GQDs15-胰蛋白酶复合物。 该研究为GQDs可能存在的毒性风险研究提供了信息。
With super properties, such as photoluminescence properties, edge effect, low cytotoxicity and great biocompatibility, graphene quantum dots (GQDs) have attracted great attention in biological and biomedical applications. The potential toxicity investigations of GQDs still need to involve. Few studies have been illuminated that GQDs could alter the function and structure of trypsin. The molecular interaction between trypsin and GQDs was systematically researched through the combination of multi-spectroscopic and chemometrics approaches. The fluorescence quenching experiment showed that GQDs quench the intrinsic fluorescence of trypsin and inhibit the biological activity of trypsin. When different concentrations of GQDs were added, the fluorescence emission peak intensity of trypsin at 350 nm continuously decreased and had a blue shifted (350 to 344 nm), indicating that GQDs could change the microenvironment of trypsin and increase its hydrophobic. Meanwhile, the higher the concentration of GQDs, the more obvious the change of trypsin fluorescence, indicating that GQDs interacts and changes the secondary structure of the macromolecule. The microenvironment of protein amino acid residues is determined by the conformation of protein molecules. The spiral structure of proteinase decreased from 19.12% to 16.23% in the circular dichroism experiment indicated that the addition of GQDs induced the alteration of the secondary structure of trypsin and relaxed the trypsin framework. The three-dimensional fluorescence further indicated that the conformation of trypsin changed with the addition of GQDs. When the microenvironment of the chromophores of serum albumin changes, its UV-visible absorption spectrum also changes. Due to the complex life system, most of the information in the test of spectrum data is implicit and overlapping. We need to use and develop the effective biological signal collection, transduction, data processing and analysis method to get the useful information that can explain life as much as possible from the measured data. To obtain sufficient and effective chemical information of life, this study adopts the continuous titration technique collecting multidimensional spectrum data. An expanded UV-Vis spectral data matrix was analyzed by the multivariate curve resolution-alternating least squares (MCR-ALS) chemometrics approach. To further understand the state and the whole dynamic change process of each component when GQDs and trypsin reached equilibrium in action, the qualitative (spectrum of each component) and quantitative (the changing trend of concentration) information were obtained from the heavily overlapped spectra. The analytical results of MCR-ALS provide a basis for further understanding of the kinetic process of the interaction between GQDs and trypsin, indicating that GQDs can interact with trypsin and form GQDs15-trypsin complex. The results offered insights into the binding mechanism of GQDs with trypsin and significant information for possible toxicity risk of GQDs to human health.
荧光石墨烯量子点(GQDs)具有良好的化学惰性和生物相容性, 以及荧光发光可调和荧光上转换等特性, 已成为生物成像、 药物运输、 疾病检测和荧光探针等领域的研究热点[1]。 因GQDs在靶向药物递送和改变血脑屏障等方面的应用受到了极大的关注, 人们对评估GQDs在生物系统中的潜在毒性风险越来越感兴趣[2]。 已有文献报道石墨烯纳米材料可能导致严重的细胞毒性和肺部疾病[3], 但有关GQDs毒性研究或生物学效应的报道较少。 Huang等[4]和Lu等[5]分别研究了GQDs与人血清白蛋白和DNA的相互作用, 为评价GQDs在生物医学领域的毒性和生物学效应提供了依据。 然而, 有关GQDs的毒理学研究还有待深入。
胰蛋白酶(trypsin, Tryp)是胰腺分泌的一种丝氨酸蛋白酶, 其分子为23 300, 由223个氨基酸残基组成, 在脊椎动物的消化、 蛋白质成熟、 凋亡、 凝血、 免疫反应等生理过程中起着重要的作用。 近年来, 胰蛋白酶得到了广泛的应用, 对胰蛋白酶的研究也时有报道, 例如药物分子和纳米粒子与胰蛋白酶的相互作用[6, 7], 谷胱甘肽包裹的CdTe量子点和CdSe量子点与胰蛋白酶的作用[8, 9]。 然而, 在分子水平上GQDs对胰蛋白酶的毒性研究尚未见报道。
纳米材料与生物大分子作用以紫外可见分光光度法、 荧光和红外光谱、 圆二色谱法等为主要技术, 由于生命作用体系都比较复杂, 需要利用和发展化学计量学方法, 把能对生命现象做出解释的有用信息尽可能地从测量数据中挖掘出来。 继以往将化学计量学方法用于银纳米粒子与血清蛋白作用后研究[10], 本工作将多维数据解析用于GQDs与胰蛋白酶的研究, 为GQDs对人体健康的潜在毒性风险提供有价值的信息。
石墨烯量子点(graphene quantum dots, GQDs, 南京先丰纳米材料科技有限公司, 纯度~90%, 量子点< 10 nm, 量子产率~5%, 分子量78)用二次蒸馏水配成1.0× 10-2 mol· L-1的溶液备用, 4 ℃以下冰箱保存。 胰蛋白酶(国药集团化学试剂有限公司, 分子量23 800, 干燥失量≤ 10.0%, 酶活力(μ · g-1)25 000, 淡黄色粉末用二次蒸馏水溶解成2.5× 10-3 mol· L-1的溶液备用。 N-苯甲酰-L-精氨酸乙酯盐酸盐(上海生工, 白色结晶或粉末, 溶于水, 纯度大于98%, 分子量342.82)用二次蒸馏水溶解成1.0× 10-3 mol· L-1的溶液备用。 日立F-7000(日本日立公司)荧光分光光度计(配备1.0 cm荧光比色皿), 狭缝宽度Ex=5 nm, Em=10 nm, 扫描电压为700 V, 扫描速度2 400 nm· min-1; Agilent 8453紫外可见光光度计(美国安捷伦公司); 圆二色谱仪(法国Bio-Logic公司); 超级恒温水槽ZC-10(宁波天恒仪器厂)。
将3.0 mL pH 7.4的Tris-HCl缓冲溶液加入比色皿中, 根据实验需要依次加入适量的GQDs和Tryp溶液。
实验1: 固定Tryp的浓度(4.00× 10-6 mol· L-1), 再加入不同量的GQDs, 使其浓度分别为0, 1.67, 3.33, …, 13.33× 10-5 mol· L-1(浓度间隔1.67× 10-5 mol· L-1), 放置5 min以达平衡, 在298 K测其荧光光谱。
实验2: 固定Tryp的浓度(3.33× 10-6 mol· L-1), 加入不同量的GQDs, 使[GQDs]:[Tryp]=0, 5, 10, 15, 20, 在298 K测其圆二色谱。 所得到的CD(circular dichroism, CD)谱为三次扫描的平均值, 并通过缓冲信号进行校正。
实验3: 固定Tryp的浓度(4.17× 10-6 mol· L-1), 再加入GQDs, 使[GQDs]:[Tryp]=10, 在298 K下测其三维荧光。
实验4: N-苯甲酰-L-精氨酸乙酯盐酸盐(BAEE)在波长253 nm下的紫外吸收远小于其水解产物N-苯甲酰-L-精氨酸(BA)的紫外吸收[11]。 在胰蛋白酶的催化下, BAEE酯键水解产物BA逐渐增多, 体系的紫外吸收也随之增加。 胰蛋白酶的活性以Δ A253 nm计算。 在3.0 mL Tris-HCl缓冲溶液中固定Tryp的浓度(4.17× 10-6 mol· L-1), 然后加入不同量的GQDs(0~8.33× 10-5 mol· L-1, 间隔8.33× 10-6 mol· L-1), 在37 ℃孵化2 h后, 再加BAEE(1.67× 10-4 mol· L-1), 测GQDs存在和不存在下的紫外吸收光谱。
实验5: (UV-Vis(ultraviolet visible, UV-Vis)法, DTryp): 固定Tryp的浓度(1.67× 10-5 mol· L-1), 加入不同浓度的GQDs(0~3.33× 10-4 mol· L-1, 间隔1.67× 10-5 mol· L-1)。
实验6: (UV-Vis法, DGQDs): 固定GQDs的浓度(1.67× 10-5 mol· L-1), 加入不同浓度的Tryp(0~2.22× 10-6 mol· L-1, 间隔1.11× 10-7 mol· L-1)。
以上紫外光谱数据采集范围均为200~500 nm(间隔1 nm, 301个数据点), 见表1。
| 表1 不同实验条件下得到的2个波谱数据矩阵 Table 1 The experimental conditions to build up the extension matrixes |
经典的多元曲线分辨-交替最小二乘法(multivariate curve resolution-alternating least squares, MCR-ALS)法在处理体系测量数据时所得到的信息相对简单孤立, Tauler对其进行了拓展和改进[12]。 本工作通过UV-Vis技术采用两种滴加方式获得2个波谱数据矩阵(见表1)后, 将这2个矩阵Dtrypsin(实验5)和DGQDs(实验6)在列的方向组成一扩展矩阵, 再用MCR-ALS解析; 多维数据解析方法最大的优势在于可获得多维阵唯一的分解结果, 即分辨出体系中感兴趣组分的相对浓度和光谱趋势图。
式(1)中
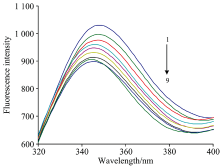
酶的荧光性质来自色氨酸、 酪氨酸、 苯丙氨酸残基。 固定胰蛋白酶, 加入不同浓度的GQDs。 表明GQDs可改变胰蛋白酶所处的微环境, 使其疏水性增加[14]。 与此同时, GQDs浓度越高, 胰蛋白酶荧光变化越明显(1→ 9, 下降了近16%), 说明GQDs可与胰蛋白酶作用并改变大分子的二级结构[15]。
2.2.1 圆二色谱法
圆二色谱是一种准确灵敏的检测蛋白质构象变化的方法。 利用远紫外(200~260 nm)可测定蛋白质的二级结构。 在没有和存在GQDs的情况下, 胰蛋白酶的CD谱如图2所示。 胰蛋白酶在210 nm左右呈负谱带, 不存在正峰。 在胰蛋白酶溶液中加入GQDs后, CD信号出现小幅下降, 但峰的形状和位置没有明显变化, 说明与GQDs结合时胰蛋白酶的二级结构展开, 但基本结构保持完整。 用公式[16]计算得到胰蛋白酶的α -螺旋结构由19.12%下降至16.23%, 进一步说明GQDs加入可诱导胰蛋白酶的二级结构发生改变, 使胰蛋白酶骨架松弛[17]。
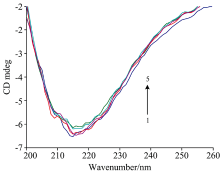 | 图2 GQDs与胰蛋白酶作用的CD图 cTryp=3.33× 10-6 mol· L-1, 1→ 5表示[GQDs]:[Tryp]=0, 5, 10, 15, 20Fig.2 The CD spectra of the interaction of GQDs with trypsin cTryp=3.33× 10-6 mol· L-1, [GQDs]:[Tryp]=0, 5, 10, 15, 20 |
2.2.2 三维荧光光谱法
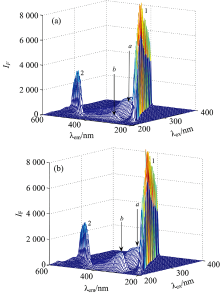
三维荧光光谱可证明蛋白质构象特征的变化。 图3(a)和 (b)是胰蛋白酶和石墨烯量子点的三维荧光光谱。 峰1和峰2是瑞利散射峰(λ ex=λ em)和二阶散射峰(2λ ex=λ em)[19]。 峰a(λ ex=280 nm, λ em=344 nm, I=1 548)与色氨酸和酪氨酸残基从π → π * 跃迁的荧光特性有关, 加入石墨烯量子点后[λ ex=280 nm, λ em=337 nm, IF(fluorescence intensity)=1 529]反映了胰蛋白酶的二级结构变化。 峰b(λ ex=225 nm, λ em=322 nm, I=1 474)是胰蛋白酶多肽骨架结构的荧光特征, 主要是由n→ π 的跃迁引起的, 表明石墨烯量子点存在下胰蛋白酶的二级结构变化(λ ex=225 nm, λ em=318 nm, I=366)。 加入GQDs后, 胰蛋白酶峰a和峰b的荧光强度值明显下降并出现蓝移, 这些数据表明GQDs与胰蛋白酶作用使胰蛋白酶构象的变化[19]。
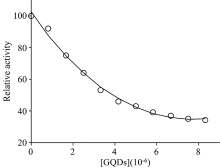
随着GQDs的加入, 胰蛋白酶活性降低, 但不能被完全抑制(图4, 纵坐标为相对活性)。 通过GQDs酶活性下降的半抑制浓度IC50为4.35× 10-6 mol· L-1, 说明GQDs对胰蛋白酶活性相对较弱的抑制作用。 这可能是GQDs在酶中有较高的高生物利用度, 胰蛋白酶的活性位点被GQDs稳定和保护[20]。
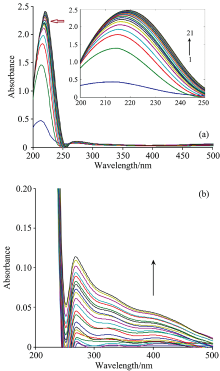
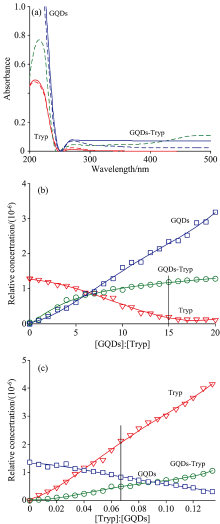
紫外-可见吸收是测定小分子与生物分子相互作用的一种简单而常用的技术。 如图5(a)所示, 胰蛋白酶在大约215和276 nm处呈现出两个吸收峰, 在215 nm处的吸收峰随着GQDs的加入逐渐向220 nm移动直至平衡, 而在276 nm处的吸收峰基本保持不变。 从图5(b)可知, 随着胰蛋白酶的加入, GQDs的峰强度明显增加, 峰位移不明显。 图5(a)和(b)的光谱重叠严重, 难以区分GQDs-胰蛋白酶复合物的光谱而无法分析反应过程。 通常利用化学计量学扩展多元曲线分辨-交替最小二乘法(MCR-ALS)可从复杂系统的重叠波谱信号中提取较多的额外信息, 例如组分的浓度分布、 纯光谱、 特别是确定GQDS-胰蛋白酶复合物等方面的信息。
通过实验5和实验6得到扩展光谱数据矩阵[Dtrypsin, DGQDs], 然后利用奇异值分解(SVD)方法计算矩阵, 得到与化合物相关重要因子个数, 得到的前4个特征值分别为113.6, 30.7, 10.3和1.4, 说明反应体系中有3个主要因素可能与3个化合物有关, 分别为胰蛋白酶、 GQDs和GQDs-胰蛋白酶复合物。 MCR-ALS法提取的胰蛋白酶和GQDs的光谱[虚线, 图6(a)]与实测图(实线)吻合较好。 此外, 还得到了GQDs-胰蛋白酶复合物的紫外-可见吸收光谱(虚线)。 计算与实测光谱曲线吻合较好, 说明MCR-ALS解析得到的浓度曲线合理可靠。 如图6(b)所示, 随着GQDs的加入, GQDs-胰蛋白酶复合物浓度逐渐升高, 同时胰蛋白酶浓度下降, 在[GQDs]:[胰蛋白酶]=15时, 体系趋于平衡; 从图6(c)可知, 随着胰蛋白酶的加入, GQDs浓度呈逐渐下降趋势, GQDs-胰蛋白酶复合物浓度增加直至体系基本平衡。 MCR-ALS法的解析结果为进一步了解GQDs与胰蛋白酶相互作用的动力学过程提供了依据, 说明GQDs可以与胰蛋白酶相互作用, 并形成GQDs15-胰蛋白酶复合物。
采用光谱法结合化学计量学方法研究GQDs与胰蛋白酶的相互作用。 三维荧光光谱法和圆二色谱实验说明GQDs的存在不仅改变了胰蛋白酶所处的微环境并使胰蛋白酶的构象发生变化。 运用多元曲线分辨-交替最小二乘法(MCR-ALS)解析扩展光谱数据矩阵, 进一步认识GQDs与胰蛋白酶在作用中达到平衡时各组分的状态和整个动态变化过程。 而酶活性实验表明GQDs会影响胰蛋白酶的活性。 该研究从分子水平研究GQDs与生物大分子的作用机制, 并为GQDs的毒副作用研究提供了有价值的信息。 下一步计划研究不同尺寸和层数的石墨烯量子点对生物大分子构象和蛋白酶活性的影响。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|