作者简介: 李鑫星, 1983年生, 中国农业大学信息与电气工程学院食品质量与安全北京实验室副教授 e-mail: lxxcau@cau.edu.cn
应用紫外(Ultraviolet, UV)光谱技术对水产养殖水质总氮含量进行快速检测。 为了消除各种系统误差与偶然误差对模型预测性能造成的影响, 将88个水样的总氮浓度实测值数据和光谱吸光度数据作为原始数据, 将模型建立分为样本集划分、 数据预处理、 特征波段提取、 模型选择与LV数量选择5个阶段, 以求达到最优预测效果, 其中前4个阶段分别使用多种方法进行比较。 结果证明每个阶段都是必不可少的, 只有通过对比其优劣才能找到最适合总氮含量测定的建模过程及方法。 首先用浓度梯度(CG)法对原始数据进行相同的样本集划分处理, 然后在此基础上分别建立主成分回归(PCR)、 逐步回归(SR)和偏最小二乘回归(PLSR)三种模型, 选择预测效果最好的PLSR作为本文的预测模型。 PLSR的建模效果会在很大程度上受到潜在变量(LVs)数量的影响, 通常选取模型预测均方根误差RMSEP值最小时所对应的LV个数为最优LV个数。 其次, 选用CG法、 随机抽样(RS)法、 Kennard Stone(KS)法和SPXY法4种样本集划分算法对样本进行处理, 并对所建立的PLSR模型预测效果进行比较, 最终选择SPXY算法作为最优样本划分算法。 然后在对样本集进行SPXY法划分的基础上, 运用多种预处理算法对光谱吸光度数据进行预处理, 包括小波变换(WT)、 一阶导数法(Der1st)与二阶导数法(Der2nd)三种单一算法和小波变换与两种导数法的组合预处理算法WT-Der1st和WT-Der2nd。 然后在预处理的基础上分别使用连续投影变换(SPA)和逐步回归(SR)两种特征波段提取方法, 对比可知, SPA特征提取方法比SR的提取效率高且建模效果好。 SPA算法既可以大大地简化模型, 又可以在一定程度上提升模型的预测精度。 基于WT-Der1st-SPA提取的特征波段为218 nm, 与总氮特征波段区间相一致, 由此说明该方法比较科学。 综合上述建立的10个PLSR模型, 考虑到预测精度与模型复杂度2个因素, 最终选择基于WT-Der1st-SPA建立的PLSR模型作为最优模型, 该模型预测决定系数 r2为0.996, 预测均方根误差RMSEP为0.042 mg·L-1。 由此可见, 所建立的模型预测效果非常好, 可以快速准确地测定水体的总氮含量, 为实现光谱技术在水产养殖其他水质监测指标的在线检测以及快速测定提供了经验。
The paper is intended to achieve rapid determination of total nitrogen (TN) concentration by using Ultraviolet (UV) spectroscopy technology, which was one of the most important indicators to measure the pollution degree in aquaculture water. The original dataset used in the paper contains 88 samples data with actual concentration value and spectral absorbance value. It is helpful to select the optimal model through the five stages that include sample set division algorithms, data preprocessing algorithms, feature band extraction algorithms, model selection algorithms and latent values (LVs) selection method. In the first four stages, the comparison results of different methods show that each stage is necessary, and only by comparing the advantages and disadvantages of modeling results with various algorithms can we find the most suitable modeling process and method. First of all, the original sample set is processed by the concentration gradient (CG) method, then three models are built which respectively are principal component regression (PCR), stepwise regression (SR) and partial least squares regression(PLSR), and it proves that the PLSR is the best prediction model. The number of LVs can greatly influence the accuracy of model, and usually when the value of the model root mean square error of prediction (RMSEP) is the minimum, the LV number is optimal. Secondly, it is testified that the SPXY algorithm is the best by comparing the effect of random sampling (RS) algorithm, concentration gradient (CG) algorithm, kennard stone (KS) algorithm and SPXY algorithm. Thirdly, based on SPXY algorithm, the paper uses five preprocessing algorithms which are wavelet transform (WT) method, first derivative (Der1st), and second derivative (Der2nd) three single preprocessing algorithms, WT-Der1st and WT-Der2nd. Fourthly, according to the results of data processing, using successive projections algorithm (SPA) and stepwise regression (SR) for feature band extraction algorithms, the results show that the extraction efficiency of SPA not only can greatly reduce the complexity of model, but also improve the prediction accuracy. The feature band extracted based on WT-Der1st-SPA is 218 nm, which is consistent with the characteristics of total nitrogen band range, indicating the method was relatively scientific. Finally, considering the prediction accuracy and complexity of model, the PLSR based on WT-Der1st-SPA with the best results with the determination coefficient ( r2) and RMSEP being 0.996 and 0.042 mg·L-1 for the prediction set in 10 models. In short, the prediction model established could be applied to the rapid and accurate determination of total nitrogen concentration. Moreover, this study laid the foundation for further implementation of online analysis of aquaculture water and rapid determination of other water quality parameters.
近年来, 我国水产养殖在各类养殖行业中占有十分突出的地位, 为保障食物供给、 改善饮食结构、 促进经济增长做出了巨大贡献。 水产品的脂肪含量较低, 蛋白质的含量较为丰富, 深受大众的喜爱, 但水产品一旦出现质量安全问题, 就可能带来极其严重的后果。 水产品质量与水产养殖水环境密切相关, 而水中总氮(TN)含量是评价水体受污染严重程度的关键性指标之一[1]。 随着经济的迅猛增长, 工业废水、 生活污水中的含氮物质有可能进入养殖池塘, 造成其水质污染; 在集约化养殖过程使用的是高蛋白饵料, 只有一少部分被鱼虾类等摄入体内转换为蛋白质, 更大一部分则是在池塘中残留, 造成养殖池塘含氮营养物质水平过高。 如果超标排放则会破坏水体自身的平衡, 导致水体中含氮物质的富集, 造成水体中的水藻和浮游生物的快速生长, 致使水体中的溶解氧浓度下降, 鱼虾类等生物大面积死亡, 最终使得水质恶化而不适合于水产养殖。
紫外(Ultraviolet, UV)光谱技术能够实现快速无损检测, 且检测成本较低, 近年来已在水质检测领域得到广泛应用。 郑一力等[2]以金镶玉竹叶片为样本, 建立了4种竹叶片氮含量高光谱估测模型, 最后比较发现基于主成分分析的BP神经网络构建的竹叶片氮含量估测模型效果较好。 赵进辉等[3]运用小波分析方法对原始光谱数据进行数据预处理, 建模效果明显改善。 刘思伽等[4]、 Wang等[5]采用SPA算法对农产品光谱数据进行特征波段的选择, 简化了模型并提升了其预测性能。 Chen等[6]、 Guo等[7]基于UV光谱技术分别建立了水质COD与重金属离子的PLS模型, 结果显示其预测值十分接近于真实值。 传统的总氮浓度检测方法通常与化学分析方法结合紧密, 但是这类方法通常过程繁复, 耗时长, 不适宜大范围使用。 目前, 光谱技术在水质COD含量和重金属离子的浓度检测方面应用较多, 且较为成熟, 但在水质氮磷含量的检测方面应用偏少。 光谱分析技术是水质监测领域一个十分有前景的应用方向, 与传统方法相比, 它的操作简易、 试剂消耗量小、 重复性良好、 测定精度较高且检测速度快, 非常适合于水质的快速在线检测[8]。 本文基于紫外光谱技术, 设计出一种快速无损, 又相对精确的水产养殖水质总氮含量快速检测方法。
本实验采用UV-2450 UV/Vis光谱仪, 其测定波长范围为190~900 nm, 吸光度的测量范围为-4~5 Abs, 光谱分辨率为1 nm。 通过查阅文献并结合水样所测吸光度值绘制的光谱曲线可知, 测量190~350 nm波段的吸光度即可满足需要。 每个样本测量5次, 计算5次的平均值为最后的光谱测量值。 本文的光谱数据处理与数据建模软件为Excel 2010、 Matlab R2016a。
本实验于2018年3月到6月进行实验数据的采集工作, 于2018年4月到9月集中进行实验数据处理工作。 实验样本采集地点是苏州大学试验养殖基地。 试验水样主要成分为硝酸盐水样总氮含量由专业检测机构检测, 水样光谱吸光度数据由实验室光谱仪采集。 采集的样本量共90个, 首先测定90个样本的总氮浓度值, 然后用光谱仪分别测定其在190~350 nm的吸光度值, 绘制光谱曲线图。 结合样本光谱数据与实测浓度值, 剔除2个明显不合规律的样本后剩余88个样本。
模型的建立需要经过样本集划分、 数据预处理、 特征波段提取、 LV数量选取与模型构建5个步骤, 每个步骤都是必不可少的环节, 其处理效果的好坏与否可能会影响到最终模型的预测效果。
1.3.1 样本集划分
样本集划分是依照特定方法把研究所用样本集分成建模集和预测集, 以分别用于模型的构建与验证, 样本集划分是建立模型的过程中必不可少的一步, 其划分是否科学将直接影响模型的预测效果。 随机抽样(random sampling, RS)法、 浓度梯度(concentration gradient, CG)法、 Kennard Stone(KS)算法和SPXY算法是常见的4种样本划分方法。 这4种方法各有特色, 通过比较不同方法所建立模型预测效果的优劣以确定最适合的方法[9, 10]。
1.3.2 光谱数据预处理
为了消除光谱仪在扫描过程中引发的噪声问题, 减轻多种外界干扰, 并简化数据处理过程, 因此对原始光谱进行数据预处理是一项十分有必要的操作。 小波变换(wavelet transform, WT)、 一阶导数法(first-derivative, Der1st)和二阶导数法(second-derivative, Der2nd)是常见的3种光谱数据预处理算法[11]。 在某些情况下, 小波变换与导数法相结合的方法小波-一阶导数法(WT-Der1st)和小波-二阶导数法(WT-Der2nd)会较3种单一预处理算法效果好。 比较经5种预处理算法处理后所建模型的效果, 选出效果最好的预处理算法。
1.3.3 特征波段提取
光谱数据是连续采集的多波段吸光度数据, 光谱多波段数据之间相关性非常高, 存在数据冗余现象。 运用特征波段提取算法能够减少数据冗余, 提升模型的运算效率。 连续投影变换(successive projection algorithm, SPA)与逐步回归(stepwise regression, SR)两种特征波段提取算法均能实现从所有采集的光谱波段中选择具有代表性的几个波段作为模型的输入, 从而使构建的模型更加简单, 并在一定程度上提升模型预测性能[12]。
1.3.4 潜在变量(latent variables, LVs)
LV数量是PLSR模型的一个内部参数, 它的数量会直接影响到PLSR模型的预测性能。 用于建模的LV个数过多或者过少, 均会降低PLSR的模型精度。 因此确定合适数量的LV至关重要, 通常会选择RMSEP值最小时所对应的LV个数为PLSR模型的最佳LV数量。
1.3.5 建模方法
逐步回归(stepwise regression, SR)、 主成分回归(principal component regression, PCR)和偏最小二乘回归(partial least squares regression, PLSR)是建立光谱定量模型中常见的3种线性模型。 当因变量与自变量之间存在显著的线性关系时, 这3种方法均较为适用[13, 14]。 比较分别运用3种方法所建模型的预测效果, 选择最适用的方法。
1.3.6 模型评价指标
通常用于评价定量模型精度的指标有决定系数与均方根误差。 R2表示建模集的决定系数, r2表示预测集的决定系数, 决定系数越接近于1, 模型精度越高。 RMSEC表示建模集的均方根误差, RMSEP表示预测集的均方根误差, 均方根误差越小, 模型精度越高。 决定系数越高, 均方根误差越小, 建立的模型越理想, 反之表示模型越不理想。
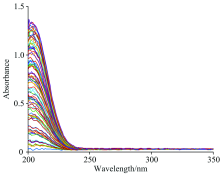
图2为水产养殖水体总氮样本的UV原始光谱曲线, 从图2中可以看出不同样本的光谱曲线走势大致相同, 没有呈现出显著性的差异。 由于不同样本的总氮浓度不同, 相对应的吸光度曲线会出现整体细微平移的现象。 由图2可知, 水体总氮浓度与吸光度之间的线性关系较为显著, 水体总氮浓度越高, 吸光度值越大。
由于本文的样本数据量有限, 因此建模集与预测集的比例选取会大大地影响模型的预测效果, 本文对两者比例的确定过程中将会十分谨慎。 通常建模集与预测集的比例在2:1~3:1之间比较合适[10], 因此本文选用了建模集:预测集=3:1和建模集:预测集=2:1两种方式, 通过比较选择最适合的比例进行建模。 第一种方式选择建模集与预测集为建模集66个, 预测集22个, 建模集:预测集=3:1, 其划分方式是从第2个样本起, 每隔3个样本取样为预测集, 剩余的为建模集。 第二种方式选择建模集59个, 预测集29个, 建模集:预测集≈ 2:1, 其划分方式是从第2个样本起, 每隔2个样本取样为预测集, 剩余的为建模集。 CG法对样本分别进行两种比例的划分, 其样本集统计结果如表1所示。
| 表1 CG法对样本集进行两种比例的划分结果 Table 1 Two sample division results of TN concentration of aquaculture water |
2.2.1 建模方法选取
为了确定小比例的样本集划分方法能否达到所需预测精度, 本文用两种比例的样本划分方式分别建立PCR, SR和PLSR 3种线性模型。 表2、 表3分别为建模集:预测集=3:1与建模集:预测集≈ 2:1的模型预测效果, 并通过对比找出最适合本文的建模方法。
| 表2 PCR, SR和PLSR的预测效果 (建模集=66, 预测集=22) Table 2 Prediction results of PCR, SR and PLSR models (calibration set number=66, prediction set number=22) |
| 表3 PCR, SR和PLSR的预测效果 (建模集=59, 预测集=29) Table 3 Prediction results of PCR, SR and PLSR models (calibration set number=59, prediction set number=29) |
由表2可知, 当模型建模集与预测集比例为3:1时, R2与r2均超过0.97, R2较高, 且二者差别为0.01左右, RMSE均小于等于0.1 mg· L-1, 且RMSEP与RMSEC相差不超过0.04 mg· L-1, 因此说明数据的质量比较好。 当模型建模集与预测集比例近似为2:1时, 同样建立三种模型, 建模结果如表3。 由表3可知, R2与r2仍均超过0.97, r2较高, 且二者差别小于0.01, RMSE仍均小于0.1 mg· L-1, RMSEP与RMSEC的差距缩小为0.01 mg· L-1。 比较可知, 运用第二种比例的样本划分方法所建模型RMSEP较第一种整体下降, 且R2比第一种整体上升。 因此, 当模型建模集与预测集比例近似为2:1时, 模型的预测效果更好。 除此之外, 由表2和表3对比可知, PLSR的建模效果是3种方式中最好的, 因此, 本文选择PLSR模型进行建模。
通常PLSR模型的LV数量会在很大程度上影响模型精度。 在模型较为理想时, RMSEP一般会随LV个数的增加而降低并达到最小值, 然后随着LV个数的增加反而再升高。 在极少数情况下, LV个数为1时就可以使模型最理想。 图3是在对样本集进行SPXY划分后, 没有经过数据处理的情况下, 所建PLSR模型LV个数为1~10时RMSEP的变化曲线。 当LV个数等于2时, RMSEP最小, 模型最理想。 但是不同PLSR模型理想的LV个数不一定相同, 因此需要针对每一个PLSR模型分别确定其最优LV个数。
2.2.2 样本划分方法比较
选择合适的样本划分方法有助于提升模型的预测性能, 相反如果方法不当, 极有可能无法获得理想的预测效果。 本文使用CG法、 KS算法和SPXY算法三种样本划分方法进行样本集划分。 三种方法如前文介绍, 均有自己的优势。 本文欲根据所采集到数据样本的特征, 选出适合本文的样本集划分方法。 样本总量共88个, 取建模集:预测集≈ 2:1, 即建模集为59个, 预测集为29个。 运用CG法分两种方法取样, 第一种方法是从第2个样本起, 每隔2个样本取样作为预测集。 第二种方法是从第3个样本起, 每隔2个样本取样为预测集。 4种样本集划分统计结果如表4所示。
| 表4 四种样本集划分统计结果 Table 4 Statistics description of sample selection for the aquaculture sample based on four different algorithms |
由表5所建立的6种PLSR模型的预测效果可知, CG法1的预测效果比CG法2好, 两种SR法所建模型的预测效果差别较小, CG法和SR法所建模型的建模效果与预测效果的差别较其他两种方法相对较小, 但这两种方法的随机性较强, 需要经过多次试验才能得到较好的建模效果。 KS法与SPXY法的划分结果是确定的, 相对来说建模效果比较稳定。 KS算法的建模集R2最高, RMSEC最小, 而预测集的预测效果比建模集差。 SPXY算法情况恰好相反, 建模集r2最高, RMSEP最小, 预测集的预测效果较建模集更好。 本文以预测集预测精度的高低作为最终评价模型优劣的指标, 因此选择SPXY为本文的样本集划分方法。
| 表5 基于四种样本划分方法建立的 PLSR模型的预测效果 Table 5 Prediction results of PLSR models based on four different subsets partition algorithms |
本文运用的预处理算法包括小波变换(WT)、 一阶导数(Der1st)、 二阶导数(Der2nd)、 小波-一阶导数(WT-Der1st)以及小波-二阶导数(WT-Der2nd)5种预处理算法。 运用了SPA和SR两种特征波段提取算法, 通过比较运用不同预处理及特征波段提取算法的PLSR模型的预测效果, 以确定最优预处理及特征波段提取算法。 表6综合比较了不同数据处理算法所对应的模型预测效果, 分析可选出最优数据处理方法。
| 表6 基于多种预处理和特征提取方法建立的PLSR模型效果对比 Table 6 PLSR models’ effect of TN concentration based on different pretreatment and feature extraction methods |
由表6可以得出如下7个结论: (1)除了模型4使用Der2nd预处理的模型预测效果较差之外, 其他9个模型的预测效果均较好; (2)模型1, 2, 5和7的决定系数均为最高, 但模型7的RMSEP最低, 由此可知, 预处理算法、 LV变量个数和特征波段提取算法的选择, 对于模型精确度的提升均有影响; (3)由模型1, 2, 3和4对比可知, WT在一定程度上可以改善模型的预测效果, 主要表现在RMSEP的降低, 而单纯的导数预处理则没有使模型的预测效果得到改善; (4)由模型2, 5和6对比可知, WT-Der1st的预处理后的模型预测效果比单一的WT好, WT-Der2nd预处理后的模型预测效果则不如WT好; (5)由模型5和7对比可知, 经过同样的预处理方法, 然后经SPA特征提取后, 模型的预测效果比只经过WT-Der1st预处理后的建模预测效果略好; (6)由模型6和8对比可知, 基于WT-Der2nd预处理, 经SPA特征提取后, 模型的预测效果比只经过预处理后的建模预测效果差; (7)由模型7和9对比, 模型8和10对比可知, 运用 SPA进行特征提取的波段数量小于SR, 其特征提取效率比SR高, 基于WT-Der1st预处理后, 再经过SPA特征波段提取后建模的效果比SR好, 基于WT-Der2nd预处理后, 再经过SPA特征波段提取后建模的效果却不如SR好。
综上表明, 本文以模型预测精度尽可能高且模型复杂度尽可能低为模型选择原则, SPA特征提取算法能够大大地简化模型。 因此选择模型7作为最优模型, 即WT-Der1st预处理方法作为最优预处理方法, SPA作为最优特征提取方法。
通过对比不同预处理方法、 不同特征提取方法所建立的模型预测效果, 最终通过比较10个模型的r2与RMSEP, 发现基于小波-一阶导数-连续投影变换的PLSR模型的预测效果与运行效率是最好的。 首先通过小波变换对原始光谱曲线进行噪声去除, 使得曲线变得更加平滑, 其次再通过求一阶导数, 可以消减光谱数据的基线漂移, 提高数据质量等。 然后通过SPA进行特征提取, 提取到的特征波段是218 nm, 该特征波段与查阅文献所获知的总氮的吸收峰位置相一致。
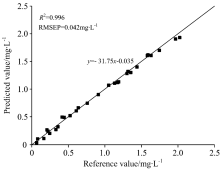
基于上述数据处理方法, 建立了总氮PLSR模型, 所建模型为y=-31.75x-0.035, 该模型的r2为0.996, 模型的RMSEP为0.042 mg· L-1, 图4为总氮预测值和实测值的比较结果, 发现所有数据点均在y=x直线左右分布, 非常靠近直线。 由此可见, 本文所使用的一系列数据处理方法相对科学, 所得模型的预测效果十分理想, 能够满足要求。
使用紫外光谱技术进行总氮含量快速检测方法与传统的化学试剂检测法相比是一种快速、 有效、 简便、 环保的检测方法。 本文大量地应用算法的对比方法, 比较运用不同数据处理方法建立的10个PLSR模型, 得出结论可知, SPXY法是最合适的样本集划分算法, 在此基础上建立的基于WT-Der1st-SPA的总氮PLSR模型的预测效果最好, 其预测结果r2=0.996, RMSEP=0.042 mg· L-1。 相比原始光谱数据的全波段建模效果, 本文所建模型仅使用1个波段, 而RMSEP却降低了8.7%。 由此可见, 光谱预处理算法能够降低原始光谱数据的噪声, 提高数据精确度; 特征波段提取算法与LV变量选择能够解决光谱的数据冗余问题, 简化模型。 由此可见使用紫外-可见光谱技术能够快速准确地预测出总氮含量, 为实现光谱技术在水产养殖其他水质监测指标的在线检测以及快速测定提供了经验。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|