作者简介: 季晓菲, 女, 1986年生, 四川省草原科学研究院助理研究员 e-mail: jixiaofei2012@qq.com
虉草( Phalaris arundinacea L.)为多年生冷季型禾本科高产牧草, 粗蛋白(CP)是评价饲草品质的关键指标, 但目前的化学分析方法存在诸多缺点, 寻求高效、 快速、 准确、 安全的虉草CP测定方法是现代草地畜牧业发展和草原生态恢复急需解决的实际问题。 本研究旨在利用近红外光谱(NIRS)建立虉草CP的定量分析模型, 为快速测定虉草CP提供有效方法。 试验采集不同品种(品系)、 生育期、 栽培条件、 干燥方式、 生长年限、 部位以及刈割次数的虉草样品454份, 采用瑞士Buchi公司的傅里叶近红外光谱仪和Operator软件采集原始光谱, 应用K-S算法剔除具有相似光谱的样品, 筛选出210份用于建模和模型评价。 通过凯氏定氮法测定210份样品的粗蛋白含量并在Management console软件中对光谱进行赋值, 再采用软件NIRcal 5.4按照6∶3的比例将样品随机分为校正集和验证集, 并剔除异常样品, 运用不同的光谱预处理、 回归算法、 建模波段和主成分数建立8个虉草CP含量的近红外定量分析模型, 通过外部验证表明8个模型均可以进行实际测定。 最后比较不同的统计学参数获取最佳模型。 结果表明, 采用4 000~10 000 cm-1的建模光谱波段、 sa3+ncl+db1(3点平滑+趋近归一化+一阶导数处理)的预处理方法、 8/1-4的初/次级主成分数和偏最小二乘法(PLS)所建的模型为最佳模型, 其校正决定系数(
Reed canary grass ( Phalaris arundinacea L.) is a perennial cool-season gramineae grass with a high yield. Crude protein (CP) is a key indicator in the evaluation of forage quality, but the use of chemical analytical methods to determine the CP content is disadvantageous. Therefore, a fast, efficient, accurate, and safe determination method is required in the development of modern grassland agriculture, animal husbandry and grassland ecological restoration. The purpose of this study is to use near-infrared spectroscopy (NIRS) techniques to develop a quantitative model for the analysis of CP in reed canary grass and provide an effective method for a rapid determination. We collected 454 samples of reed canary grass from various resources, including different cultivars (or strains), different growth stages, different cultivation conditions, different drying methods, different growth years, different parts and different harvest times. The original spectra of all of the samples were obtained using a near-infrared spectrometer (NIRFlex N-500) and Operator software of the Swiss Buchi company. A total of 210 samples were selected for the development and evaluation of models after deleting samples with similar spectra by a K-S algorithm, and were assayed using the Kjeldahl nitrogen method to obtain the chemical values of CP; we then assigned them to spectra in a Management console software. The samples were randomly divided into a calibration set and a validation set according to the proportion of 6∶3, using the NIRcal 5.4 software; the outliers were then eliminated. We established 8 quantitative analysis models for the CP content of reed canary grass by applying different spectral data pretreatments, primary/secondary principal components, spectral regions, and regression algorithms. We revealed that all of the 8 models can be used in the determination of CP by performing an external validation. The best model was chosen by comparing statistic parameters. The results showed that the best calibration model was developed by the spectral data pretreatment of sa3+ncl+db1 (smoothing average 3 points+ normalization by closure+first derivative BCAP), choosing the primary/secondary principal component of 8/1-4 and spectral region of 4 000~10 000 cm-1 in combination with the partial least square (PLS) regression algorithm. Its calibration coefficient of determination (
虉草(Phalaris arundinacea L.)为禾本科虉草属多年生冷季型高产牧草, 具有良好的适应性和优异的生产性能被广泛用于人工草地建设、 草地生态保护、 草地改良等, 对草牧业发展和生态建设都起着重要作用[1]。 粗蛋白(crude protein, CP)是衡量饲草品质的关键指标, 虉草粗蛋白受物候期、 发育水平、 基因型、 栽培措施等诸多因素影响差异较大[2]。 但目前常用的化学测定方法存在耗时、 费力、 成本高、 步骤繁琐、 污染环境等缺点[3], 因此寻求快速、 高效、 准确、 安全的虉草粗蛋白测定方法, 成为现代草牧业发展和草原生态恢复急需解决的实际问题。
采用近红外光谱(NIRS)测定CP含量已成为农作物及饲草品质研究的重要手段[4, 5], 具有操作方便、 检测效率高、 成本低、 安全无污染、 稳定性高和样品制备简单等优点。 目前, 已应用在牧草领域, NIRS已在紫花苜蓿、 燕麦、 羊草、 老芒麦以及天然牧草等粗蛋白含量测定, 但尚未见用于分析虉草粗蛋白的报道。 本工作在国内首次建立了虉草粗蛋白含量的近红外定量分析模型, 为快速测定虉草粗蛋白提供有效方法, 为虉草品质分析、 育种、 家畜日粮配置以及草产品流通提供技术支撑。
2015年9月至10月和2016年4月至11月采集不同品种(品系)、 生育期(分蘖期、 拔节期、 孕穗期、 抽穗期、 开花期、 灌浆期、 乳熟期、 蜡熟期、 完熟期、 枯黄期)、 栽培条件(8种行距、 7种施肥梯度)、 干燥方式(自然晾干、 65 ℃烘干、 杀青后65 ℃烘干)、 生长年限(1~6年)、 部位(茎、 叶、 穗和全株)以及不同刈割次数的虉草样品454份。 除不同干燥方式的样品按要求干燥然后65℃烘干至恒重外, 其余样品采集后直接经105 ℃杀青后65 ℃烘干至恒重, 再采用高速粉碎机粉碎, 过40目筛后装袋, 放于避光处备用。
采用瑞士Buchi公司的傅里叶近红外光谱仪(NIRFlex N-500)和附带软件Operator采集样品光谱, 谱区范围4 000~10 000 cm-1, 扫描次数32, 分辨率8 cm-1, 扫描间隔4 cm-1。 在Petri培养皿中装入粉状样品, 保证表面平整且装样量约在培养皿的3/4处, 重复扫描3次。
应用Kennard-Stone算法(Matlab 7.01)剔除具有相似光谱的样品[6], 挑选210份用于建模和模型评价。 按照GB/T 6432-94凯氏定氮法测定所选样品的粗蛋白含量, 每个样品重复测定3次取平均值。
在软件Management console(瑞士Buchi公司)中将测定的化学值赋值到光谱, 再采用软件NIRcal 5.4将样品光谱按照6∶ 3的比例随机分为校正集(Calibration-set)和验证集(Validation-set), 以保证同一样品的三条光谱在同一样品集。 粗蛋白含量的最大、 最小值划归校正集, 以避免模型外推。 接着运用不同的回归算法、 光谱预处理方法、 建模波段和主成分数建立校正模型, 并对光谱残差、 属性残差进行计算以剔除异常样品。 最后通过外部验证对模型的实际预测能力进行评价, 采用决定系数(R2)、 校正标准差(SEC)、 验证标准差(SEP)、 残差(BIAS)、 相对分析误差(RPD)[6]等统计学参数验证模型效果和筛选最佳模型。
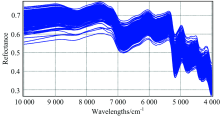
454份虉草样品的原始光谱如图1所示, 光谱曲线趋势一致, 说明样品中无干扰杂质或干扰杂质的影响可忽略, 而不同样品表现出不同的反射率, 其差异说明各样品成分含量不同, 光谱数据符合建立近红外定量分析模型的要求。
从所选的样品中剔除了6个异常值, 共204份样品, 其化学测定结果如表1所示。 虉草样品粗蛋白含量变幅为2.70%~29.08%。 陈光吉等[8]测得不同生育期变幅为7.77%~10.83%; 张永亮等[2]报道分蘖期、 抽穗期、 开花期和结实期的CP分别为23.8%, 13.6%, 9.6%, 5.1%; 贾有余等[9]研究表明不同栽培条件下的CP变幅为3.05%~20.64%。 骆秀梅等[10]发现不同行距与施氮量下虉草各器官CP变幅为5.61%~25.69%。 相比之下, 本试验用于建模的粗蛋白变幅更大, 具有广泛的适用性, 同时校正集和验证集的最大值、 平均值、 最小值和标准差都非常接近, 表明校正集和验证集的分组合理, 满足建立近红外分析模型的基础。
| 表1 校正集和验证集虉草样品的粗蛋白化学分析结果(%DM) Table 1 CP of Phalaris arundinacea samples used in calibration and validation sets (%DM) |
表2为采用软件NIRcal 5.4通过不同的预处理方法、 建模波段、 回归算法和主成分数组合得到的CP定标模型。 试验所建立的8个定标模型的校正决定系数(
| 表2 建立的8个粗蛋白近红外定标模型的条件及对应结果 Table 2 Conditions of establishing NIRS models and results of 8 calibrations for CP |
建立校正模型后, 通过外部验证对模型的实际预测能力进行评价, 验证结果见表3。 验证决定系数(
| 表3 虉草粗蛋白含量的外部验证参数 Table 3 Parameters of external validation for CP of Phalaris arundinacea samples |
根据
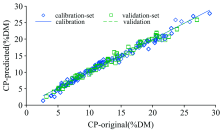 | 图2 校正集与验证集化学测量值与预测值的相关性Fig.2 Correlation between original and predicted values of CP in calibration and validation sets |
由于近红外光谱技术是一种间接分析技术, 其检测准确性与样品的代表性、 化学分析误差、 样品状态、 装样条件、 光谱预处理方法等诸多因素相关。 本研究采集了不同品种(系)、 部位、 栽培条件、 干燥方式、 生育期、 生长年限以及刈割次数的虉草样品454份, 样品来源广泛, 所测粗蛋白含量变幅为2.70%~29.08%, 包含了目前科研与生产上可能出现的含量水平, 使建立的虉草粗蛋白模型能够满足实际生产和科研的要求。 同时化学测定采用国家标准凯氏定氮法, 并三次重复测定, 能够有效减少化学分析误差。 在样品含水量、 样品温度、 样品粒度和装样条件上都采用统一干燥标准、 重复装样的方法来消除干扰。 并且通过比较不同的光谱预处理、 回归算法、 建模波段和主成分数建立了较优的8个模型, 再通过统计学评价参数综合分析选出最优模型, 保证了建模质量。
目前国内外尚未见报道近红外在虉草品质分析上的应用, 国内学者对其他饲草的粗蛋白近红外模型已有研究, 严旭等[6]以110份样品建立的老芒麦粗蛋白近红外定量模型的
因此, 本研究结果表明虉草CP模型预测效果良好, 可用于科研和生产的实际分析。 为综合评价虉草的营养价值后期将尝试建立虉草的其他品质指标模型。 同时, 为了在生产中广泛应用NIRS模型, 将进一步在实际应用中添加不同年份的样品数据, 对方程进行纠正和优化[13, 14], 以提高模型的适应性和预测能力。
虉草粗蛋白最优模型为采用4 000~10 000 cm-1的光谱波段、 sa3+ncl+db1的预处理方法、 8/1-4的初/次级主成分和偏最小二乘法PLS所建的模型, 其
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|