作者简介: 于云华, 1969年生, 中国石油大学信息与控制工程学院教授 e-mail: yuyunhua@upc.edu.cn
单倍体育种技术是玉米育种新方法, 该方法可有效缩短产生纯合系的周期, 提高育种效率。 该技术需首先挑选足量单倍体籽粒, 而玉米在未加人工干预时, 单倍体在混合籽粒中仅占0.05%~0.1%, 即使采用生物诱导技术, 单倍体籽粒数一般也不到籽粒总数的10%。 高速、 精准地从大量混合籽粒中挑选得到占比少于10%的单倍体籽粒, 才能够满足工程化育种需要, 而实际育种工作中挑选单倍体时常用的分子生物学、 田间形态学辨别等方法存在耗时长、 成本高、 破坏样本等缺点, 难以高效精准地得到玉米单倍体籽粒。 相关研究已经证明高油玉米的单倍体与二倍体之间具有明显含油率差异, 目前低场核磁共振技术可用于检测玉米单籽粒的含油率, 并根据含油率对单倍体进行鉴别, 但核磁共振仪存在价格贵、 维护难、 速度慢、 效率低等弱点, 现有设备完成单籽粒分选需用时4 s, 无法满足工程化育种中大量筛选的速度需求。 使用VIAVI微型近红外光谱仪能够达到0.25 s每颗的检测速度, 相比核磁共振技术速度快, 仪器价格较低, 维护方便。 使用近红外光谱仪分析技术对单倍体与二倍体籽粒进行鉴别, 可以取代核磁共振鉴别单倍体的方法。 采用近红外光谱定性鉴别单倍体籽粒虽然取得了一定效果, 但目前研究中所采集玉米品种相对较少, 研究只针对某一品种单倍体建立模型, 对该品种单倍体进行分类; 国内外尚无多品种混合单倍体鉴别相关研究, 而工程化育种亟需一种能够识别多个品种玉米单倍体的鉴别方法。 为此, 本文提出一种基于深度信念网络的多品种混合玉米籽粒单倍体鉴别方法, DBN是一种多层深度神经网络, 每层由受限玻尔兹曼机构成, 采用逐层训练策略, 可解决传统神经网络训练方法不适用于多层网络训练的问题。 对比实验结果表明使用DBN方法建立多品种单倍体鉴别模型具有较高分类性能, 能够满足玉米工程化育种精度要求。
Haploid breeding technology is a new method for maize breeding, which can effectively shorten the cycle of homozygous lines and improve the breeding efficiency. The technology needs to select enough haploid grains first, and the haploid grains only account for 0.05%~0.1% of the mixed grains without artificial intervention. Even with the biological induction technology, the number of haploid grains is generally less than 10%. High-speed and accurate identification of haploid grains can meet the!needs of engineering breeding. However, molecular biology and morphological identification methods commonly used in practical work are time-consuming, costly and destroying samples. It is difficult to select Maize Haploid grains efficiently and accurately. Relevant studies have proved that there are obviousoil content differences between haploid and diploid of high-oil maize. At present, low-field nuclear magnetic resonance technology can be used to detect oil content of maize and identify haploid according to its oil content. However, nuclear magnetic resonance (NMR) instrument has some weaknesses, such as high price, difficult maintenance, slow speed and low efficiency. It takes 4 seconds for each single-grain sorting. It cannot meet the needs of large number identification for engineering breeding. Using VIAVI near infrared spectrometer (NIRS) can achieve the detection speed of 0.25 seconds for each maize. The NIR technology is faster, cheaper and easier to maintain. The NIR identification method can replace the method of NMR. Qualitative identification of haploid by NIRS has achieved some results, but currently there are relatively few maize varieties collected in the study. The study only establishes models for haploid of one variety, and classifies haploid of that variety. There are no studies on identification of multiple hybrid haploids at home and abroad, but engineering breeding urgently needs a method to identify multiple varieties of maize haploids. In this paper, a method for identifying haploids based on deep belief network is proposed. DBN is a multi-layer deep neural network. Each layer is composed of a restricted Boltzmann mechanism. By using layer-by-layer training strategy, the problem that traditional neural network training methods are not suitable for multi-layer training can be solved. The comparative experimental results show that the identification model of multiple varietieshaploid established by DBN method has high classification performance and can meet the requirements of maize engineering breeding accuracy.
针对单倍体玉米籽粒快速分选问题[1, 2, 3, 4, 5, 6, 7], 采用近红外定性分析鉴别玉米单倍体籽粒虽然取得了一定效果, 但目前研究中所采集玉米品种相对较少, 研究只针对某一个品种的单倍体二倍体建立模型, 对该品种的单倍体二倍体进行分类[8]。
覃鸿等采用了微型近红外光谱仪对玉米单倍体近红外方法定性鉴别时, 漫反射与漫透射两种采集方式对识别效果影响进行了比较研究, 实验中分别在两种方式下采集了单一品种的玉米籽粒的近红外光谱, 并使用支持向量机(support vector machina, SVM)的二分类方法鉴别单倍体, 漫透射方式下分类器能够获得高于90%的分类准确率, 而漫反射方式的识别率显著低于漫透射方式, 已不能满足单倍体鉴别的精度要求, 研究认为漫透射方式比漫反射方式更适合玉米单倍体籽粒的鉴别[9]; 覃鸿等研究了在使用MicroNIR1700型微型近红外光谱仪漫透射方式下采集光谱时, 光源强度以及光阑孔径对单倍体识别效果的影响, 并通过实验确定了单倍体识别率最优时的光源功率及光阑孔径[10]。 上述研究为采用近红外光谱漫透射方式鉴别单倍体打下了重要基础。
针对单个品种单倍体鉴别模型存在通用性不足的问题, 本研究提出一种基于深度信念网络的多品种混合的玉米籽粒单倍体鉴别方法, 首先采用多层受限玻尔兹曼机无监督学习及BP神经网络训练得到多品种单倍体籽粒光谱的深度信念网络模型, 然后对各个品种的单倍体二倍体籽粒进行分类识别, 实验结果表明与其他几种常规分类方法相比, 使用深度信念网络方法建立的多品种单倍体鉴别模型具有较高的分类性能, 实验中所用10个品种的单倍体识别率均能达到90%以上, 为实现玉米工程化育种提供了新的方法。
Geoffey Hinton教授在2006年提出了一种基于概率生成模型的深度信念网络(deep believe net, DBN)。 DBN是一种多层深度神经网络结构, 每层由受限玻尔兹曼机(restricted Boltzmann machine, RBM)构成, DBN采用逐层训练策略, 可解决传统神经网络训练方法不适用于多层网络训练的问题, 整个DBN训练过程分为预训练与调优两个阶段[13, 14]。
对于近红外光谱定性分析问题, 深度信念网络以获取原始输入信息深层特征为最终目标, 通过逐层无监督训练策略, 避免传统多层神经网络在使用独立梯度下降法时陷入局部极小值的问题。
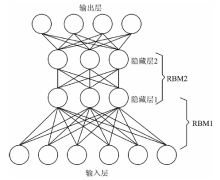
DBN网络结构如图1所示, 最下层为输入层, 最上层为输出层。 该网络中包含两层RBM, RBM是一种两层随机神经网络, 本质是使得学习所得模型能够产生符合条件的样本, 并使得该样本的概率最大。 RBM包含1个可见层和1个隐含层, 其特点是同层节点之间并无权值连接, 因此同层中每个节点之间概率独立分布。
对于一组给定的状态(v, h), 可定义如下能量函数
式(1)中, vj是可见层, hi是隐藏层, wij是前层与后层的连接权重, aj是可见层的偏置, bi是隐藏层偏置。
RBM中的调整参数是θ =(w, a, b), 给定训练样本后, 训练RBM意味着调整参数θ , 并拟合给定的训练样本。
状态(v, h)联合概率分布为式(2)
配分函数
通过梯度下降算法计算网络参数, 其中, ln(P(v|θ ))关于参数θ 的偏导数如式(3)所示
式中, < · > 表示分布P的数学期望。 P(h|v(t), θ )表示条件为v(t), θ 的概率分布。 P(v, h|θ )表示可见单元与隐单元的联合分布。
RBM可由如下几个步骤的对比散度方法并逐层训练得到:
(1)设定模型的参数初值θ 0=(w0, a0, b0), 并设定程序结束的条件。
(2)使v0等于输入的向量。
(3)计算h0, v1, h1, 其激活概率计算如式(4)和式(5)
其中, 激活函数σ (x)=
式(6)— 式(8)中, α 为学习率, < · > data为训练样本集所定义的分布上的数学期望。 < · > recon为重构后的模型所定义的分布上的数学期望。
(4)对模型参数进行更新。
(5)若满足程序结束条件, 则训练结束, 否则将以θ t-1赋值, 并转步骤(3)。
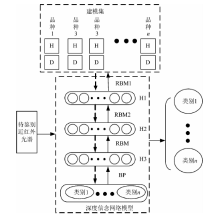
算法设计以玉米单倍体(Haploid, H)、 二倍体(Diploid, D)[1, 2, 3, 4, 5, 6, 7]近红外光谱为例, 建模集中每个品种玉米光谱都分为单倍体与二倍体两类, 对于本节玉米单倍体二倍体籽粒的分类任务, 深度信念网络输入为125维近红外光谱, 而输出为近红外光谱所对应的单倍体二倍体类别标签。
基于DBN的近红外光谱定性分析模型如图2所示, 首先选择足量近红外光谱作为建模集, 其后使用多层受限玻尔兹曼机与BP算法训练得到深度信念网络定性分析模型, 最后将待鉴别近红外光谱代入模型即可判定其类别。
基于DBN的近红外光谱定性分析模型网络首先采用逐层贪心算法训练模型参数, 其后在上一步参数初值基础上, 使用BP神经网络[14]对整个网络所有节点进行参数优化, 并进行全局训练, 最终得到基于DBN的近红外光谱定性分析模型。
实验使用国家玉米改良中心提供的10个经过高油诱导的玉米品种, 二倍体籽粒胚部带有明显紫色记号, 而单倍体胚部没有紫色标记。 单倍体占比约为3%6%, 实验所用的玉米籽粒样本于2015年在北京某郊区实验基地种植并收获得到, 每个品种在田间预选足量的备选籽粒, 再通过有经验的技工根据R1-nj颜色标记从各个品种中挑选得到单倍体与二倍体籽粒[11]。 其中, 单倍体与二倍体籽粒分别为100粒, 10个品种的单倍体籽粒以及二倍体籽粒各1 000粒作为实验研究对象。 为保证实验结果可靠性与准确性, 在人工挑选阶段, 只选择颜色标记非常清晰的个体, 舍弃颜色标记模糊、 属性难以分辨、 籽粒形状畸变及胚部发育不良的籽粒。
为叙述方便, 10个品种的单倍体、 二倍体混合籽粒的近红外光谱数据分别命名为K1, K2, …, K10数据集, 经过光谱仪自带软件预处理后得到125维光谱向量, 后期数据分析处理使用Matlab2016a。
采用支持向量机描述光谱质量判定方法剔除异常光谱, 预处理方法如平滑、 求导等操作去除光谱噪声、 提升光谱信噪比, 为下一步实验做好数据准备。
针对上述10个品种的2000粒玉米单倍体二倍体混合籽粒, 使用SVM, BPR和DBN三种方法分别实验。
对比实验方法设置如下:
SVM: 特征提取环节采用偏最小二乘(partial least squares, PLS)与正交线性判别分析(orthogonal linear discriminant analysis, OLDA)方法, 原始光谱降维后为4维[14], 使用LIBSVM工具箱, 设置SVM分类器类型为二分类类型, 以最优识别率为标准, 在高斯核参数σ 及正则化参数C指数增长的过程中, 通过多重交叉验证以网格的方式搜索最优高斯核参数σ 及正则化参数C, 其中高斯核参数σ =3.2, 正则化参数C=0.56。
BPR: 特征提取环节采用PLS与OLDA方法, 原始光谱降维后为4维, 使用样本选择算法(kennard-stone, KS)方法选择构网样本点, 超香肠作为基本覆盖单元, 通过交叉验证确定超香肠神经元的半径[14]。
实验1
三种定性分析方法对比实验。 在各个品种的数据集独立实验时, 随机抽取各个品种数据的二分之一作为训练集, 共100粒(训练集中单倍体二倍体数目相等各50粒), 剩余二分之一作为测试集测试得到识别率, 共重复10次取平均值。
表1— 表3是分别使用SVM, 仿生模式识别(biomimetic pattern recognition, BPR), DBN三种方法对各个品种分别建立模型所得的正确识别率与正确拒识率。
| 表1 SVM方法各品种单倍体鉴别准确率(%)(各品种独立建模) Table 2 SVM: Recognition rate (%) (Independent modeling) |
| 表2 BPR方法各品种单倍体鉴别准确率(%)(各品种独立建模) Table 2 BPR: Recognition rate (%) (Independent modeling) |
| 表3 DBN方法各品种单倍体鉴别准确率(%)(各品种独立建模) Table 3 DBN: Recognition rate (%) (Independent modeling) |
由表1— 表3可知, 三种方法对于K1, K3, K5和K8等四个品种鉴别准确率均出现了低于85%的情况。 其中, K5和K8均低于80%。 说明单独使用这四个品种的样本集, 进行建模训练, 测试本品种数据时, 所得识别效果低于其他六个品种, 该四个品种单倍体与二倍体之间差异信息小于其他六个品种差异信息。 以上现象说明在近红外光谱单倍体鉴别中, 同样的机器学习方法对于不同品种建立模型识别效果并不一致。
实验2
三种定性分析方法在各个品种的数据集上独立识别与多个品种混合建模识别对比实验。
品种独立实验: 随机抽取各个品种数据的二分之一作为训练集, 共100粒(训练集中单倍体二倍体数目相等各50粒), 剩余二分之一作为测试集测试, 得到鉴别准确率, 重复20次取平均。
多品种联合实验: 2 000粒玉米籽粒的光谱数据构成混合品种实验数据集, 从混合品种实验数据集中随机抽取各个品种数据的二分之一作为训练集(训练集中单倍体二倍体数目相等各占一半), 剩余二分之一作为测试集测试, 得到鉴别准确率, 重复20次取平均。
表4— 表6为使用SVM, BPR, DBN三种建模方法建立定性分析模型所得鉴别准确率。
| 表4 SVM方法各品种单倍体鉴别准确率(%)(混合建模) Table 4 SVM: Recognition rate (%) (Mixed modeling) |
| 表5 BPR方法各品种单倍体鉴别准确率(%)(混合建模) Table 5 BPR method: Recognition rate (%) (Mixed modeling) |
| 表6 DBN方法各品种单倍体鉴别准确率(%)(混合建模) Table 6 DBN method: Recognition rate(%) (Mixed modeling) |
各表第一行为各品种样本分别单独建模并识别各自品种测试集样本的预测结果。 各表第二行为共10个品种的玉米籽粒光谱混合来建立模型对单倍体二倍体进行分类, 并单独统计各个品种识别率所得识别结果。
从表4— 表6可见, 使用K1— K10共10个品种玉米籽粒联合建模后, 统计得到各品种识别率相对于对各品种单独建模单独测试各个品种识别率均有所提升。
以鉴别准确率较低的几个品种为例, 三种定性分析模型性能提升有如下差异:
(1)使用SVM方法时, K1品种鉴别准确率提升了1.8%, K3品种鉴别准确率提升了1.4%, K5品种鉴别准确率提升了3.1%, K8品种提升了3.2%。
(2)使用BPR方法时, K1品种提升了1.9%, K3品种提升了2.1%, K5品种提升了3.1%, K8品种提升了3.2%。
(3)使用DBN方法时, K1品种提升了6.9%, K3品种提升了4.9%, K5品种提升了10.5%, K8品种提升了12.3%。
由表4— 表6可得: 使用品种混合后, 单个品种独立建模识别效果较差的品种的识别率都具有上升趋势, 三种方法中以DBN方法识别率上升最为明显, 识别率上升也与对比实验中品种混合的数据集相对于单个品种独立建模时的数据量增大相关。 在建模集相同的情况下, 在多个品种混合实验中, 在DBN方法获得了最优的分类效果, BPR获得了次优的分类效果。
上述结果说明DBN方法在使用多个品种混合籽粒进行训练时, 能够充分发掘不同品种单倍体与二倍体之间共同差异信息, 同时进行多个品种学习时, 能够提升单个品种单倍体二倍体的分类性能, 实现类似于多任务学习中不同分类任务之间的信息共享。
针对玉米单倍体籽粒快速分选问题, 提出了基于深度信念网络多品种的单倍体二倍体混合籽粒定性鉴别方法, 对单个品种数据集独立建模以及多个品种混合建模, 并进行对比实验。 实验结果表明采用同样方法对每个品种单独建立模型时, 识别效果有优有劣, 性能并不一致; 而使用品种混合后进行实验, 10个品种能够达到90%以上的识别率, 在实验所用的3种机器学习方法中, 以DBN方法性能最优, 能够达到类似于多任务学习的目标, 实现品种之间信息共享, 并提高多品种单倍体识别效果。
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|