作者简介: 张 钰, 1992年生, 云南农业大学农学与生物技术学院硕士研究生 E-mail: m15343842322@163.com
由于部分毒菌与野生食用菌形态和生物学特征相似, 农民仅凭经验采集, 难免将两者混淆, 从而导致严重的食品安全事故。 云南省作为国内野生食用菌产量最高、 出口量最大的省份, 野生食用菌产业发展为云南农村经济发展做出了突出贡献, 对不同种类野生食用菌进行快速鉴别, 有利于野生食用菌产业的健康发展; 分析食用菌亲缘关系, 对食用菌育种工作具有积极作用。 七种牛肝菌样品, 采自云南及周边七个产地, 利用FTIR光谱仪分别采集菌柄和菌盖红外指纹图谱, 基于低级与中级数据融合策略, 将预处理后的菌柄和菌盖FTIR光谱数据进行融合, 结合Decision Trees, Discriminant Analysis, Logistic Regression Classifiers, Support Vector Machines, Nearest Neighbor Classifiers和Ensemble Classifiers中的17种算法, 分别建立菌柄、 菌盖、 低级数据融合和中级数据融合模型, 每个分类模型连续进行10次运算, 通过比较训练集分类正确率平均值, 确定牛肝菌种类鉴别最佳分类算法。 中级数据融合数据集进行系统聚类分析(
Many wild nocuous fungi are similar to the edible in morphology and biological characteristic, which easily leads to serious food safety incident because it is difficult for farmers to distinguish them just by experience. The progress of wild edible production makes a great contribution to rural economy of Yunnan province where the yield and export volume are highest in China. Rapid authentication of wild edible fungi variety is beneficial for wild edible industry towards healthy development. Meanwhile, the authentication also contributes to the analysis of the genetic relationship between edible mushroom and their breeding. Seven kinds of fungi were collected from Yunnan and other seven origins around Yunnan. Fingerprint of caps and stipe were obtained with Fourier transforms infrared (
毒蘑菇亦称毒菌, 全世界有1 000种以上, 我国至少有435种, 其中39种为牛肝菌, 约占我国已知牛肝菌种类的11%[1, 2, 3]。 目前, 多数野生牛肝菌无法人工栽培, 主要来源于农民野外采集。 由于许多毒菌与野生食用菌形态和生物学特征相似, 农民仅凭经验采集, 难免将两者混淆, 从而导致严重的食品安全事故。 例如, 2012年四川一次婚宴上, 200多人出现集体食品中毒症状, 研究后发现华丽牛肝菌、 毒牛肝菌和中华牛肝菌三种毒菌的混入是导致此次事故的罪魁祸首[4]。 云南省作为国内野生食用菌产量最高、 出口量最大的省份, 野生食用菌产业发展为云南农村经济发展做出了突出贡献。 因此, 对不同种类野生食用菌进行快速鉴别, 有利于野生食用菌产业的健康发展。 另一方面随着野生食用菌驯化、 栽培等过程, 菌种出现异种同名或同种异名的现象, 以及品种退化、 混杂、 病毒感染等问题[5], 分析食用菌亲缘关系, 对食用菌育种工作具有积极作用。
对遗传背景、 有机成分、 矿质元素等特征性指纹信息差异进行分析, 可用于鉴别不同种类野生食用菌[6, 7]。 经过加工后失去原有形态特征的样品, 凭借传统形态学鉴别很难区分开。 采用交配亲和试验分类[8]、 同工酶分析[9]、 分子生物学鉴别分析等方法[10, 11], 操作繁琐、 检测时间长且费用高等, 难以对大量的商品野生食用菌进行快速鉴别。 傅里叶变换红外光谱(Fourier transform infrared spectrometer, FTIR)鉴别分析技术, 是有机成分指纹分析中常用的一种方法, 具有快速、 经济、 可靠、 简便等特点, 现在已广泛应用于中草药、 食品等领域[12, 13]。
数据融合通过数据获取、 预处理、 特征筛选, 将两个或两个以上光谱数据进行融合, 得到的新数据集进行模型训练, 从不同的信息层面反映样品间差异, 更加全面解释样品属性[14]。 Má rquez等[15]基于数据融合策略将拉曼光谱和近红外光谱融合, 对榛子酱中掺入杏仁酱与鹰嘴豆酱的食品欺诈行为进行鉴别, 结果显示数据融合分类判别结果优于单一信息。 Reis等[16]采用傅里叶变换衰减全反射与漫反射红外光谱数据的融合, 用来鉴别咖啡掺假, 对四种掺假方式进行判别分析, 结果显示, 数据融合模型分类效果优于采用单一数据模型。 以上研究结果表明数据融合较单一信息, 显示更多化学指纹信息差异, 有利于准确的样品表征。
食用菌样品特征性指纹信息不仅受到遗传信息影响, 还可能受产地、 储存年限、 采集年份等多种因素干扰, 为了在复杂的牛肝菌背景信息中, 探讨产地因素对不同种类样品鉴别的影响, 本研究7种牛肝菌样品, 采自云南及周边7个产地, 采用FTIR光谱仪测定牛肝菌的菌柄和菌盖红外指纹图谱。 基于低级与中级数据融合策略, 将预处理后的菌柄和菌
盖FTIR光谱数据进行融合, 结合Decision Trees, Discriminant Analysis, Logistic Regression Classifiers, Support Vector Machines, Nearest Neighbor Classifiers和Ensemble Classifiers中17种算法, 分别建立菌柄、 菌盖、 低级数据融合、 中级数据融合模型。 通过比较分类鉴别的结果, 确定最佳分类模型。 最佳分类数据矩阵进行系统聚类分析(hierarchical cluster analysis, HCA), 探讨不同种类牛肝菌的亲缘关系, 以期为野生食用菌质量控制提供一种有效的新方法。
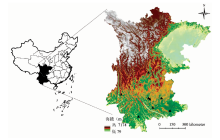
246份七种牛肝菌的不同部位(菌柄、 菌盖)的样品, 于2012年采自云南省及周边七个产地(每个州或市代表一个产地)见图1, 材料来源见表1。
| 表1 不同种牛肝菌材料来源 Table 1 Information of Boletaceae samples with different species |
傅里叶变换红外光谱仪(美国珀金埃尔默公司, 配有DTGS检测器)、 奥豪斯电子分析天平(梅特勒-托多仪器有限公司)、 YP-2型压片机(上海市山岳科学仪器有限公司)、 101A-1型电热鼓风恒温干燥箱(上海市崇明实验仪器厂)、 60目不锈钢筛盘(浙江上虞市道墟五四仪器厂)、 溴化钾(分析纯; 天津市风船化学试剂科技有限公司)。
样品采集后去除杂质, 清水洗净, 50 ℃烘干至恒重。 牛肝菌样品分为菌盖和菌柄两部分粉碎后过60目筛, 分别装于自封袋中避光保存备用。 称取样品粉末(1.5± 0.2) mg及溴化钾粉末(100± 0.2) mg, 在玛瑙研钵中充分混合并研磨成细粉, 放入压片模具压成厚度均匀的薄片。 仪器预热1 h后测定光谱, 光谱扫描范围4 000~400 cm-1, 扫描前扣除压片背景的干扰, 每个样品重复测定3次, 取平均光谱作为样品测量光谱。
Omnic8.2软件对样品FTIR原始光谱进行吸光度转换、 纵坐标归一化处理。 SIMCA-P+13.0对FTIR原始光谱, 进行标准正态变换(standard normal variate, SNV)、 二阶导数(second-derivative, 2D)预处理, Origin8.0软件绘制菌柄、 菌盖FTIR平均光谱图, 以及对FTIR光谱数据进行PLS-DA降维, 提取特征变量。 利用MATLAB2017a软件中Classification Learnertoolbox对数据集进行模型训练。
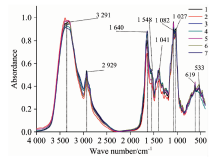
图2为经过吸光度转换、 纵坐标归一化预处理后, 不同种类牛肝菌FTIR平均光谱。 由图可知, 不同种类牛肝菌FTIR光谱在3 291, 2 929, 1 640, 1 548, 1 401, 1 082, 1 027, 619, 533和469 cm-1等附近有明显的特征吸收峰。 3 291 cm-1附近吸收峰主要为多糖和蛋白质中羟基的O— H伸缩振动以及N— H伸缩振动, 2 929 cm-1附近吸收峰主要为多糖、 蛋白质中甲基、 亚甲基伸缩振动, 1 640 cm-1附近主要为芳香环中C=C伸缩振动, 1 548 cm-1附近主要为蛋白质酰胺Ⅱ 带的N— H变形和C=N伸缩振动, 1 401 cm-1附近可能为羧酸根离子中C— O弯曲振动和O— H变形, 1 239 cm-1附近可能为羧酸中的C— O伸缩振动和O— H变形或者是酚类C— O伸缩振动, 1 082 cm-1附近为脂肪族C— OH伸缩振动, 1 082~1 027 cm-1范围内吸收峰为多糖或类多糖C— O伸缩振动, 1 027~619 cm-1附近主要是糖类的异构体[17, 18, 19]。 吸收峰强度的差异, 表明不同种类牛肝菌内蛋白质、 脂肪酸、 多糖、 芳香族类等化学成分含量可能存在差异。
模式识别(pattern recognition)是通过机器处理和分析各种信息, 以对事物进行描述、 解释和分类[20], 其过程为数据获取、 预处理、 特征筛选和分类决策, 现在广泛应用于食品安全监控、 计算机、 生物信息学、 海洋探测、 分析化学等领域[21, 22, 23, 24, 25]。 支持向量机(support vector machine, SVM)由Vapnik于1995年提出, 是一种基于风险最小化与VC维理论建立的机器学习方法, 其优势是解决小样本、 高维数据、 非线性、 局部极小点等问题[26], K-最近邻分类算法(K-nearest neighbor, KNN)是将最邻近的一个样品扩展为K个, 当K个样品中属于哪一类样品较多, 就归为这一类。 决策树(decision tree)从一组无规则的事例推理出决策树表示形式的分类规则, 具有计算量较小、 分类准确率较高等优点。 传统的SVM, KNN, Tree等方法解决二分类问题存在一些局限, 为了克服不足, 对其进行了改进, 例如Linear SVM, Quadratic SVM, Cosine KNN和Cubic KNN等算法。
柄和盖FTIR光谱预处理后, 分别建立柄和盖的独立决策模型。 将归一化后柄和盖FTIR光谱数据串联, 得到新的数据集进行模型训练, 建立低级数据融合模型。 柄和盖FTIR光谱数据进行PLS-DA降维, 筛选特征值大于1的前几个主成分, 分别提取柄和盖模型的前16与前18个主成分得分进行融合, 建立中级数据融合模型。 不同模型的数据集采用Kennard-Stone算法, 选择83个样品作为训练集, 其余40个样品为预测集, Complex Tree, Medium Tree, Simple Tree, Linear Discriminant, Linear SVM, Quadratic SVM, Cubic SVM, Fine Gaussian SVM, Medium Gaussian SVM, Fine KNN, Medium KNN, Cosine KNN, Cubic KNN, Weighted KNN, Bagged Trees, Subspace Discriminant和Subspace KNN算法进行模型训练, 比较训练集正确率(表2), 确定不同模型的最佳分类算法。 结果显示, 菌柄、 菌盖和低级数据融合模型最佳分类算法均为Linear Discriminant, 训练集正确率分别为92.8%, 96.4%和97.6%。 中级数据融合模型为Subspace Discriminant, 训练集正确率为100%。 表明Linear Discriminant与Subspace Discriminant算法能改善模型分类效果。
| 表2 不同分类器的训练集预测结果(%) Table 2 Predicting results of training set with different classifier (%) |
Linear Discriminant与Subspace Discriminant对模型数据集进行运算, 模型随机选择有代表性的数据进行训练, 运算结果可能出现偏差, 需要对数据进行多次运算[27, 28]。 本次研究对菌柄、 菌盖、 低级数据融合和中级数据融合模型进行10次运算, 训练集预测结果混淆矩阵见图3, 横坐标表示样品的真实标签, 纵坐标表示预测标签。 菌柄、 菌盖、 低级数据融合和中级数据融合最佳分类模型, 训练集(
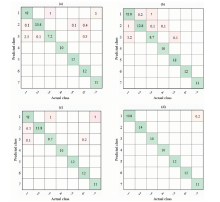 | 图3 训练集预测结果混淆矩阵 (a): 菌柄; (b): 菌盖; (c): 低级数据融合; (d): 中级数据融合Fig.3 The confusion matrix of training set (a): UV-Vis; (b): FTIR; (c): Low-level data fusion; (d): Mid-level data fusion |
| 表3 最佳分类模型预测结果(%) Table 3 Prediction results of the optimal model(%) |
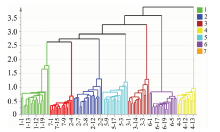
系统聚类分析(hierarchical cluster analysis, HCA)依据样品特征性指纹信息相似程度, 将比较相似样品聚为一类[29]。 采用不同部位FTIR中级数据融合数据进行HCA, 首先对不同部位FTIR光谱中有利于分类的化学信息进行挖掘, 比较化学信息差异, 推测样品亲缘关系。 不同种类牛肝菌中级数据融合HCA见图4, 图中相同颜色的样品代表同一种类, 横坐标为样品编号, 纵坐标为不同种类间临界值的距离, 距离越小, 聚为一类的样品越相似。 聚类分析树状图显示, 当临界值距离为2时, 不同种类牛肝菌样品分类全部正确, 七种牛肝菌样品被分为七组, 第一到七组分别为1号(华丽牛肝菌)、 7号(美味牛肝菌)、 2号(小美牛肝菌)、 5号(栗色牛肝菌)、 3号(砖红绒盖牛肝菌)、 6号(绒柄牛肝菌)和4号(皱盖疣柄牛肝菌)样品, 聚类临界值距离从第一组到第七组逐渐变大, 表明华丽牛肝菌与美味牛肝菌样品化学信息较相似, 这两种牛肝菌亲缘关系可能比较近。 华丽牛肝菌与皱盖疣柄牛肝菌样品化学信息差异最大, 这两种牛肝菌亲缘关系可能较远。
用FTIR光谱仪分别采集七种牛肝菌, 菌柄和菌盖样品红外光谱指纹图谱, 基于低级与中级数据融合策略, 将预处理后的菌柄和菌盖FTIR光谱数据进行融合, 结合Complex Tree, Medium Tree, Simple Tree, Linear Discriminant等17种算法, 分别建立菌柄、 菌盖、 低级数据融合和中级数据融合分类模型。 结果显示, 菌柄、 菌盖和低级数据融合模型最佳分类算法为Linear Discriminant, 中级数据融合模型为Subspace Discriminant。 中级数据融合分类结果优于菌柄、 菌盖和低级数据融合, 表明中级数据模型可以将相似度较高的样品区分开, 且减少了产地对种类鉴别的影响。 HCA结果显示, 华丽牛肝菌和美味牛肝菌聚类临界值距离最小, 表明样品化学信息较相似, 这两种牛肝菌亲缘关系可能比较近, 华丽牛肝菌与皱盖疣柄牛肝菌聚类临界值距离最大, 表明样品化学信息差异最大, 这两种牛肝菌亲缘关系可能比较远。 综上表明, 基于中级融合策略将不同部位FTIR光谱数据融合, 结合Subspace Discriminant与HCA, 可以准确鉴别不同种类牛肝菌和快速推测样品亲缘关系, 可作为野生食用菌种类鉴别和推测亲缘关系的一种新方法。
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|