作者简介: 李丹丹, 1990年生, 中国科学院理化技术研究所博士研究生 e-mail: lddgem@mail.ipc.ac.cn
纳米金刚石(NDs), 作为一种具备良好生物兼容性、 化学稳定性、 药物负载能力和众多不可比拟优越性能的材料, 其在生物医学领域的应用被广泛关注, 尤其是在生物成像和抗癌药物传输领域。 首先对不同尺寸纳米金刚石的拉曼性能进行评价, 确定了100 nm高温高压合成的NDs更适宜作为拉曼生物探针。 之后, 为了生物领域的应用, 这些NDs表面的杂质经过羧基化方式处理获得均一表面性能, 并采用扫描电镜、 红外、 拉曼和粒径分析手段对该过程进行验证。 然后, NDs作为拉曼探针被用于快速定位HeLa细胞内NDs的分布, 验证了HepG2细胞对NDs内吞过程的时间依赖性。 此外, 借助非侵入性的三维(3D)共聚焦拉曼成像技术, 可视化观察了四种不同细胞(HeLa, HepG2, C6和MDCK)对NDs内吞量和滞留量的差异。 其中, MDCK这种正常细胞内部极少发现NDs, 而其他三种癌细胞中有大量NDs信号, 显示出不同种类细胞对于NDs的吞入和滞留量的明显差异。 实验结果表明, 纳米金刚石拉曼生物探针不仅可以用于生物成像, 更为癌症的定位和诊断提供可能性。
Nanodiamonds, with good bio-compatibility, chemical stability, drug accommodatingability, fluorescence signals and many other superior properties, have attracted growing attention for anti-cancer drug delivery and bio-imaging applications in recent decades. In this work, the intrinsic Raman signal (1 332 cm-1) of nanodiamonds (NDs) with different sizes synthesized by high temperature high pressure (HTHP) and explosion methods were investigated and compared, in order to select suitable NDs as Raman probes for bio-application, indicating that HTHP synthesized NDs with the size of 100 nm were preferable. NDs were surface homogenized to remove the impurity for further bio-application through acid carboxylation, which was verified with Scanning Electron Microscopy (SEM), Frourier Transform Infared Spectroscopy (FTIR), Raman Spectroscopy and size analysis. And the cytotoxicity of NDs to different cell lines was confirmed with Cell Counting Kit-8 (CCK-8) assay, assuring the good bio-compatibility of NDs and their potential application in bio-system. Besides, based on the sharp Raman peak of NDs at 1 332 cm-1 and the representative Raman vibration band of cells in 2 800~3 000 cm-1 range, NDs were used as Raman bio-probes for the fast localization of NDs in HeLa cells, and the non-invasive dual-color two-dimensional (2D) confocal Raman imaging technique with 532 nm laser was successfully realized. Furthermore, the uptake process of NDs into HepG2 cells was visualized with Raman mapping, revealing the time-dependent behavior of this internalization process. Besides, the endocytosis and localization of NDs as Raman probes by different cells, including HeLa, HepG2, C6 and MDCK cells, were further investigated using the advanced three-dimensional (3D) confocal Raman imaging microscopy, which could render detailed information about the interaction of NDs and cells. And it was observed that the endocytosis of NDs was influenced by cell lines, not only for cancer and non-cancer cells, but also for different cancer cell lines, providing further potential applications for bio-imagingand cancer diagnosis. These results were encouraging and sufficient for following bio-medical studies.
近年来, 纳米材料在生物成像、 抗癌药物运输和癌症治疗领域有非常突出的表现, 得到大量关注[1, 2]。 通常, 肿瘤部位的血管孔隙率高, 缺乏淋巴引流, 具备高通透性和滞留性(enhanced permeability and retention, EPR), 这使得纳米材料更趋向于聚集在多数实体瘤中, 为纳米材料在癌症治疗领域的应用提供便利[3]。 目前, 在众多被广泛关注的纳米材料中, 纳米金刚石(nanodiamonds, NDs)在生物医学方面的应用成为新的研究热点。 NDs有非常多的优越性能, 比如化学性质稳定、 生物兼容性好、 表面可修饰性强以及具备载药能力等, 这为NDs在生物医药领域的应用提供基础, 且已有大量相关工作被报道[4, 5, 6, 7, 8, 9]。 这些工作中, NDs不仅能作为药物载体, 同时也可以作为标记物或生物探针以确定NDs载药体系在生物体内的位置。 NDs作为标记物或生物探针, 在生物成像领域的优越表现也备受瞩目。 通常而言, NDs依赖其荧光特性, 作为荧光探针被广泛应用在多种生物医学和成像领域, 这种传统的荧光成像方式存在许多问题, 例如, 通常需要对细胞进行固定和染色处理, 操作流程复杂; 特别需要注意的是, 细胞生物体系常有自发荧光, 还可能存在光漂白等。 这些问题限制了NDs荧光探针的应用。 但是, 对于NDs作为拉曼光谱探针就不存在上述问题。 NDs自发的特征拉曼信号(位于1 332 cm-1)清晰而锐利, 而且不会被生物细胞的自发信号(位于2 800~3 200 cm-1)干扰, 更不存在光漂白和背景荧光干扰等影响因素[10]。 因此, 以NDs为拉曼探针, 借助非侵入性的拉曼技术手段, 实现细胞生物领域的快速成像, 为NDs在细胞内的定位、 其与细胞间相互作用的可视化观察以及其他生命活动过程的观察提供极大的便利[11, 12]。 不过, 这些工作普遍集中在NDs拉曼探针在生物体系中的二维(2D)成像, 其成像方式由于技术手段受限, 仅能给出单一方向的拉曼图像, 导致数据量不足, 信息不全面等问题。 近年来, 随着整个拉曼显微镜的发展, 共聚焦三维(3D)拉曼成像技术能给出两个扫描方向(平面扫描和深度扫描)的多层图像, 并对图像中大量数据进行叠加拟合, 为单个细胞内结构细节的可视化和细胞与拉曼探针相对位置的确定提供了可能[13, 14]。 因此, 尝试和探索将3D拉曼成像技术应用在NDs拉曼探针与细胞相互作用及其他过程的可视化领域, 给出三维方向拉曼图像, 不仅可以获得更多、 更详尽完备的数据信息, 更能为进一步分析生物作用过程提供极大便利。
本工作中, 基于纳米金刚石良好的生物兼容性和锐利的特征拉曼信号, 尝试将其作为生物探针, 依赖共聚焦二维拉曼成像技术, 以实现在单个人宫颈癌细胞(HeLa)内快速定位纳米金刚石探针, 并可视化人肝癌细胞(HepG2)内吞纳米金刚石的过程。 进一步, 利用3D拉曼成像技术, 对比HeLa细胞、 HepG2细胞、 大鼠神经胶质瘤细胞(C6)和犬肾细胞(MDCK), 对于NDs吞入量和滞留量的差异, 获得更全面和详尽的信息, 为后续纳米金刚石拉曼探针在生物成像、 定位追踪和癌细胞治疗方面提供研究基础。
为了选取合适的纳米金刚石拉曼生物探针, 实验中对常见的5, 50, 100和200 nm的纳米金刚石(NDs)进行拉曼性能评价。 这些NDs由广州克斯曼有限公司(中国)提供, 其中5 nm的NDs通过爆炸法制备得到, 原始粒径为50, 100和200 nm的NDs通过高温高压合成后破碎获得。 不同粒径纳米金刚石的特征拉曼光谱由显微共聚焦拉曼光谱仪(inVia-Reflex, 英国)获得。 细胞培养过程中用到的细胞培养基(DMEM)和磷酸缓冲液(PBS)购自Hyclone(赛默飞世尔, 中国), 青霉素-链霉素和0.25%的胰酶-EDTA由Hyclone(赛默飞世尔, 美国)提供, 胎牛血清(FBS)购买于Hyclone(赛默飞世尔, 新西兰)。 采用购买于同仁化学研究所(东京, 日本)的细胞增殖试剂盒(CCK-8)对NDs就不同种类细胞的生物兼容性进行评价。 其他使用到的试剂均购买于中国医疗集团(上海, 中国), 属于优级纯, 均未经进一步纯化, 直接使用。
为了获得具有均一表面的NDs, 以用于细胞体系, 首先将纳米金刚石进行羧基化修饰。 本实验主要通过强酸氧化以实现表面羧基化和均一化, 即: 将NDs置于浓硫酸和浓硝酸(体积比3:1)的混合液中, 70 ℃下加热反应24 h, 然后除去混合酸, 将处理后的纳米金刚石置于0.1 mol· L-1的NaOH溶液中, 在70 ℃下加热1 h, 再用HCl溶液中和。 之后, 上述处理后的纳米金刚石被多次离心清洗, 最终在空气中干燥, 获得羧基化后均一的纳米金刚石(carboxylated NDs), 以备实验使用。 纳米金刚石的表面羧基化通过红外测试(FTIR, Excalibur 3100, Varian, 美国)验证, 其粒径分布通过动态光散射仪(Malvern Instruments, 英国)测试得到, 特征拉曼光谱由显微共聚焦拉曼光谱仪(inVia-Reflex, 英国)获得。
采用10, 25, 50及100 μ g· mL-1四种不同浓度的NDs溶液, 对试验中选取的人宫颈癌细胞(HeLa)、 人肝癌细胞(HepG2)、 大鼠神经胶质瘤细胞(C6)和犬肾细胞(MDCK)共四种不同细胞的生物兼容性进行评价。 四种细胞分别置于96孔板中, 用含有青霉素-链霉素和血清的培养基进行培养至细胞汇合到50%~60%, 之后三次清洗, 换用含有10, 25, 50及 100 μ g· mL-1四种不同浓度NDs的新鲜培养基, 分别培养24 h。 之后细胞活性通过CCK-8试剂盒, 在450 nm波长下测试获得。
首先, 人肝癌细胞(HepG2)细胞被PBS清洗三次, 然后用含有50 μ g· mL-1 NDs的培养基分别处理2, 4, 6和8 h, 再用PBS清洗三次后用于拉曼光谱观察。 二维(2D)拉曼成像通过双通道共聚焦拉曼显微镜(Raman-11, Nanophoton, Osaka, 日本)实现, 观察时采用60X/1.2的水镜(UPLSAPO60XW, Olympus), 激发波长用532 nm, 激光功率为0.7 mW· μ m-2, 扫描方式为线扫描, 获得一张2D拉曼图像需要5~8 min。 通过截面扫描(cross-section)和深度扫描(depth)对NDs处理不同时间的HeLa细胞进行观测, 可观察细胞对NDs生物拉曼探针的内吞过程。 实验重复三次, 每次选取5~8个细胞进行成像分析。
对人宫颈癌细胞(HeLa)、 人肝癌细胞(HepG2)、 大鼠神经胶质瘤细胞(C6)和犬肾细胞(MDCK)共四种不同细胞对NDs内吞效果的差异进行可视化研究。 首先, 四种细胞分别被PBS清洗三次, 然后用含有25 μ g· mL-1 NDs的培养基分别处理4 h, 再用PBS清洗三次, 之后用新鲜培养基再培养48 h, 再用PBS清洗用于拉曼观察。 同上述2D拉曼成像一致, 三维(3D)拉曼成像也通过双通道共聚焦拉曼显微镜实现, 观察时采用60X/1.2的水镜, 激发波长用532 nm, 激光功率为0.7 mW· μ m-2, 扫描方式为线扫描, 获得一张拉曼图像需要5~8 min。 按照z轴方向, 从高到低, 将细胞厚度分成5~8份, 每隔1~2 μ m进行一次平面扫描, 之后利用Image J软件对获得的多层图像进行叠加拟合, 获得不同种类的3D拉曼图像。 这样, 3D拉曼成像技术成功用于定性评价不同细胞对NDs内吞量的差异。 实验重复三次, 每次选取5~8个细胞进行成像分析。
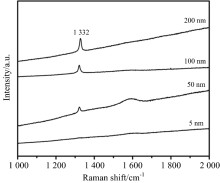
为了获得合适的纳米金刚石拉曼生物探针, 我们对四种不同粒径的纳米金刚石进行拉曼分析(图1), 以确定实验中选取的纳米金刚石生物探针具备清晰且不被干扰的拉曼信号。 对于初始粒径为5 nm的NDs, 拉曼信号非常微弱, 几乎观察不到; 50 nm的NDs样品, 出现两个较弱的特征拉曼峰, 即: 位于1 590 cm-1的G峰和1 310 cm-1的D峰; 而对于100~200 nm的NDs样品, 出现位于1 332 cm-1的尖锐的金刚石信号。 通常而言, 非金刚石相的结晶金刚石颗粒具备单晶核; 而50 nm以上的单晶金刚石, 通常内部具有完好金刚石结构, 表面有石墨层。 图1中观察到, NDs原始粒径大于100 nm, 出现尖锐和高强度的金刚石振动峰, 而原始粒径为5~50 nm的NDs, 其拉曼信号较弱, 主要来自于石墨和不定形碳结构。 而作为拉曼生物探针的NDs必须具备清晰的高强度且锐利的拉曼信号, 因此, 我们选取100~200 nm的NDs作为生物探针。 此外, 为了便于NDs在生物体和细胞内的内吞、 代谢等活动, 以及考虑到NDs之后可能作为药物载体, NDs粒径不宜过大, 因此, 本工作中选取原始粒径为100 nm的NDs作为拉曼生物探针。
直接购买获得的100 nm的纳米金刚石(NDs)表面通常有多种基团, 如羧基、 氨基和羟基等, 这些基团的存在, 可能会影响NDs在生物体系中的应用, 因此需要获得表面均一的NDs。 按照经典纳米金刚石羧基化方法, 如实验部分描述, 对样品进行强羧基化(COOH)修饰处理, 得到表面含均匀COOH基团的羧基化纳米金刚石(carboxylated NDs)。 通过SEM、 粒径分析、 FTIR和Raman等手段对纳米金刚石和羧基化后纳米金刚石的形貌、 尺寸、 表面基团及拉曼信号进行对比分析。 如图2中可以看到, 图2(a)中SEM对比表明羧基化前后, 纳米金刚石的单粒粒径均在100 nm左右; 图2(b)中粒径分布测试表明羧基化前后的纳米金刚石粒径分布均一, 都在100 nm左右, 表明羧基化修饰不会改变纳米金刚石尺寸; 图2(c)中FTIR测试对比表明, 羧基化修饰后, NDs表面的羧基特征基团红外振动强度增加, 验证了羧基化的顺利实现; 图2(d)中拉曼光谱分析表明, 羧基化修饰前后, NDs的特征拉曼峰的位置都在1 332 cm-1, 且清晰锐利, 峰强度几乎不受羧基化影响。 以上结果表明: NDs表面羧基化修饰过程对NDs本身的性能几乎没有影响, 也为NDs作为拉曼生物探针提供便利条件。
另外, 为了确定NDs在生物细胞体系中的应用潜力, 实验中对表面均一化处理后的100 nm NDs的生物兼容性进行评价(图3)。 选取四种不同浓度的NDs溶液, 即10, 25, 50及100 μ g· mL-1, 分别对四种常见的细胞, 即HeLa细胞, HepG2细胞, C6细胞和MDCK细胞处理24 h, 然后测试细胞活性。 图3中结果表明: 不同浓度的NDs溶液对不同种类细胞的细胞活性几乎没有影响, 从而证明NDs具备良好的生物兼容性, 进而验证其作为拉曼生物探针的可行性。
作为一种非侵入性的观察手段, 共聚焦拉曼成像技术被广泛讨论、 应用和研究。 本工作中, 以NDs作为拉曼生物探针, 依靠拉曼成像技术, 实现了对NDs与细胞相对位置的可视化观察。 图4中给出经过100 μ g· mL-1NDs处理2h的单个HeLa细胞的普通显微镜结果[图4(a)], 和对应于图4(a)中细胞上三个红点的拉曼信号[图4(b)]。 如图4(b)所示, 谱线1中位于1 332 cm-1处尖锐的拉曼信号, 来源于NDs中的伸缩振动, 该信号指示NDs的存在。 而谱线2中位于2 800~3 000 cm-1处的拉曼信号归属于细胞结构中CH2和CH3基团的对称性伸缩振动[15], 该信号表明细胞的位置。 值得注意的是, 细胞在该位置的拉曼信号几乎不会随着细胞种类的变化产生明显差异。 谱线3为同时出现的NDs信号和细胞信号, 表明细胞与NDs在该位置的共存。 这些结果表明: NDs锐利清晰的信号, 不会被细胞内多种物质的信号干扰, 从而进一步证明了NDs作为拉曼探针所具备得不可比拟的独特优势。
 | 图4 单个HeLa细胞的普通显微镜下观测结果(a)和其中标记红点的拉曼光谱(b)(比例尺: 10 μ m)Fig.4 Bright field microscopic image of a HeLa cell (a) and Raman spectra (b) of red spots in (a) (scale bar: 10 μ m) |
通过对上述NDs和细胞特征拉曼信号的空间强度分布, 进行双通道谱图拟合, 可以得到二维(2D)拉曼成像中的图像。 图5给出上述HeLa细胞的双通道共聚焦2D拉曼扫描图。 其中, 图5(a)和(b)分别表示平面扫描(XY)和深度扫描(XZ)系列图像; 图5(a1)和(b1)为平面扫描和深度扫描方向的示意图; 图5(a2)和(b2)为HeLa细胞分布的拉曼扫描图, 该成像通道呈红色; 图5(a3)和(b3)为NDs分布的拉曼扫描图, 该成像通道呈绿色; 图5(a4)和(b4)为上述HeLa细胞和NDs分布的叠加拉曼图, 可以明显观察到, 在目前的实验条件下, NDs不仅聚集在细胞周围, 而且能够进入细胞内部, 分散在细胞质内。 因此, 以NDs作为拉曼生物探针, 基于2D拉曼成像技术, 可以实现对NDs与细胞相对位置的可视化观测。
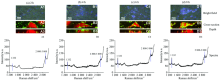
为了可视化观测细胞对NDs的内吞过程, 我们将HepG2细胞由50 μ g· mL-1的NDs分别处理2, 4, 6和8 h, 并分别获得了这些细胞的2D拉曼图和对应细胞内特定位置(黑点)的拉曼光谱(图6)。 如图6(a)所示, 经过2 h的作用, NDs主要分布在HepG2细胞的外围, 而细胞内几乎没有检测到NDs位于1 332 cm-1的拉曼信号, 这表明2 h的作用时间太短, 不足以使NDs大量进入HepG2细胞内。 而经过4 h的相互作用, HepG2细胞内出现较明显的NDs信号[图6(b)], 但NDs依然主要聚集在细胞表面[图6(b3)]。 当时间延长到6 h, 细胞内的NDs拉曼信号强度明显增强[图6(c4)], 深度扫描图显示细胞内NDs分布在细胞内[图6(c3)]。 图6(d)中可以看到, 经过8 h的NDs处理, HepG2细胞的细胞质内充满大量NDs, 而拉曼光谱信号强度也很高[图6(d4)]。 这部分实验表明, 依赖NDs为探针的2D拉曼成像技术, 可以观察HepG2细胞对NDs随时间变化的内吞过程。 但是, 对后续内吞过程的极值确定和外排作用的观察, 还不完备, 需要进一步系统的研究工作。
如前所述, 2D拉曼成像技术实现了对细胞与纳米金刚石探针相互作用过程的观察。 为了进一步分析细胞与纳米金刚石间的相互作用, 获得更多详尽的细节信息, 我们采用三维(3D)拉曼成像技术以观察NDs在HepG2细胞的分布特征。 这样的观测手段可以实现对单个HepG2细胞的成像, 由于NDs在HepG2细胞的分布不均匀, 随着细胞位置改变, 拉曼光谱强度变化能明显被观测到。 图7所示的HepG2细胞首先被50 μ g· mL-1的NDs溶液处理4 h, 再在新鲜培养基中进行48 h培养。 图7(a)给出3D拉曼成像时对单个细胞的多层扫描示意图, 图7(b)中分别是单个HepG2细胞的普通显微镜下图、 拉曼平面扫描图像和拉曼深度扫描图像。 图7(c)中为上述HepG2细胞的多层拉曼扫描图像, 通过将HepG2细胞按z轴方向平均分成8份, 从上到下每隔1 μ m进行一次平面扫描获得。 图7(d)中的拉曼光谱对应于图7(c)中白色小方框, 位于1 332 cm-1处的拉曼特征峰指示NDs的存在, 而2 800~3 200 cm-1范围内的拉曼峰归属于细胞结构内的C— H振动。 从图7(c)和(d)可以观察到, 随着对细胞从顶部扫描到底部, 纳米金刚石的拉曼信号先增强, 后减弱, 而且可观测到的拉曼信号仅存在于近细胞表面位置, 这表明该位置的纳米金刚石聚集体主要存在于细胞表面或近表面位置; 而该HepG2细胞内其他位置的纳米金刚石, 也随着细胞深度变化出现图像信号强弱差异, 这些纳米金刚石聚集体主要存在于细胞质内, 而且富集在接近细胞膜的位置[图7(c)]。
 | 图7 单个HepG2细胞的3D拉曼扫描示意图(比例尺: 10 μ m)Fig.7 Schematic of 3D Raman imagings for a single HepG2 cell (scale bar: 10 μ m) |
更进一步, 用Image J软件将图7(c)中的多层平面扫描图叠加拟合而成, 获得了HepG2细胞的3D拉曼重构图, 并且可以获得前面、 侧面和反面等三个不同方向的3D拉曼图像(图8)。 依赖三维拉曼双通道成像技术, 图8中不仅可以给出单独HepG2细胞的立体结构, 呈现有一定厚度的三角形形貌(图8 cell), 而且能看到单独NDs在三维空间内的分布特点(图8 NDs)。 更进一步, 通过对两成像通道的叠加(图8 merged), NDs在细胞内的聚集情况和分散规律可以被清晰观察到, NDs在细胞表面有少量聚集, 在细胞内分布不均匀, 主要出现在细胞质内靠近细胞膜的位置。 需要特别关注的一点是, 通过3D拉曼重构图中的反面图像, 为NDs与HepG2细胞的相对位置的观察提供了新的角度, 这样弥补了单纯正面扫描图像的不足, 进一步验证拉曼观察的可靠性并扩展了其适用性。 这样, 纳米金刚石探针与细胞的相对位置和相互作用过程, 可以通过3D拉曼成像技术获得更多、 更详尽的信息, 这为之后的深入研究提供基础。
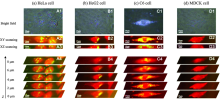
实验中选取四种不同种类细胞, 其中, 有两种常见人体癌细胞, 人宫颈癌细胞(HeLa)和人肝癌细胞(HepG2); 一种鼠癌细胞, 大鼠神经胶质瘤细胞(C6); 以及一种动物正常细胞, 犬肾细胞(MDCK)。 这些细胞分别由25 μ g· mL-1的NDs处理4 h后, 用PBS清洗细胞三次以除去未反应的NDs, 再在新鲜培养基中进行48 h恢复处理, 更便于观察研究NDs在不同细胞内的内吞量和滞留效果差异。 上述四种细胞的单个3D拉曼成像图展示在图9中, 图9(a)中为HeLa细胞, 图9(b)中为HepG2细胞, 图9(c)为C6细胞, 而图9(d)为MDCK 细胞。 其中, 每一种细胞给出单个细胞的普通显微镜下白光图[图9(a1), (b1), (c1)和(d1)], 双通道拉曼平面扫描图[图9(a2), (b2), (c2)和(d2)]和深度扫描图[图9(a3), (b3), (c3)和(d3)], 以及沿z轴方向的多层拉曼扫描图[图9(a4), (b4), (c4)和(d4)]。
如图9中可以观察到, 不同细胞系对于NDs的内吞和滞留效果形成鲜明对比。 首先, 差异源自于癌细胞与正常细胞, 对于MDCK这种正常细胞, 细胞内NDs探针的信号非常微弱, 几乎看不到NDs团聚体, 只在个别位置看到极少数的NDs的绿色斑点; 而对于另外三种癌细胞, HeLa细胞、 HepG2细胞和C6细胞, 能明显观测到NDs的绿色团聚体大量分布在细胞内部。 癌细胞与正常细胞对于NDs内吞量和滞留量有明显差异, 主要是因为癌细胞相较于正常细胞, 其细胞膜通透性增强, 细胞生命活动加速, 对具有高表面活性的纳米金刚石的内吞量比正常细胞大。 因此, 作为纳米粒子的NDs更容易被上述三种癌细胞吸收滞留, 而对于正常细胞, NDs的信号就非常微弱。 这一实验结果表明纳米金刚石作为拉曼生物探针可以用于区分正常细胞和癌细胞, 进一步在癌症识别和诊断领域的应用提供可能性[16]。 而对于C6这种大鼠神经胶质瘤细胞, 在图9(c)中明显观察到, 该细胞的细胞质几乎被NDs团聚体填充满, 展示出明亮的NDs拉曼探针的绿色信号。 而另外两种癌细胞, HeLa细胞和HepG2细胞中的NDs信号相对较弱。 这表明, NDs的内吞和滞留量的差异与癌细胞的种类密切相关, 对于不同种类的癌细胞, NDs的摄入量和其在细胞内的富集状态存在明显差异。 纳米金刚石在更多不同癌细胞系中的内吞量差异、 分布特征及有关影响因素和机理有待后续工作进一步分析和研究。
选取原始粒径为100 nm的NDs经过表面羧基化修饰后, 得到均一稳定、 具备清晰锐利拉曼特征峰的NDs生物探针。 实验证明, NDs作为具备良好生物兼容性的拉曼生物探针, 可以用于二维和三维拉曼成像, 实现单细胞水平快速定位, 可视化观测细胞对NDs的内吞过程。 通过观察不同种类细胞对NDs滞留效果的差异, 发现NDs进入和滞留在细胞内的量的差异, 与细胞种类密切相关, 癌细胞对于NDs的内吞效果明显高于正常细胞。 这些结果为纳米金刚石拉曼生物探针在生物成像、 癌症识别和诊断等领域提供广阔的应用前景。
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|