作者简介: 李 伟, 1988年生, 中国农业大学农学院, 国家玉米改良中心博士 e-mail: wellion@cau.edu.cn
在玉米单倍体技术中, 单倍体鉴别是非常重要的环节。 该研究对大量玉米单倍体与杂合二倍体的近红外透射光谱进行分析, 以期建立一套在生产上实用的单倍体鉴别模型。 通过采集三组遗传背景不同的玉米单倍体与杂合二倍体籽粒光谱, 进行不同机器学习算法对比, 光谱预处理建模效果比较, 以及分析数据集大小对模型构建的影响。 对比所有单倍体与杂合二倍体的平均光谱, 发现二者在光谱的吸收峰位置基本相同, 但是单倍体的吸光度略高于杂合二倍体, 尤其是在波长940~1 120 nm以及1 180~1 316 nm这两段谱区差异较大。 在构建的几个模型中, 采用偏最小二乘法和神经网络算法的模型单倍体鉴别准确率较高, 分别为93.26%和95.42%。 测试集验证的结果与模型准确率一致, 表明两种算法适宜进行单倍体大规模筛选。 利用偏最小二乘法模型比较了不同光谱预处理方法的模型效果, 发现仅进行移动窗口平滑预处理原始光谱进行建模准确率最高。 对不同大小数据集的建模效果对比发现, 在一定范围内增大数据集有助于提高模型准确率。 而且数据中单倍体所占比例较高时, 单倍体预测召回率可达100%。 此外, 还根据籽粒颜色标记挑选出不易鉴别的单倍体和杂合二倍体, 利用偏最小二乘法构建的机器学习模型预测准确率可达93.39%, 显示出近红外鉴别单倍体的优势, 即有可能在不依赖籽粒颜色的情况下实现准确鉴别。 基于机器学习的近红外单倍体鉴别方法具有较高的准确率, 而且该方法还能在后期数据增加的基础上不断优化, 对其开展理论研究有望为自动化智能鉴别单倍体创造条件。
Haploid ide.pngication is a very important part of doubled haploid technology in maize. In this reasearch, we studied the near-infrared transmission spectra of a large number seeds of haploids and heterozygous diploids to establish an accurate model for haploid ide.pngication. Compared with the average spectrum of all haploids and heterozygous diploids, it was found that the absorption peak position of the two spectra was almost the same, but the haploid absorbance was slightly higher than that of heterozygous diploid, especially at the wavelengths of 940~1 120 and 1 180~1 316 nm which shared larger differences. Based on the near infrared spectra of haploids and heterozygous diploids from three different sourcegermplasm, different machine learning algorithms were called to construct a haploid selection model, accuracy of models developed with different spectral preprocessing methods were compared, and the effects of datasets to model evaluation were also studied. By comparison with several models, the haploid ide.pngication accuracy of the partial least squares method and the neural network algorithm reached a high accuracy of 95.42% and 93.26% respectively. The results of the testing set were consistent with the accuracy of the model, indicating that the two algorithms are suitable for large-scale screening of haploids. By using the partial least squares model, the accuracy of the model developed from the spectral preprocessing methods of smoothing was the best. Compared with the modeling results of different data size, it was found that increasing the data set in a certain range could improve the accuracy of the model. And when proportion of haploids was high enough, the recall rate of haploid prediction would reach up to 100%. In addition, haploids and heterozygous diploids which was difficult to be ide.pngied by R1- nj color were selected to form a new dataset. The accuracy of the partial least squares method trained by this dataset was 93.39%. This showed the advantages of NIR machine learning method for haploid ide.pngication, which could be used to achieve accurate ide.pngication in the case independent of R1- nj color expression. The method of NIR haploid ide.pngication based on machine learning has high accuracy and efficiency, and the method can be optimized with increasing data. This research paved a way for the intelligent ide.pngication of haploid.
玉米单倍体技术目前已经在生产上得到广泛应用。 随着生产规模的不断扩大, 单倍体挑选的方法也在不断改进。 1966年, Nanda和Chase[1]将R1-nj标记导入诱导系Stock6, 使籽粒顶部糊粉层和胚盾片同时具有显性颜色标记。 通过颜色标记在籽粒中的表达, 方便了单倍体的早期鉴别。 2003年, 陈绍江等[2]发现利用高油型诱导系为父本与普通玉米杂交后, 母本杂交当代籽粒油分显著高于单倍体籽粒, 利用这一方法筛选单倍体准确率可达90%以上。 据此提出基于油分花粉直感效应鉴别单倍体的理论。 德国的Melchinger等[3]也在研究中证实了这一理论的有效性。 刘金等[4]基于玉米油分花粉直感效应的理论, 开发出一套通过油分自动化鉴别单倍体的系统, 实现了单倍体鉴别从人工到自动的跨越式发展。 陈绍江[5]提出利用系统科学理论和工程化思想推进我国育种学科创新及研发体系, 为单倍体技术的进一步改进提供了思路。
近红外光谱分析技术在玉米杂交种纯度鉴定和单倍体诱导后亚正常籽粒的判别中取得了很好的效果。 2009年, 陈绍江等[6]提出可以利用近红外光谱分析的方法进行玉米单倍体鉴别。 采用近红外光谱结合化学计量学的方法对玉米单倍体进行定性分析, 能够更加准确的反映单倍体与杂合二倍体在物理特性上的区别, 对于开发自动化的近红外光谱仪器实现快速准确鉴别单倍体也具有重要现实意义。 Jones等[7]通过对三个玉米杂交种的单倍体与杂合二倍体的近红外光谱进行分析, 发现除P1-102外其余品种的单倍体均能实现100%准确鉴别。 然而该方法仅利用三个玉米杂交种开展实验, 对于是否适宜更多不同玉米材料的鉴别有待进一步验证, 而且近红外仪器的性能也需要提升。 美国JDSU公司生产的微型近红外光谱仪MicroNIR1700扫描一个籽粒的时间可以缩短至1 s, 在籽粒检测速度上有着很大的优势, 为高通量鉴别单倍体提供了可能。
机器学习作为人工智能的一个重要分支, 在互联网搜索、 垃圾邮件处理、 自然语言识别和产品推荐等方面发挥了重要的作用。 本研究试图通过基于有监督的机器学习分类算法对大量单倍体和杂合二倍体的近红外光谱数据进行分析, 通过对单倍体分类特征的学习, 建立模型应用到大规模单倍体挑选中。 机器学习的优势在于后续的生产应用中, 可以将新的数据再次整合到模型中, 使模型不断优化达到智能挑选单倍体的目的。
将诱导系与不同来源的玉米材料进行杂交, 通过R1-nj颜色标记进行筛选, 得到拟单倍体与杂合二倍体籽粒。 实验所用到的玉米种子根据来源不同可分为三类数据集: (1)诱导玉米DH系(来自于齐319× 昌7-2)产生的拟单倍体和杂合二倍体籽粒, 此部分数据集记为D1。 (2)由DH系和Mo17组配的F1和自交系间相互杂交组配F1诱导产生的拟单倍体, 此部分数据集记为D2。 (3)5个玉米商业杂交种的拟单倍体和杂合二倍体籽粒, 此部分数据集记为D3。 杂交种分别为京科968、 屯玉4911、 屯玉188、 屯玉88、 屯玉168, 由北京屯玉种业有限责任公司提供。 所有的单倍体与二倍体组成的数据集命名为A。
对其中来源于DH系的部分拟单倍体和杂合二倍体籽粒按颜色清晰度进行分级, 选择不易挑选的籽粒组成数据集D4。 挑选出D1数据集中拟单倍体数和杂合二倍体数相同的数据和D3组成新的数据集D5。
采集光谱时将玉米籽粒逐一编号, 并放置于10× 10的冷冻盒中, 便于保存。 用JDSU公司的MicroNIR1700型微型近红外光谱仪对每个籽粒进行透射光扫描, 获得透射光光谱。 扫描过程中籽粒胚面朝向光源。 光谱仪采用真空钨丝灯作为光源, 光源距样品3 cm, 采用线性渐变滤光片(LVF), 光谱范围950~1 600 nm, 分辨率12.5 nm, 其他参数设定参考文献[8]。
本研究采用移动窗口平均(平滑)、 一阶差分求导和矢量归一化等方法对原始光谱进行预处理。 预处理的方式包括平滑, 平滑+求导, 平滑+矢量归一化以及平滑+求导+矢量归一化。 其中平滑窗口宽度为9。
采集完光谱以后, 将拟单倍体种子播种到田间进行真实性鉴定。 采取单粒播种的方式, 并对每一粒种子在田间的位置做好标记, 与光谱测量时的编号做好对应。 在玉米植株拔节期先进行一次初步鉴定。 为保证单倍体正常生长, 需去掉拟单倍体中的杂合二倍体植株(杂株)。 去杂标准为: 植株生长旺盛, 株型舒展且植株高大。 大喇叭口期进行第二次去杂, 保证大部分杂株都去掉。 将拟单倍体中去除的杂株编号记下, 并将其计入杂合二倍体类型。
常用的算法包括基于多元线性回归的偏最小二乘法、 基于先验概率的朴素贝叶斯、 基于逻辑判断的决策树以及基于几何特征的K近邻(距离)和支持向量机(平面)。 本研究在R软件(用于统计计算和数据分析的开源软件)中通过caret包[9, 10]可以调用这几种常用的算法并构建出单倍体籽粒分类模型。 在该模型中, 将单倍体设定为正样本, 可以实现单倍体鉴别。
为限制模型过拟合的出现, 采取10折交叉验证(cross-validation)的方法进行模型构建, 取10次模型的平均值作为最终模型。
采用等比例抽样的方法抽取部分数据作为训练数据, 利用机器学习算法构建模型。 构建好的模型使用剩余数据作为测试集, 验证模型效果。 将测试集数据代入到模型中即可进行预测, 生成混淆矩阵(表1)对比预测值与真实值的关系。 利用准确率(accuracy, ACC)、 精确率(precision)、 召回率(recall)和F1分数评估机器学习模型预测效果。 准确率是指总样本中将单倍体和杂合二倍体都预测为真的样本所占比例。 精确率指模型预测为单倍体中真正的单倍体所占比例。 召回率反映单倍体被正确分类的比例。 F1分数用以衡量二分类模型精确度, 可以看作是模型准确率和召回率的一种加权平均, 其取值范围为0~1。
准确率=(TP+TN)/(TP+FP+FN+TN)
精确率=TP/(TP+FP)
召回率=TP/(TP+FN)
F1=2TP/(2TP+FP+FN)
| 表1 单倍体鉴别混淆矩阵 Table 1 Confusion Matrix of haploid ide.pngication |
通过田间鉴定发现, 出苗的拟单倍体有3402株, 含杂合二倍体190株, 占拟单倍体总数的3.4%。 未出苗单倍体2 246株, 考虑到单倍体生长势弱的特点, 将未出苗的拟单倍体作为单倍体统计。 对不同数据集拟单倍体与杂合二倍体验证结果如表2所示。 以D2数据集为例, 该数据集中所用种子全部为根据R1-nj颜色标记挑选出的拟单倍体。 经过田间验证后, 在2 100粒拟单倍体中有94粒(4.5%)是杂合二倍体, 说明利用颜色标记进行单倍体挑选存在一定错误率。 通过肉眼观察颜色挑选单倍体发生错误的原因主要有以下两个方面, 一是颜色标记在单倍体籽粒中会受到抑制基因的调控而表达不清晰, 二是人工挑选会存在视觉疲劳而导致误判。 在构建单倍体鉴别模型时, 这些田间鉴定为杂株的数据加到杂合二倍体中进行单倍体鉴别将有助于提高准确率。 与前人研究结果相比, 本研究通过利用田间鉴定过真实性的单倍体与杂合二倍体进行单倍体鉴别模型的构建具有更高的可靠性。
| 表2 不同数据集拟单倍体与杂合二倍体田间验证结果 Table 2 Validation of putative haploids and crossed seeds in the field |
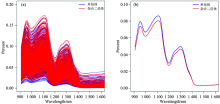
利用微型光谱仪扫描之后总共得到6 597条光谱数据, 分析发现得到的光谱中存在异常值, 通过处理之后得到单倍体光谱为5 445条, 杂合二倍体光谱为1 123条[图1(a)]。 光谱异常值提出的原则为: 计算该样本点到所有样本点中心的曼哈顿距离di, i=1, 2, 3, …, N; 和s为d1, d2, d3, …, dN的均值和标准差; 如果di> 3s, 则该样本为需要剔除的异常样本。 实际在进行大量数据分析时, 可以允许少量数据的异常。 因为数据量极大的时候, 异常的数据并不会对最终的结果造成太大的影响。 对比去除和不去除异常值所建模型准确率, 发现二者差异不大, 也证明了大数据情况下对少量异常值的容忍。 为减小测量过程中的误差, 在近红外光谱扫描过程中每隔半小时进行一次参比, 对仪器进行校正。 实际应用时光谱采集过程中一些轻微的震动也会导致测量结果的误差, 这些系统误差不可避免。
图1(b)对比了所有单倍体与杂合二倍体的平均光谱。 二者在光谱的吸收峰位置基本相同, 但是单倍体的吸光度略高于杂合二倍体, 尤其是在波长940~1 120及1 180~1 316 nm这两段谱区差异较大。 这一实验结果与Jones等[7]利用平均光谱对比单倍体与杂合二倍体的结果一致。 本研究所用单倍体由高油诱导系诱导产生。 根据花粉直感理论, 杂合二倍体油分高于单倍体油分。 同时杂合二倍体在胚部盾片为紫色, 而单倍体胚部盾片无色。 胚部油分多少和紫色物质有无可能是单倍体与杂合二倍体在近红外谱区吸光度差异的主要来源。 因此推测本研究中发现的两个差异较大的谱区可能与盾片颜色和油分有关。
为研究不同机器学习算法在近红外鉴别单倍体方面的差异。 使用单倍体与杂合二倍体数目相同的均衡数据集D5对几种常用的机器学习算法进行评估。 在R软件中的caret包分别调用朴素贝叶斯(Naive Bayes, NB)、 K近邻(K-nearest neighbors, KNN)、 随机森林(random forest, RF)、 梯度推进机(gradient boosting machine, GBM)、 支持向量机(support vector machine, SVM)、 偏最小二乘法(partial least square, PLS)、 神经网络(neutral network, NNet)算法, 构建出单倍体鉴别模型。 在这些算法中, 除偏最小二乘法是线性判别方法外, 其他算法均为非线性。 对比几种算法的准确率(图2), 我们发现神经网络算法构建处理的模型准确率最高, 可达95.42%, 偏最小二乘法次之, 为93.26%。 而朴素贝叶斯和K近邻法模型的准确率最低, 只有50%左右。 对于该结果, 我们认为有两个方面的原因, 一是PLS模型是一种线性模型, 该模型运算耗费内存小且运算量不大, 因此建模需要的时间也就短; 二是PLS模型包含了特征提取的过程, 该模型能够最大程度的提取用于准确分类的特征, 故而可以有较高的模型准确率。 而对于朴素贝叶斯和K近邻法模型都是利用所有的特征进行分类, 这样不仅运算量大, 而且还存在诸多干扰因素, 因此最终模型准确率不高且建模耗费时间长。
 | 图2 不同模型准确率的比较图中显示的是95%置信区间的准确率Fig.2 Accuracy comparison between models95% confidence interval was shown in the figure |
将测试集数据代入模型, 对比真实值与预测值结果如表3所示。 模型预测准确率最高为95.42%(神经网络模型), 其次是93.26%(偏最小二乘法)。 这一结果与模型准确率基本一致, 说明构建的模型不存在过拟合情况。 此外, 随机森林模型的召回率最高, 表明该模型可以将更多的单倍体预测出来(查全)。 而神经网络模型的精确率最高, 说明预测为单倍体的样本中真单倍体比例更高(查准)。 如果综合评估一个算法, 用F1分数则更为准确。 综合来看, 在这几种机器学习算法中, 神经网络模型的F1分数最高, 模型效果最好。
| 表3 不同模型预测效果 Table 3 Comparison between models with different parameters |
通过对不同模型的对比发现, 偏最小二乘法建立的模型准确率高, 而且建模耗费时间短。 因此使用偏最小二乘法对不同预处理的近红外光谱进行建模, 并比较模型预测效果。 光谱预处理方法比较所用数据为所有单倍体与杂合二倍体数据集A。 通过对表4结果分析发现, 基于原始光谱的模型准确率为92.71%。 采用移动窗口平滑和平滑+求导两种光谱预处理方式所得到模型准确率较原始光谱有所提高, 模型准确率均为92.88%。 而其他光谱预处理方法, 得到的模型准确率反而降低。 说明对于鉴别单倍体来讲, 仅进行光谱的平滑处理就足以放大单倍体与杂合二倍体光谱的差异。 而在此基础上进行矢量归一化和求导, 反而会因同时放大噪声而降低模型准确性。 如果仅从单倍体查全的角度来看, 平滑+矢量归一化得到的模型最好(Recall=99.71%)。
| 表4 利用偏最小二乘法对不同预处理的近红外光谱建模 Table 4 Model comparison among different pretreatment methods |
分别对DH系、 DH系杂交组合以及商业杂交种和所有数据组成的4个数据集进行建模比较。 建模所用数据来自对原数据集等比例抽取75%的样本, 模型准确率用ACCm表示, 将原始数据剩余的25%作为训练集检验模型效果, 预测准确率用ACCp表示。 模型评估结果如表5所示。
| 表5 利用偏最小二乘法对不同数据集建模预测 Table 5 Comparison of modelsbased on different datasets |
利用建立的模型对训练集数据进行预测, 仅含有500条光谱的数据集D3的模型准确率要低于样本数较多的数据集D1和D5, 而且来源于数据集D1的子集D5的建模准确率低于D1。 这些结果说明在一定范围内增加数据集规模, 所建模型准确率会相应提高。 这一点可以用机器学习的特性来解释。 鉴别单倍体是一种有监督的机器学习, 需要提供足量的数据用于建模才能建立准确率较高的模型。 对于仅含有拟单倍体数据集D2, 经过鉴定拟单倍体中含有少量杂合二倍体。 利用该数据集建模, 由于数据集中单倍体的数量远大于杂合二倍体, 因此机器学习单倍体的特征足够多, 故在测试集中所有的单倍体都能被鉴别出来。 需要注意的是, 数据集D2中正负样本严重不平衡, 其预测准确率可能不具有实际意义。 在实际生产中, 需要对大量的不同来源的玉米种子进行单倍体挑选。 因此, 我们比较了所有单倍体与杂合二倍体组成的数据集A的准确率。 虽然数据量较D1有所增加, 但是由于数据集中玉米种子的复杂度也同时增加, 导致模型准确率降低。
在对颜色进行分级的籽粒中, 杂合二倍体中发现籽粒胚部(盾片)颜色不清晰的有三个材料, 共30个籽粒。 在拟单倍体中有150个籽粒因胚部隐约有颜色而不能确定其是否为真单倍体。 如图3所示, C1是颜色表达清晰的杂合二倍体, 在盾片上有明显的紫色标记, 而C2— C6由于颜色表达不清晰, 很难确认其是否是真的杂合二倍体。 对应单倍体来说, 盾片无色是容易区分的单倍体(图3 H1), 而H2— H6则由于胚部携带少量的颜色而不易确定单倍体的真实性。 在通过颜色鉴别单倍体的过程中, 由于颜色表达不清晰, 很难通过肉眼挑选实现准确判断。 我们将这些不易判断的单倍体和杂合二倍体在田间鉴定真实性后, 通过机器学习模型预测, 准确率可达93.39%。 对模型进行验证时单倍体漏选率为0(Recall=100%)。 这充分显示出近红外在单倍体鉴别方面的优势, 即对于颜色表达不清晰的杂合二倍体和有颜色干扰的单倍体仍然有较好的识别能力。 与Dong等[11]利用油分鉴别单倍体方法相比, 该方法仅需要进行近红外透射光谱分析, 操作简单, 容易实现。 而油分鉴别单倍体是一种定量分析的方法, 需要对单倍体和杂合二倍体进行油分测定, 选择合适的油分临界值区分单倍体与杂合二倍体。 机器学习近红外鉴别单倍体的方法只需要利用已经建立的模型通过分析扫描的近红外光谱就能对籽粒是否为单倍体进行判断, 该方法操作简单, 更容易实现。
通过有监督机器学习的方式对来源广泛的大量玉米单倍体与杂合二倍体的近红外光谱进行分析, 以实现对大规模玉米单倍体的快速高通量鉴别。 分析单倍体与杂合二倍体的光谱特征, 发现二者在光谱的吸收峰位置基本相同, 但单倍体吸光度大于杂合二倍体。 利用不同机器学习算法对比建模效果, 得到准确率大于90%的两个模型, 分别为基于前馈神经网络算法构建的模型(95.42%)和偏最小二乘法模型(93.26%)。 通过对原始光谱进行不同预处理比较, 发现仅进行平滑处理的近红外光谱建模效果最好, 而其他处理方式得到的模型准确率都低于原始光谱。 利用不同数据集探索了数据集大小对模型准确率的影响。 在一定范围内增加数据集大小和样品多样性将有助于提高模型准确率。 当以单倍体鉴别准确率为考察目标时, 数据集中单倍体所占比例较高, 可实现对原样本中单倍体100%准确预测(无漏选)。 通过对颜色标记难以确定的单倍体与杂合二倍体进行建模预测, 模型准确率可达93.39%, 测试集中单倍体漏选率为0。 通过机器学习的方法所建模型可以实现在颜色表达不清晰的情况下将原样本中的单倍体全部预测出来。
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|