作者简介: 张 钰, 1992年生, 云南农业大学农学与生物技术学院硕士研究生 e-mail: m15343842322@163.com
由于国内外食品市场准入制度和溯源体系不完善, 销售商乱用虚假标签等现象的发生, 使得食品安全形势愈发严峻。 为了保障野生食用菌的安全性, 保护云南高原特色农业品牌战略, 亟需建立快速准确的产地溯源方法。 通过采集云南及其周边8个产地、 79个绒柄牛肝菌子实体的紫外-可见吸收光谱(UV-Vis)与傅里叶变换红外光谱(FTIR), 采用多元散射校正(MSC)、 标准正态变换(SNV)、 二阶导数(2D)、 平滑(SG)等算法对原始光谱进行预处理。 基于低级与中级数据融合策略, 将预处理后的UV-Vis与FTIR光谱信息进行融合, 结合偏最小二乘判别分析(PLS-DA)与支持向量机(SVM), 建立牛肝菌产地鉴别模型, 确定最佳产地溯源方法。 对光谱融合数据进行系统聚类分析(HCA), 探讨不同产地样品整体化学信息的差异性与相关性。 结果显示: (1)采用MSC+2D和SNV+2D对UV-Vis与FTIR光谱进行预处理, R2Y与Q2最大, 分别为61.58%, 95.09%和50.85%, 82.16%, 表明MSC+2D与SNV+2D为UV-Vis与FTIR光谱的最佳预处理方法; (2)基于UV-Vis, FTIR, 低级与中级数据融合建立的PLS-DA与SVM模型, 样品分类错误总数分别为24, 6, 2, 2和6, 1, 1, 0, 表明数据融合模型分类效果优于单一UV-Vis与FTIR模型; (3)中级数据融合模型中, SVM对所有样品的分类全部正确, PLS-DA的分类错误总数为2, 表明基于SVM的中级数据融合策略分类效果优于PLS-DA; (4)低级和中级数据融合HCA模型, 分别有4和1个样品不能与同一类区域样品聚为一类, 表明中级数据融合优于低级数据融合; 由中级数据融合HCA图可知, 同一产地样品聚类距离小于不同产地之间聚类距离, 表明同一产地样品整体化学成分类较相似, 且同一产地不同采集地点的差异小于不同产地之间的差异。 采用UV-Vis与FTIR光谱中级数据融合策略结合SVM, 能够对不同产地来源牛肝菌样品进行准确鉴别, 为野生食用菌产地溯源研究提供一种新方法。
Currently, since the domestic and international food marketing on the safety supervision and traceability system is defective, as well as false labels used by agency, situation on the food safety is becoming more and more serious. In order to enhance food safety, it’s essential to establish a fast and efficient geographical traceability method to protect the agricultural brand of Yunnan plateau. A total of 77 fruit bodies of Boletus tomentipes were collected from 8 geographical origins. Raw of ultraviolet-visible (UV-Vis) and Fourier transform infrared (FTIR) spectra were preprocessed by multiplicative scatter correction (MSC), standard normal variate (SNV), second derivative (2D), Savitzky-Golay (SG) smoothing. Based on pretreatment of UV and FTIR spectra, low-level and mid-level data fusion strategy combined with partial least squares discriminant analysis (PLS-DA) and support vector machine (SVM) were used to identify Boletus in different regions. The results indicated that: (1) that the best pretreatment, was SNV+2D with highest R2 Y (61.58%) and Q2 (95.09%) for UV-Vis spectra, and MSC+2D with highest R2 Y (50.85%) and Q2 (82.16%) for FTIR spectra; (2) For UV-Vis, FTIR spectra, low-level and mid-level data fusion, the number of error samples in the classification of PLS-DA and SVM analysis were 24, 6, 2, 2, and 6, 1, 1, 0, respectively; (3) In the mid-level data fusion, the best classification of SVM with none error sample was better than that of the PLS-DA with 2 error samples; (4) The classification of HCA analysis in the mid-level data fusion with 4 error samples had the better performance than that in the low-level data fusion with 1 error sample. In addition, HCA analysis of mid-level data fusion showed that the distance of samples collected from same area were longer than that collected from different sites. It indicated that the differences of samples collected from different sites in the same area were less than that collected from different regions. Those results indicated that mid-level data fusion combined with SVM model using UV-Vis and FTIR spectroscopy can accurately identify Boletus collected from different geographical origins. It will provide a new strategy on the research of geographical traceability of wild edible fungus.
食品安全是重要的民生问题, 关系到人民身体健康, 社会和谐稳定发展。 近年来, 由于土壤、 大气、 水、 微生物等污染问题日趋严峻, 食品安全引起各国相关政府管理部门的高度关注[1, 2]。 研究表明, 食品原材料品质易受温度、 光照、 海拔等生态因子的影响, 不同产地环境因素的差异会导致食品原材料营养物质的积累差异, 从而影响其品质[3]。 目前, 由于食品污染与品质差异, 越来越多消费者倾向于选择具有地理标志的优质产品[4]。 我国食品市场准入制度不完善, 以及不法商贩受利益驱使乱用虚假标签, 使得污染和冒牌产品流入市场, 因此亟需建立有效的产地溯源体系。 一方面追溯污染物来源, 保证消费者健康[5]; 另一方面为保护地理标志产品提供技术支持[6]。
野生食用菌口味独特, 具有抗氧化、 抗肿瘤、 免疫调节、 抗炎等药用活性[7, 8], 随着人们生活水平的提高和膳食结构的改变, 野生食用菌受到越来越多人的喜爱。 云南独特的地理环境与多样的气候, 孕育了丰富多样的野生食用菌; 全国已知1 000余种野生食用菌中, 云南有900种左右, 其年产量达8万吨, 占全国的70%。 将丰富的野生食用菌资源优势, 转化为产业优势和竞争优势, 符合云南高原特色农业的发展要求[9]。 不同产地野生食用菌生长环境的差异, 导致风味物质和药用成份含量改变, 从而影响其口感与药用功效。 此外, 野生食用菌生长环境不可控制, 加工、 储藏和销售没有统一标准, 可能会造成其重金属、 农残、 微生物等有害成分超标[10, 11]。 被污染或冒牌野生食用菌一旦流入市场, 将损害消费者健康与利益。 为打造云南高原特色野生食用菌产业品牌战略, 需要对其进行系统的科学研究。 因此建立有效的牛肝菌产地溯源体系, 追溯被污染的产品产地, 确定其源头, 再追踪产品流向, 采取相应措施防止事态进一步发展, 达到野生食用菌安全风险防范管控目标, 鉴别是否为地域保护品牌的产地, 保护云南高原特色农业品牌健康发展。
针对食品产地溯源研究, 目前稳定同位素指纹分析[12, 13, 14]、 矿物元素指纹分析[15, 16, 17]、 有机成分指纹分析[18, 19, 20]和分子生物学指纹分析[21, 22]等技术应用广泛。 这些方法采用单一种仪器, 显示的样品化学信息比较单一, 样品分类准确率不高[23]。 而多源光谱数据融合鉴别, 将两个或两个以上光谱数据进行整合, 得到的新数据集进行模型训练, 从不同的层面反映样品间差异, 更加全面解释样品属性, 与采用单一仪器相比具有更好的分类效果[23]。 Spiteri等[24]采用中级数据融合策略, 将核磁共振图谱与高效液相色谱融合, 成功鉴别了不同花粉来源的蜂蜜。 Biancolillo等[25]将五种光谱(热重谱、 中红外光谱、 近红外光谱、 紫外光谱和可见光谱)数据融合, 准确区分了不同产地的啤酒。 傅里叶变换红外光谱(Fourier transform infrared spectrometer, FTIR)和紫外-可见吸收光谱(ultraviolet-visible spectroscopy, UV-Vis)技术, 是有机成分指纹分析技术中常用的两种方法, 具有价格便宜、 简便快速、 准确度高、 灵敏可靠等特点。 FTIR光谱技术通过光谱扫描, 使样品分子中基团或化学键吸收能量产生振动, 形成振动吸收光谱用于鉴别分析[26], UV-Vis技术通过紫外光谱扫描, 样品中化合物在紫外-可见光区吸收光子后, 其价电子在分子的电子能级之间跃迁, 而产生的分子吸收光谱[27]。
目前基于数据融合策略对野生食用菌进行产地溯源的研究较少, 为进一步完善野生食用菌产地溯源体系, 本研究采集云南及周边8个产地、 79个绒柄牛肝菌(Boletus tomentipes)子实体的UV-Vis与FTIR光谱, 数据预处理后, UV-Vis, FTIR, 低级数据融合与中级数据融合, 结合偏最小二乘判别分析(partial least squares discriminant analysis, PLS-DA)、 支持向量机(support vector machine, SVM), 分别建立判别模型, 并通过比较模型分类正确率, 确定快速甄别绒柄牛肝菌地域来源的方法。 对数据融合进行系统聚类分析(hierarchical cluster analysis, HCA), 比较低级和中级数据融合模型聚类效果, 探讨同一区域不同采集地点样品关系, 以期为野生食用菌质量控制体系提供一种有效的新方法。
8个产地(每个州或市代表一个产地)、 10个不同采集地点的绒柄牛肝菌(B. tomentipes)样品, 于2011年采集(表1), 均由云南农业大学刘鸿高教授鉴定。 样品采集后去除杂质, 清水洗净, 50 ℃烘干至恒重。 样品粉碎后, 过60目筛, 保存于自封袋中备用。
| 表1 不同产地绒柄牛肝菌信息 Table 1 Information of Boletus tomentipes samples with different regions |
傅里叶变换红外光谱仪(美国珀金埃尔默公司, 配有DTGS检测器)、 UV-2550紫外可见光分光光度计(日本岛津公司)、 奥豪斯电子分析天平(梅特勒-托多仪器有限公司)、 YP-2型压片机(上海市山岳科学仪器有限公司)、 SY3200-T超声波清洗仪(上海声源超声波仪器设备有限公司)、 Milli-Q Academic纯水系统(美国密理博公司)、 60目不锈钢筛盘(浙江上虞市道墟五四仪器厂)、 双圈定性滤纸(杭州沃华滤纸有限公司)、 101A-1型电热鼓风恒温干燥箱(上海市崇明实验仪器厂)、 溴化钾(分析纯; 天津市风船化学试剂科技有限公司)、 氯仿(分析纯; 云南汕滇药业有限公司)。
准确称取样品(1.5± 0.2) mg, 溴化钾(100± 2) mg, 置于玛瑙研钵中混合研磨成细粉, 将细粉倒入模具中压成薄片。 将仪器分辨率设置为4 cm-1, 扫描范围4 000~400 cm-1, 预热30 min后测定光谱, 扫描前使用空白去除背景中二氧化碳和水的干扰, 样品重复测定3次, 取平均光谱作为样品测量FTIR光谱。
精密称取0.07 g样品于25 mL试管中, 加入8 mL氯仿, 超声提取30 min后过滤得氯仿提取液。 紫外光谱扫描范围230~600 nm, 采样间隔1.0 nm, 狭缝宽1.0 nm。 测定前氯仿溶液作为参比溶液与测试溶液进行基线校正, 然后将测试液换为样品提取液, 测定样品UV-Vis。
受仪器噪音、 实验环境、 样品差异等因素干扰, 原始光谱中包含大量无关信息[28]。 采用Omnic8.2软件对样品FTIR原始光谱进行吸光度转换、 纵坐标归一化、 自动基线校正处理。 采用SIMCA13.0+软件对样品UV-Vis和FTIR光谱进行标准正态变换(standard normal variate, SNV)、 多元散射校正(multiplicative scatter correction, MSC)、 正交信号校正(orthogonal signal correction, OSC)、 一阶导数(first-derivative, 1D)、 二阶导数(second-derivative, 2D)和平滑(Savitzky-Golay, SG)处理, 比较不同预处理方法的分类效果, 选择最佳预处理方法。 数据融合前用MATLAB-R2014a对UV-Vis光谱数据进行归一化处理。 不同模型数据集利用SIMCA-P+13.0和MATLAB-R2014a软件, 分别进行PLS-DA与SVM训练, 建立判别模型。 Origin 8.0软件绘制UV-Vis与FTIR光谱图。
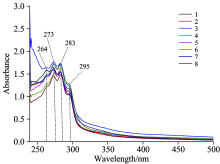
图1为8个产地绒柄牛肝菌平均UV-Vis光谱, 光谱在200~240 nm波长受外界干扰严重, 500 nm以后波长无明显特征吸收峰, 因此选取240~500 nm波段样品信息, 用于区分不同产地绒柄牛肝菌。 样品光谱在波长264, 273, 283与295 nm附近有明显的吸收峰, 其吸光度存在差异。 比较不同产地样品波形, 可知3号(云南省普洱市思茅区六顺乡)与其他产地样品差异较大, 其余7个产地样品相似。 结果表明不同产地来源的样品, 提取液中低极性化合物含量可能存在差异。
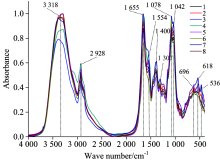
图2为预处理后绒柄牛肝菌平均FTIR光谱。 由图可知, 不同产地样品FTIR光谱在3 318, 2 928, 1 655, 1 554, 1 400, 1 078和1 042 cm-1等附近有明显特征吸收峰。 3 318 cm-1附近主要为O— H与N— H伸缩振动吸收峰, 2 928 cm-1附近为亚甲基C— H反对称的伸缩振动吸收峰, 1 655 cm-1附近主要为蛋白质酰胺Ⅰ 带的C— O伸缩振动吸收峰, 1 554 cm-1附近主要为蛋白质酰胺Ⅱ 带的C— O伸缩振动, 1 400 cm-1附近为亚甲基的弯曲振动, 1 078~1 031 cm-1吸收峰为C— O和C— C伸缩振动[29]。 以上特征吸收峰, 可能为糖类、 蛋白质、 氨基酸、 维生素等化合物吸收峰[30], 吸收峰吸收强度和位置的差异, 表明不同产地样品间化学成分含量可能存在差异。
偏最小二乘判别分析(partial least squares-discriminant analysis, PLS-DA), 利用自变量矩阵X和分类变量Y建立回归判别模型, 能解决数据集噪声大、 样本少和维度高的问题[31]。 Q2表示PLS-DA模型预测能力, R2Y表示对模型自变量的解释能力, Q2与R2Y值越接近于1模型越好[32]。 表2为不同方法预处理UV-Vis和FTIR光谱后, 建立PLS-DA模型的Q2和R2Y。 比较不同预处理PLS-DA模型的Q2可知, 采用MSC+2D对UV-Vis进行预处理, 前6个主成分的Q2最大, 为50.85%, R2Y为61.58%, SNV+2D处理FTIR光谱, 前17个主成分的Q2最大, 为82.16%, R2Y为95.09%。 表明分别采用MSC+2D与SNV+2D处理UV-Vis与FTIR光谱, 能减少干扰对模型的影响, 放大重叠谱带的化学信息, 使PLS-DA模型分类准确率增加。
| 表2 不同方法预处理UV-Vis和FTIR光谱后PLS-DA模型Q2和R2Y Table 2 Q2 and R2Y of PLS-DA models of UV-Vis and FTIR spectra with different pre-processing method |
低级数据融合将预处理后的UV-Vis与FTIR光谱数据串联, 得到新的数据集进行模型训练, 中级数据融合将UV-Vis, FTIR光谱数据进行PLS-DA降维, 提取UV-Vis, FTIR的前6与前17个主成分进行融合[33]。 Kennard-Stone算法选择26个样品作为预测集, 其余53个样品为训练集, 对UV-Vis, FTIR, 低级和中级数据融合进行PLS-DA训练, 建立判别模型。 假设模型训练集与预测集样品的预测值在0.5~1.5范围, 表示分类正确, 不在这个范围的样品, 表示分类错误, 预测值越接近于1, 分类效果越好[34]。 不同PLS-DA模型的预测结果见表3, 比较四种模型判别效果, 结果显示, 数据融合策略分类准确率最高, 且低级与中级融合模型分类效果一样, 仅有2个样品分类错误, UV-Vis模型效果最差, 24个样品分类错误。 表明对不同产地来源的绒柄牛肝菌进行PLS-DA分析, 数据融合优于单一种仪器。
SVM是由Vapnik等[35]针对小样本训练和分类提出, 是一种基于机器学习理论, 进行数据挖掘的新方法, 其优势是解决小样本、 非线性与高维数据等问题。 采用Kennard-Stone算法选择26个样品作为预测集, 其余53个样品为训练集, 网格搜索法对UV-Vis, FTIR, 低级和中级融合数据集进行最佳参数筛选, 建立不同的SVM模型, 图3为不同SVM模型训练集参数c和γ 的优化结果图, 数据融合SVM模型交叉验证正确率高于UV-Vis与FTIR模型。 将参数c和γ 带入SVM进行训练, 得到训练集和预测集分类正确率(表4), 比较不同SNV模型的预测结果。 结果显示, 中级数据融合模型> 低级数据融合模型> FTIR模型> UV-Vis模型, 中级数据融合模型分类准确率最高, 79个样品分类全部正确。 表明对不同产地的绒柄牛肝菌进行SVM鉴别分析, 中级数据融合策略能区分开相似度较高的样品, 分类效果最佳。
| 表3 不同模型的预测结果 Table 3 The parameter and result of different models |
| 表4 不同SVM模型的预测结果 Table 4 The results of different SVM models |
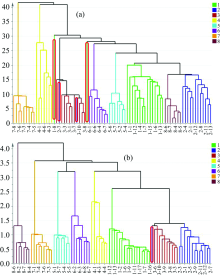
HCA依据不同测定方法提供的样品整体化学信息相似程度, 将比较相似的样品聚为一类[36]。 图4为绒柄牛肝菌数据融合HCA图, 图中同一种颜色的样品为同一产地, 标红色的样品为不能与同一产地聚为一类的样品, 横坐标为样品名, 纵坐标为不同产地间临界值的距离, 距离越小, 表示样品越相似。 结果显示, 低级数据融合[图4(a)]中4个样品不能与同一类产地聚为一类, 中级数据融合[图4(b)]仅1个样品不能与同一类产地样品聚为一类, 中级数据融合优于低级数据融合, 能将相似度高的样品区分开。 分析中级数据融合HCA结果, 当距离为2.5时, 8个产地样品被分为七组, 第一到六组分别为8(云南大理州), 7(四川省凉山州)、 5(云南省红河州)、 6(云南省迪庆州)、 4(云南省曲靖市)、 1(云南省玉溪市)号产地样品, 第七组为3(云南省普洱市)和2(云南省楚雄州)号产地样品, 聚类距离从第一组到第七逐渐变小, 表明云南省普洱市、 楚雄州与大理州样品整体化学成分差异最大, 云南省普洱市与楚雄州样品的整体化学成分较相似。 当聚类距离为1.3时, 云南省玉溪市峨山县富良棚乡、 小街乡, 以及云南省楚雄州姚安县前场镇、 南华县沙桥镇的样品分别聚为一类(1-1~1-7, 1-8~1-17和2-1~2-7, 2-8~2-14), 表明同一产地样品间整体化学成分类较相似, 且同一产地不同采集地点的差异小于不同产地之间的差异。
通过测定8个不同来源的绒柄牛肝菌UV-Vis和FTIR光谱, 采用MSC, SNV, 1D, 2D, SG等预处理方法, 对原始光谱进行优化处理并建立产地溯源模型。 结果显示, MSC+2D和SNV+2D分别为UV-Vis和FTIR光谱的最优预处理方法。 UV-Vis, FTIR, 低级和中级数据融合数据结合PLS-DA和SVM, 建立牛肝菌产地鉴别模型, 并对光谱融合数据进行HCA分析, 结果显示, 数据融合策略优于单一UV-Vis和FTIR模型, 表明数据融合策略利用不同来源数据的互补性, 增加样品整体化学信息, 从不同的层面反映牛肝菌差异, 提高了样品分类正确率。 SVM中级数据融合优于低级数据融合模型, 表明中级数据融合对原始数据中有效信息进行了更深入的挖掘, 显示出更高的分类准确率。 基于低级与中级数据融合建立的PLS-DA与SVM模型, 样品分类错误总数分别为2和0, 表明SVM模型分类效果优于PLS-DA模型。 分析中级数据融合的HCA图, 可知同一产地不同采集地点的样品聚为一类, 表明同一产地样品整体化学成分类较相似, 且同一产地不同采集地点的差异小于不同产地之间的差异。 采用UV-Vis, FTIR, 中级数据融合策略结合SVM, 为野生食用菌产地溯源提供了方法, 进一步完善野生食用菌产地溯源体系。
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|