作者简介: 冯旭萍, 1984年生, 浙江大学生物系统工程与食品科学学院博士后 e-mail: pimmmx@163.com
突变体的筛选与鉴定是育种工作中的重要环节。 该研究基于高光谱成像技术实现了水稻CRISPR/Cas9突变体种子的可视化鉴别。 采集了水稻HD野生型和CRISPR/Cas9突变体种子共1 200粒样本的高光谱图像数据, 通过Kennard-Stone算法, 按照2∶1的比例构建了建模集(800)和预测集(400)。 对水稻种子的原始光谱经过WT预处理后, 通过2nd derivative提取了24个特征波长, 分别基于全谱和特征波长建立径向基函数神经网络(RBFNN), 极限学习机(ELM)和K最邻近法(KNN)模型。 试验结果表明, 无论是基于全谱还是特征波长神经网络模型都取得了良好的识别能力。 通过2nd derivative提取的特征波长结合RBFNN模型也取得了较好的鉴别结果, 其建模集和预测集分别达到了92.25%和89.50%。 基于2nd derivative-RBFNN结合图像处理技术, 可以实现水稻CRISPR/Cas9突变体种子的可视化鉴别, 实现种子的定位和识别。 结果表明应用高光谱成像技术, 结合化学计量学方法和图像处理技术对水稻CRISPR/Cas9突变体的鉴别具有可行性, 可为水稻育种中大量突变体的快速、 准确地筛选和鉴定提供技术手段。
Mutant screening is an important step for CRISPR/Cas9 gene editing technology employed in crop breeding program. The present study proposes a visual identification method of CRISPR/Cas9-induced rice mutants based on near-infrared hyperspectral image technology. A total of 1 200 samples of rice seeds were collected, comprising 600 wide types and 600 CRISPR/Cas9-induced mutant samples. The whole data set was divided into two groups according to the Kennard-Stone algorithm, a calibration set (400 samples) and a prediction set (200 samples) for each class. 24 optimal wavelengths were selected by 2nd spectra algorithm after preprocessing the selection spectral region with absolute noises by wavelet transform. Radial basis function neural network (RBFNN), extreme learning machine (ELM) and K-nearest neighbor (KNN) were used to build discrimination models based on the preprocessed full spectra and feature wavelengths. The results demonstrated that neural networks models achieved good recognition ability. The RBFNN model calculated on the optimal wavelength showed classification rates of 92.25% and 89.50% for calibration set and prediction set, respectively. Finally, the classification of mutant seeds could be visualized on prediction maps by predicting the features of each pixel on individual hyperspectral image based on 2nd derivative-RBFNN model. It was concluded that hyperspectral imaging together with chemometric data analysis was a promising technique to identify CRISPR/Cas9-induced rice mutants, which offered a powerful tool for evaluating large number of samples from CRISPR/Cas9 gene editing performance trials and breeding programs.
引 言
随着现代生物技术的发展和大量物种测序工作的完成, 作物育种学已经开始进入基因组定向编辑辅助育种的阶段。 近年来备受瞩目的基因编辑技术CRISPR/Cas9(clustered regularly interspaced short palindromic repeats/CRISPR-associated nuclease 9, Cas9)技术可以实现对目的基因组的敲入、 缺失、 碱基突变等修饰, 可以精准地改变基因组, 为农作物的遗传改良提供了广阔的前景[1]。 但是该基因编辑技术, 转染难度相对较大, 具有碱基识别偏好性, 局限了基因编辑的运用范围, 而且会导致不同基因位点编辑效率不同[2]。 因此CRISPR/Cas9阳性突变体的筛选和鉴定仍然需要较大工作量。 然而目前突变体的筛选主要是通过基因测序筛查的方法, 操作程序复杂, 对实验操作人员的专业技能要求高, 前处理繁琐, 需要多种生化试剂, 不适应于现代农业中大规模生产的要求。
虽然光谱仪器不足以检测DNA的浓度, 却可以记录伴随DNA修饰后的蛋白质变化, 这是因为近红外光谱可以反映蛋白质中大量X-H含氢基团倍频和合频的吸收信息, 因此也就具备了识别作物基因突变体的理论基础[3]。 王海龙等以近红外高光谱成像技术, 采用偏最小二乘判别分析(partial least squares-discriminant analysis, PLS-DA)模型实现了对大豆多个品种的转基因鉴别[4]。 Liu等[5]利用可见/近红外光谱技术, 结合主成分分析方法和支持向量机(support vector machine, SVM)模型实现了转基因水稻种子的鉴别, 建模集和预测集都达到了近100%的效果。 此外, Munck[6], Biradar[7]等也应用近红外光谱技术结合化学计量学方法实现转基因作物的识别。 但是这些研究都是鉴别转入异源基因的农作物的识别, 目前国内外基于光谱技术对CRISPR/Cas9基因编辑技术获得的突变体的筛选和鉴别还没有涉及。
因此, 本研究的主要目的包括: (1)结合波谱和光谱成像技术, 研究利用高光谱技术实现水稻CRISPR/Cas9突变体的育种筛选; (2)基于最佳建模集, 提取特征波长, 简化预测模型, 提高预测速度; (3)对水稻突变体种子进行可视化鉴别, 直观显示突变体的位置和数量。
研究以淮稻5号(HD野生型)为遗传转化受体, 应用CRISPR技术进行目标基因的敲除, 编辑和构建载体。 突变体的构建利用试剂盒(北京唯尚立德生物科技有限公司)将gRNA靶点序列插入到Cas9/gRNA质粒中。 构建好的 Cas9/gRNA质粒能够同时表达植物密码子优化的Cas9蛋白及gRNA, 然后将构建好的质粒进行遗传转化获得阳性转化个体, CRISPR/Cas9转基因材料种植于浙江省农科院转基因试验基地, 进行常规田间种植及管理。
采用CTAB法提取水稻分蘖盛期单株叶片的基因组DNA, 之后放置于-20 ℃冰箱中保存。 将提取的DNA用于PCR扩增实验, PCR体系如下: DNA 5 μ L, 正反向引物(10 μ mol· L-1)各1.5 μ L, dNTPs(2 μ mol· L-1)10 μ L, 2× 缓冲液25 μ L, KOD Fx酶1 μ L, 加ddH2O补足50 μ L。 PCR产物在 2%琼脂糖凝胶上进行电泳, 电泳结束后 EB 溶液显色并拍照, 需要测序的样品直接切胶回收送上海博尚生物技术有限公司。
利用CRISPR技术对THOUSAND-GRAIN WEIGHT 6 (TGW6) 基因进行编辑[8], 获得了阳性转基因植株, 对转基因苗提取相应的DNA, 利用传统测序方法对编辑目标基因序列进行PCR测序。 测序结果表明, 突变材料在编辑位点附近发生基因突变(HD突变体)。 所用突变材料都仅在靶标基因位点存在碱基突变, 在其他性状方面则没有差异(图1)。
水稻种子光谱数据的采集使用高光谱成像仪, 其波长范围是874~1 734 nm, 共有256个波段。 本研究所用系统配置有ImSpector N17E光谱仪(Spectral imaging LTD., Oulu, Finland), 两个150 W卤钨灯的2900Lightsource线光源(Illumination Technologies Inc., USA), 型号为OLES22的C-mount成像镜头(Spectral imaging LTD., Oulu, Finland), 型号为IRCP0076d的电控位移平台(Isuzu Optics Crops., Taiwan, China), 高光谱成像系统采集软件(Isuzu Optics Crops., Taiwan, China)以及配套的电脑。
在采集近红外光谱信息以前, 需要对高光谱仪器进行最优参数调整, 从而获得高质量的高光谱图像信息, 这些参数主要包括相机的曝光时间, 载物台的移动速度和物镜高度。 本研究中N17E高光谱成像仪的最优参数设置为: 曝光时间是3.2 ms, 载物台的运行速度为22 mm· sec-1, 镜头至样本的高度为28.7 cm, 图像的分辨率为320× 256。
对采集到的近红外高光谱图像进行校正, 图像校正如式(1)
式(1)中, Ic为校正后的高光谱图像; Iraw代表原始采集的图像, Iwhite代表反射率接近于100%的白板校正图像, Idark代表反射率接近于零的黑板校正图像。
高光谱数据采集时除了样本本身的信息外, 还受到噪声、 散色光以及背景等因素的影响。 在用化学计量学方法建立模型时, 光谱数据的预处理方法对模型的准确性起到了关键的作用, 因此, 需要对采集到的原始光谱进行预处理, 从而提高信噪比。 小波变换(wavelet transformation, WT)是一种有效的图像和行号处理的方法, 可用于光谱的平滑去噪[9]。 本研究采用WT进行光谱预处理, 小波基函数为db8, 分解尺度为3。 选取预处理后高光谱图像中每个水稻种子作为感兴趣区域(region of interest, ROI), 并且以ROI内所有像素点光谱的平均值作为该水稻样本的光谱数据进行分析。
本研究对所有水稻种子进行类别赋值, 将野生型HD赋值为1, 突变体都赋值为2。 共扫描获得野生型HD和HD突变体分别为600个。 通过Kennard-Stone算法[10], 按照2∶ 1的比例将HD野生型和HD突变体划分为建模集和预测集, 见表1。
| 表1 HD水稻野生型及其CRISPR/Cas9编辑后突变体品种赋值与建模集和预测集样本划分 Table 1 Class value assignment and dataset split of rice wide type and CRISPR/Cas9-induced mutants |
近红外光谱首尾两端明显受到噪声的影响, 为了获得较好的辨别效果, 本试验采用了波长范围在975~1 646 nm之间的光谱进行分析。
分别计算水稻HD野生型及其突变体的近红外光谱数据的平均值, 其平均光谱曲线如图2。 由图可知, 水稻种子平均光谱的曲线具有一致的趋势, 波峰、 波谷的位置一致, 但是反射率有所差别。
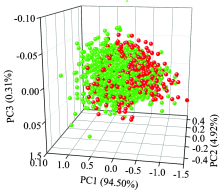
主成分分析(principal component analysis, PCA), 是一种被广泛用于数据降维的有损压缩技术, 基本原理是将原来具有一定相关性的众多变量, 在损失较少信息的前提下通过线性变换至新的坐标系转化为几个综合变量, 即主成分(PC)[11]。 本研究将WT预处理后的全谱光谱数据作为输入进行PCA分析, 以定性研究区分水稻野生型和其CRISPR/Cas9突变体的可能性。 样本中PC1, PC2和PC3分别解释了94.50%, 4.92%和0.31%的变量, 前3个主成分共解释了99.73%的变量。 获得基于3个主成分的3D散点图(图3), 由图3可知, HD水稻野生型和其突变体都聚集成簇, 但是没有明显的分类趋势, 需要采用其他的辨别模型进行区分。 无明显分类趋势主要是因为HD水稻野生型和其突变体仅在单基因TGW6上有所差别, 两个品种的整体具有相似性。
高光谱图像获得的全波段光谱中包含有大量共线性、 冗余信息及噪声, 从而影响了模型的预测性能。 特征光谱的选择可以大大减少建模所用变量的个数, 去除冗余、 共线性的波长, 从而提高建模速度, 减少计算量[12]。 导数能够消除背景光谱信息, 提高光谱的分辨率。 导数光谱中峰和谷代表的是非背景的有用信息, 而出现的峰谷差异表明不同样本间光谱的差异, 因此可以作为特征波长[13]。 本研究基于二阶导数光谱法(2nd Derivative)进行特征波长的选择, 与全波段相比, 基于2nd Derivative提取的特征波长只占其全波段变量数的12%(图4)。
 | 图4 水稻HD野生型和CRISPR/Cas9突变体种子基于2nd Derivative方法选取的特征波长Fig.4 Optimal wavelength selection of HD rice wide type and CRISPR/Cas9 mutants by 2nd Derivative method |
近红外光谱能够反映物质分子中单个化学键的基频振动的倍频和合频, 其中包含了多数有机化学物的分子构造和组成成分, 它们常常包含一些氢基团(N— H, O— H, C— H, S— H等)振动的倍频和合频重叠。 采用2nd Derivative获得的相关波长(图4)与蛋白质中官能团(— CH基团, 1 200 nm附近[14]; N— H单键, 1 490~1 530 nm附近[15]; — N
判别分析是一种重要的统计分析方法, 该方法的基本原理是按照一定的判别准则, 根据类别已知的样本信息总结出分类的规律和准则, 建立判别函数, 判别新的样本点所属类别。 本研究主要应用了三种建模方法来区分和筛选突变体水稻种子, 包括: 径向基函数神经网络(radial basis function neural networks, RBFNN)[17]、 K最邻近法(K-nearest neighbor, KNN)[18]和极限学习机(extreme learning machine, ELM)[19]。
RBFNN是一种高效的三层前馈神经网络, 其基本原理是基于RBF构成的隐单元的“ 基” 将输入数据映射至隐空间, 实现输入层和隐含层之间的数据变化[17]。 在网络训练中, RBFNN的输入函数为:
RBFNN的输出函数为
式中, φ i(‖ X-Ci‖ )为径向基函数, K为隐含层节点数, wi为第i个隐含层与输出层的权值, Ci为第i个隐含层节点中心, 一般φ 0为1。
KNN是一种非参数机械学习算法, 其模型原理是通过计算检测样本与训练集样本数据中不同类别数据间的距离进行分类和预测, 距离近者归为一类[18]。 KNN的最大优点是不需要训练集样本必须是线性划分的, 也不需要单独的训练过程, 并且能够处理多类问题。
ELM是一种新型的单隐含层前馈神经网络学习算法, 可以随机初始化输入权重和隐含层神经元的阈值, 并得到相应的隐节点输出, 不需要调整就可以获得最优解[19]。 ELM的最大优点是学习速度快、 泛化性能好。
分别基于WT预处理后的全波段和特征光谱建立RBFNN, KNN和ELM判别分析模型, 并以识别准确率作为建模有效性的评价指标, 模型的识别效果如表2。 对这三种判别分析方法进行比较发现, 基于全波段的神经网络RBFNN和ELM模型对水稻CRISPR/Cas9突变体的识别率较好。 其中RBFNN的模型判别能力最佳, 建模集和预测集的判别正确率达到了95.75%和92.50%。 而KNN模型的判别效果较差, 建模集和预测集的正确率只有75.62%和84.25%。 基于特征波长建立判别模型后, 模型的判别效果都有所降低。 KNN模型的判别效果最差, 而RBFNN和ELM模型均取得了较好的判别结果, 建模集和预测集的判别正确率在90%左右。
| 表2 水稻HD野生型和CRISPR/Cas9突变体基于全谱和特征波长的判别分析结果 Table 2 Discriminant results of HD rice wide type and CRISPR/Cas9 mutants based on full wavelength and optimal wavelength |
结果表明, 采用近红外光谱技术能够用于水稻CRISPR/Cas9突变体的识别, 特别是RBFNN模型能有效的识别突变体水稻种子。
高光谱图像能够同时提供样本的光谱信息和空间信息, 基于图像与光谱或者两者相结合进行相关分析[20]。 由于光谱信息和空间信息的对应关系, 基于样本平均光谱建立的校正模型可以用于样本像素点光谱的预测。 但是基于高光谱图像信息数据量较大, 基于全波段范围光谱预测时对计算机硬件要求高而且计算速度慢。 为提高计算速度, 本研究基于2nd Derivative提取的特征波长建立的RBFNN模型用于样本像素点的预测(图5)。
由图5可以发现, 从肉眼上很难区别HD水稻野生型和CRISPR/Cas9突变体品种, 但是从高光谱可视化伪彩图上可以清晰地辨别突变体样本, 虽然有较小部分种子有所判错。 由于高光谱图像分割算法影响和成像系统图像分辨率较低, 可视化图中水稻种子存在一定的变形, 但总体维持原有的外形特征。 通过高光谱的可视化图可以实现对水稻单粒样本的快速定位和识别, 为简化水稻CRISPR/Cas9育种中大量突变体的筛选过程提供了思路。
CRISPR/Cas9基因编辑技术在作物遗传育种中有着不可估量的前景, 然而由于该技术突变效率和遗传特性等问题使得突变体的筛选仍然需要大量的研究工作。 为了有效地缩短水稻育种进程, 提高育种效率, 本研究探索基于高光谱成像技术实现水稻CRISPR/Cas9突变体种子可视化鉴别的可能性。 我们以水稻HD野生型和CRISPR/Cas9突变体为研究对象, 对经过WT处理后的光谱基于Kennard-Stone算法构建了建模集和预测集, 并分别基于全谱和2nd Derivative提取的特征波长分别建立判别分析模型。 研究发现, 在水稻CRISPR/Cas9突变体种子的判别分析模型中, 基于全谱和特征波段的RBFNN和ELM模型都取得了良好的判别效果。 其中基于2nd Derivative提取的24个特征波段构建的RBFNN模型, 其建模集和预测集识别正确率分别达到了92.25%和89.50%。 通过高光谱光谱信息建立的2nd Derivative-RBFNN模型结合图像处理技术, 对水稻种子突变体进行了可视化鉴别, 实现了对单粒种子的快速定位和准确识别。 研究结果表明, 提出的方法可以用于水稻育种过程中突变体的有效快速筛选, 有效地缩短育种进程, 进而指导育种工作向功能化、 专业化方向发展。
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|