作者简介: 梅从立, 1978年生, 浙江水利水电学院电气工程学院副教授 e-mail: clmei@ujs.edu.cn
近红外技术广泛应用于食品、 药品等生产过程和产品质量检测, 具有样品无需预处理、 成本低、 无破坏性、 测定速度快等优点。 但是, 全光谱数据维数高、 冗余信息多, 直接应用于建模会导致模型复杂性高、 稳定性差等问题。 siPLS是最常见的光谱数据降维方法, 但是难以处理光谱数据的共线性问题。 LASSO是一种相对新的数据降维方法, 但在小样本应用中具有不稳定性。 针对siPLS和LASSO在近红外光谱数据应用中存在的问题, 提出了基于siPLS-LASSO的近红外特征波长选择方法, 并将其应用于秸秆饲料蛋白固态发酵过程pH值监测。 该方法首先采用siPLS算法, 实现对光谱波长最佳联合子区间的优选; 然后, 对优选联合子区间使用LASSO算法进行特征波长选择, 在此基础上建立PLS校正模型。 同时, 将siPLS-LASSO方法与其他传统特征波长选择方法进行了对比。 结果表明: 建立在siPLS-LASSO方法优选33个特征波长基础上的PLS模型预测结果更好, 其预测方差(RMSEP)和相关系数( Rp)分别为0.071 1和0.980 8; 所提siPLS-LASSO方法有效选取了特征波长, 提高了模型预测性能。
Near-infrared spectroscopy (NIR) is widely used in entire production processes and product quality test, especially in food and drug industries. It has many advantages, e. g. no requirement of sample pretreatment, low cost, non-destructive detection, and fast determination. However, the application of the whole spectrum data in modeling can lead to complexity and poor stability. The synergy interval PLS(siPLS) is the most common dimensionality reduction method for spectral data. However, it cannot deal with the collinearity problem of spectral data. Least absolute shrinkage and selection operator (LASSO) is a relatively new method for data dimensionality reduction. However, when it comes to small samples, its instability cannot be ignored. For disadvantages of siPLS and LASSO in NIR calibration, a novel wavelength selection method named siPLS-LASSO was proposed. It was validated in a wheat-straw solid-state fermentation process by monitoring pH values. In the method, siPLS was firstly used to selected intervals of NIR spectroscopy. Secondly, LASSO was used to select wavelengths on the selected intervals. Finally, the selected wavelengths were used to construct PLS model for prediction. For comparisons, several conventional wavelength selection methods were also studied. In the case study, 33 wavelengths were eventually selected by the siPLS-LASSO method and used for PLS modelling. The RMSEP and Rp of the model were 0.071 1 and 0.980 8 respectively. Results showed that the proposed siPLS-LASSO was an effective method of wavelength selection and can improve prediction performance of models.
引 言
近红外光谱是20世纪90年代以来发展最快、 最引人注目的现代分析技术, 是光谱测量技术和化学计量学的有机结合[1]。 近红外光谱区主要由含氢基团的倍频和组频吸收峰组成, 波长点多、 吸收强度低、 光谱信息之间存在严重的共线性关系。 如果将全光谱数据用于建模, 一方面会提升模型复杂度; 另一方面冗余信息过多往往会影响模型预测精度和稳定性。 因此, 很少将采集的全光谱数据直接用于建模, 往往建模前首先要进行波长优选[2]。
光谱波长优化选择方法通常包括无信息变量消除法(UVE)、 间隔偏最小二乘法(iPLS)、 组合区间偏最小二乘法(siPLS), 遗传算法(GA)等[2, 3]。 siPLS是一种应用相对广泛的波长优选方法。 但是, 经过该方法优选的最佳联合区间, 没有能考虑到优选区间内光谱信息间的共线性问题。 目前, 关于波长优选仍然在进一步的研究中。
最小绝对收缩和选择算子方法(least absolute shrinkage and selection operator, LASSO), 是一种相对新的特征变量选择方法。 该方法是一种基于线性回归模型的降维方法, 最初由Tibshirani于1996年提出[4]。 LASSO能够同时实现变量选择和参数估计, 有效解决抗干扰能力差, 无法产生稀疏解等问题。 因此被广泛应用于机器学习、 生物医学、 图像处理等多个领域[5, 6]。 LASSO通过引入范式约束产生稀疏模型, 可以将大量的冗余变量去除, 只保留与响应变量最相关的解释变量, 有效地解决了高维数据建模中的诸多问题。 但是, 高维数据中经常面临数据维数大于样本个数的问题。 当出现这一问题时, LASSO特征波长选择个数将小于样本个数, 无法保证数据集中所有重要信息被完全保留[7]。 由于许多光谱数据样本数据集存在光谱波长数大于样本个数的情况, 不适合将LASSO方法直接应用于波长优选问题。
针对siPLS方法和LASSO方法在波长优选中存在的问题, 提出了siPLS-LASSO方法。 首先采用siPLS方法对全光谱区域分区, 选取最佳联合子区间。 然后, 对所选最佳联合子区间采用LASSO算法进一步降维。 最后, 基于优选变量建立PLS校正模型, 并将其应用于秸秆蛋白饲料发酵过程pH值快速检测。
siPLS是Norgaard[8]提出的iPLS波长选择方法的一种扩展, 其主要思想是采用全谱区域的一部分子区间建立局部PLS模型。 这一部分光谱对于目标变量变化最为敏感, 同时避免了其他区间光谱与噪声的影响。 具体实现步骤简述如下: 在iPLS的基础上, 将同一次区间划分中, 精度较高的几个局部模型所在的子区间联合起来, 以RMSECV值为联合模型衡量指标, 在所有模型中选出最佳联合子区间(RMSECV最小)。 在该最佳联合子区间基础上建立的PLS模型预测能力最强。
LASSO[4]方法是一种基于线性回归模型的降维方法, 基本思想是将回归系数的绝对值之和约束在一个常数条件下, 使残差平方和最小。 从而产生某些严格等于0的回归系数, 产生稀疏解实现变量选择。
已知线性回归模型
式(1)中y∈ RN为响应向量, X∈ RN× P为设计矩阵, β ∈ RP为回归系数向量, ε ∈ RN为误差向量。 LASSO最小化残差平方和为
式(2)中t≥ 0为可调参数(tuning parameter), 当t设置较小时, 绝对值小的参数被自动压缩为0, 这些系数为0的变量即冗余变量, 可以被剔除; 当t的值过大, 则约束条件失效。 为确保模型性能, 采用五折交叉验证法确定可调参数t的范围。 将原始样本随机拆分成5份, 取其中一份为验证集, 其余4份作为训练集, 建立LASSO模型并计算每个模型预测误差。 取使得预测误差最小的作为最优调整参数。
siPLS通过选择与响应变量最相关的最佳联合光谱子区间来实现剔除冗余变量的目的, 从而对光谱数据进行降维。 然而, 所选最佳联合子区间内仍然存在一些共线性的冗余变量, 这些冗余变量不可避免的会影响模型性能。 因此有必要对优选联合子区间作进一步变量筛选。
提出的siPLS-LASSO可以解决上述问题。 该方法采用siPLS算法, 首先对全光谱区域分区, 对每个局部子区间分别建立PLS模型, 选取RMSECV较小的子区间作为入选子区间; 然后, 优化入选子区间, 以最低RMSECV确定最佳联合子区间; 最后, 对所选区间采用LASSO方法选出最优变量。
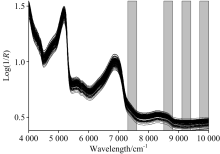
秸秆蛋白饲料固态发酵过程中pH值难以在线测量, 研究采用近红外光谱校正模型实现对pH值的快速检测。 研究使用的数据为采集样本近红外光谱数据和离线实验分析pH值数据组成。 试验使用AntarisTM Ⅱ 型傅立叶变换近红外光谱仪采集发酵物样本光谱, 检测器采用InGaAs, 内置参比背景, 以漫反射式积分球附件采集光谱。 光谱波数范围为10 000~4 000 cm-1, 分辨率为8 cm-1, 扫描次数设定为16次, 采样间隔3.865 cm-1, 图1为固态发酵试验原始光谱数据, 每条光谱共计包含1 557个数据点。
实验过程简述如下: 将预处理后的小麦秸秆和麦麸按4∶ 1比例混合并均匀搅拌, 按50%的装料系数将混合后的固态培养基装入GTG型固态发酵罐中, 进行初始pH 和湿度的调节, 湿度调至65%, pH为6.3左右; 在121 ℃下高温灭菌15 min, 冷却后接种, 在(30± 2) ℃条件下培养72 h。 在此期间, 发酵罐的气体流速率设置为65%, 搅拌速率为2 r· min-1, 每4 h搅拌5 min。 在蛋白饲料发酵过程中, 每12 h采集4个样本(采样点分别为0, 12, 24, 36, 48, 60和72 h), 整个发酵过程可收集发酵产物样本28个。 再利用相同的材料及条件发酵5个批次, 共采集固态发酵产物样本140个。 选取4批样本数据作为训练集样本, 一批样本作为验证集样本。 表1为固态发酵物样本的pH值在训练集和验证集中的分布情况。
| 表1 固态发酵过程pH值分布 Table 1 pH distribution of the solid state fermentation process |
采用siPLS方法进行波长优选, 并建立PLS模型预测过程参数pH值。 经过多次计算测试, 当子区间划分数为20, 且最佳联合子区间包括12, 16, 18和20时, 获得最小RMSECV值(如表2)。
| 表2 不同联合子区间选择PLS建模对比 Table 2 Comparisons of PLS models built on different synergy intervals |
所选取4个子区间, 波长分别为7 308.89~7 605.87, 8 512.25~8 809.23, 9 113.93~9 407.06和9 707.90~10 001.03 nm, 共310个波数点集合(如图2)。
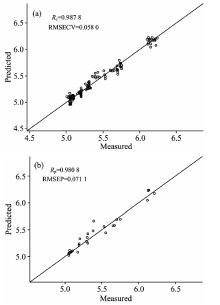
为简化模型, 剔除冗余变量, 对所选择的最佳联合子区间, 进一步使用LASSO算法选择特征波长。 LASSO算法最终选择33个特征波数点, 与原始全光谱1 557个数据点相比, 只使用其中2.1%特征光谱。 图3为基于所选特征波长建立PLS模型的训练集散点图和测试集散点图。 由图3可知, 在训练集中, RMSECV和Rc分别为0.058 0和0.987 8; 在验证集中, RMSEP和Rp分别为0.071 1和0.980 8, 模型训练和预测均达到令人满意的效果。
为进一步说明siPLS-LASSO方法的优越性, 表3给出了与多种特征波长选择算法比较结果, 包括GA, UVE, iPLS, siPLS, LASSO和siPLS-GA等[9, 10]。
| 表3 不同波长选择算法PLS建模性能比较 Table 3 Comparisons of PLS modeling with different wavelength selection methods |
GA因其较强的全局搜索能力, 被应用在全光谱范围内搜索与响应变量相关的有效波长。 计算中, GA参数设定为: 初始种群数目为50, 交叉概率为0.8, 变异概率为0.01, 最大迭代次数为200。 由表3可以看出, GA方法比siPLS-LASSO方法选择变量数更多, 然而模型预测精度却不优于siPLS-LASSO。
在UVE分析中, 样本光谱矩阵加入500个随机噪声变量, 将变量稳定性作为变量筛选条件, 设定变量稳定性绝对值的99%作为阈值, 最大主成分数设为15。 对于全光谱1 557个变量, UVE方法去除了无信息变量829个, 保留原始变量46.75%建立了PLS预测模型。 如表3所示, 该模型预测精度与相关系数与全光谱模型相比有所提高, 但是与其他模型相比结果较差。 主要原因是, UVE可以去除噪声和部分冗余, 但由于近红外光谱数据间差异较小, 导致很多冗余信息被保留, 因此模型变量需进一步筛选。
同时, 表3表明, 基于iPLS与siPLS的PLS模型预测性能仍然存在进一步提升的空间, 原因在于这两种方法都忽视了子区间内部光谱相关性与冗余问题, 因此波长仍然有进一步筛选的空间。 由表3可以看出, siPLS-LASSO优于LASSO方法, 且与其它方法比较各项指标最优。 siPLS-GA与siPLS-LASSO各项指标最接近。 因此在时间复杂度与预测精度方面对siPLS-GA与siPLS-LASSO进行了对比研究。 表4为基于两种方法的PLS模型性能比较结果。 由表4可以看出, siPLS-LASSO与siPLS-GA相比较, 具有更好的预测效果, 同时拥有更少的计算时间。 GA由于其算法复杂度较高, 所需计算花费时间更长。 因此, 所提出siPLS-LASSO算法在计算时间具有明显的优势。
本工作仿真环境: 处理器: Intel(R) Core(TM) i3-2350M CPU: @2.30GHzf, (RAM): 4.00G, 64位Windows7操作系统、 Matlab2012b。
| 表4 基于siPLS-GA与siPLS-LASSO的PLS建模性能比较 Table 4 Comparisons of PLS modeling with siPLS-GA and siPLS-LASSO |
提出了siPLS-LASSO特征波长选择方法。 该方法能克服LASSO与siPLS存在的不足, 实现近红外光谱特征波长优化选择。 以蛋白饲料固态发酵过程实验为研究对象, 采用不同光谱特征波长选择方法, 建立PLS校正模型, 以实现对过程pH值的软测量。 研究表明, siPLS-LASSO算法与多种传统特征波长选择算法相比, 基于其所选波长建立PLS模型具有更好的预测性能。 并且, 通过与性能相近siPLS-GA方法比较, siPLS-LASSO方法在波长选择时间效率上具有明显的优势, 是一种有效地光谱特征波长选择算法。
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|