作者简介: 李秀萍, 1993年生, 云南农业大学农学与生物技术学院硕士研究生 e-mail: 1586182913@qq.com
牛肝菌营养丰富, 味道鲜美, 备受各国消费者青睐。 因种间差异和环境因素的多层次影响, 不同种类及产地牛肝菌品质参差不齐。 目前, 利益驱动导致商家在牛肝菌销售过程中以次充好、 以假乱真的行为扰乱了食用菌市场, 不仅给消费者带来健康风险, 也制约了牛肝菌的国际化贸易。 采用多源异构信息融合策略对牛肝菌种类与产地进行鉴别, 以期为追溯食用菌来源以及正确评价其品质提供一种快速有效的解决方法。 试验样品灰褐牛肝菌( Boletus griseus)、 栗色牛肝菌( B. umbriniporus)、 美味牛肝菌( B. edulis)、 皱盖疣柄牛肝菌( Leccinum rugosicepes)和绒柄牛肝菌( B. tomentipes)五种牛肝菌科( Boletaceae)真菌子实体采于云南省保山市、 昆明市、 玉溪市与红河州。 采用傅里叶变换红外光谱仪(FTIR)和紫外可见分光光度计(UV-Vis)采集样品信息。 Kennard-Stone算法将样品原始数据分为校正集和验证集。 校正集基于FTIR、 UV-Vis、 低级、 中级与高级数据融合建立偏最小二乘判别分析(PLS-DA)模型, 其中决定系数(
Boletus is rich in nutrition, which is favored by consumers all over the world. Due to the differences of species and environmental factors, the quality of boletus of different species and origin vaires. At present, the shoddy, which undermines the sales of genuine boletus and the mushroom market, not only poses a health risks to consumers, but also restricts the international trade of boletus. In this study, the data fusion strategy was used to identify the species and origin of boletus, in order to provide a rapid and effective solution for tracing the source of edible fungi and correctly evaluating their quality. The test samples Boletus griseus, B. umbriniporus, B. edulis, Leccinum rugosicepes and B. tomentipes of five species of boletus fungi fruiting bodies collected from Baoshan, Kunming, Yuxi and Honghe Prefecture of Yunnan province. The chemical information was collected with Fourier transform infrared spectroscopy (FT-IR) and UV-Visible spectrophotometer (UV-Vis). The Kennard-Stone algorithm was used to divide the raw data of samples into calibration sets and validation sets. The calibration set established partial least squares discriminant analysis (PLS-DA) models based on FT-IR, UV-Vis, low-level, mid-level and high-level data fusion. The determination coefficients
牛肝菌为牛肝菌科大型真菌, 多数牛肝菌肉质肥厚, 味道鲜美, 其子实体富含氨基酸、 无机盐、 纤维素、 多糖、 膳食纤维、 生物碱等人体必需营养元素[1, 2, 3], 有抗菌抗癌、 抗氧化、 降血糖、 增强免疫、 促进认知等功效[4, 5, 6]。 云南野生牛肝菌224种, 约占全国种类的57.44%, 年出口1.3万吨左右, 占全省野生食用菌总出口的50%以上, 创汇约8× 107美元[7, 8], 具有极高的经济价值。 由于食用菌种间差异和环境因素的综合影响, 不同种类及产地食用菌品质存在差异[9, 10]。 不良商家见机在存储和销售过程中以次充好, 以假乱真的行为使有毒品种或矿产区毒菌流入市场, 造成误食死亡及重金属超标出口受阻。 这不仅成为影响人类健康的公共问题, 也引起了国家食品安全部门的高度重视。 为此, 亟需一种快速准确的牛肝菌来源鉴别方法, 以正确评价不同种类及产地牛肝菌的真实品质, 为食品监督提供依据。
目前, 红外光谱和紫外光谱技术以其操作简单、 低成本、 高效以及无污染等特点, 广泛应用于食品鉴别[11, 12]与质量评价[13, 14]。 Hirri等[15]利用傅里叶变换红外光谱(FTIR)和偏最小二乘判别分析(PLS-DA)模型对摩洛哥4种不同品质橄榄油进行了鉴别, 结果显示, FTIR模型外部验证正确率100%, 能准确鉴别高品质初榨橄榄油。 D’ Archivio等[16]利用紫外可见光谱(UV-Vis)结合线性判别分析(LDA)对81份藏红花进行产地鉴别, 结果显示, 8个UV-Vis吸光度值所建模型对两组验证集的预测正确率分别为85%和83%, 实现了样品产地鉴别。 El等[17]利用UV-Vis、 衰减全反射红外光谱(ATR-FTIR)和高效液相色谱(HPLC)对石榴糖浆进行了欺诈检测, 结果发现购于中东地区的石榴糖浆中掺有价格便宜的枣浆。 食品原材料在贮藏、 加工和运输过程中会发生化学反应, 利用单一技术采集的样品信息存在特征性弱甚至缺失的风险。 为解决这一潜在不足, 提高信息丰富度和真实性, 需要多尺度收集样品信息, 将其进行组合、 选择或优化, 实现样品的快速精确鉴别。
数据融合是一种将多源信息进行结合的理论, 其目的是提高被测样品的信息质量, 以期得到更加精确的研究结果[18, 19]。 数据融合具有扩大样品信息、 增强模型鲁棒性等优点, 广泛应用于环境监测、 医疗、 军事、 科研等领域[20]。 Spiteri等[21]利用核磁共振氢谱(1H-NMR)和液相色谱-高分辨率质谱(LC-HRMS)结合中级数据融合对法国、 澳大利亚、 智利等产地的4种花蜜进行鉴别, 结果发现中级数据融合策略能更好鉴别出薰衣草、 橙花和桉树蜜, 成功的对蜂蜜种类及产地进行了鉴别。 Casale等[22]采集了不同种类与产地牛肝菌的3个部位近红外光谱信息, 应用低级数据融合策略, 将菌盖表皮、 菌肉及孢子层光谱数据融合后分析。 结果发现, 孢子层具有更大的化学特征价值, 较不平等分散类(UNEQ), 软独立模型(SIMCA)和偏最小二乘密度模型(PLS-DM)能更有效处理复杂数据, 给出了满意的伪品鉴别结果。 以上研究采用数据融合策略, 取得了理想的样品种类、 产地及伪劣品鉴别结果, 对于食品监督和保护消费者权益具有重要意义。
本研究采用简单快速的FTIR与UV-Vis采集不同种类及产地牛肝菌样品的光谱信息; 为弥补信息单一缺陷, 将这两种异构光谱信息进行融合建立PLS-DA模型。 所采集的样品数据用Kennard-Stone算法分为校正集(2/3)和验证集(1/3), 基于FTIR、 UV-Vis、 低级、 中级和高级融合的数据分别建立牛肝菌种类及产地判别模型, 通过比较各模型验证集样品预测正确率, 选出最佳种类及产地鉴别方法, 提供一种快速准确的样品来源鉴别及质量评价方法。
126份牛肝菌鲜样采自云南省保山、 昆明、 玉溪和红河州, 分别属于牛肝菌科(Boletaceae)的5个不同物种: 灰褐牛肝菌(Boletus. griseus)、 栗色牛肝菌(B. umbriniporus)、 美味牛肝菌(B. edulis)、 皱盖疣柄牛肝菌(Leccinum rugosicepes)、 绒柄牛肝菌(B. tomentipes)见表1。 样品用软毛刷去掉泥土, 陶瓷刀切掉菌体基部和腐烂部位, 纯净水洗净, 通风处晾1 h, 放入烘箱50 ℃烘干, 高速粉碎机粉碎, 过80目筛, 自封袋密封, 避光保存备用。
| 表1 牛肝菌样品信息表 Table 1 Information of Boletes samples |
FW-100型高速粉碎机(天津市华鑫仪器厂)、 80目标准筛盘(浙江上虞市道墟五四仪器厂)、 AR1140型万分之一分析天平(梅特勒-托利多仪器上海有限公司)、 YP-2型压片机(上海市山岳科学仪器有限公司)、 Frontier型傅里叶变换红外光谱仪(美国Perkin Elmer公司)、 TU-1901紫外-可见分光光度计(北京普析通用仪器有限责任公司)、 KQ5200型超声波清洗机(昆明市超声仪器有限公司)。 溴化钾(天津市风船化学试剂科技有限公司), 氯仿(西陇化工股份有限公司)。 试剂均为分析纯(AR)。
红外光谱采集: 称取样品粉末(1.0± 0.2) mg, KBr粉末(100± 0.2) mg, 玛瑙研钵中混合均匀并充分研磨, 填满模具压成透明薄片, 光谱仪扫描。 扫描前先用KBr空白片扣除水和二氧化碳的影响。 设定: 扫描范围4 000~400 cm-1, 分辨率4 cm-1, 信号累计扫描次数16次, 每个样本重复扫描3次, 取平均光谱。
紫外光谱采集: 称取(100± 0.2) mg牛肝菌样品粉末于25 mL试管中, 加入10 mL氯仿, 超声提取40 min, 过三层滤纸, 滤液供试。 光谱仪预热30 min, 提取液扣除背景, 供试液光谱仪扫描, 重复扫描3次, 取平均光谱。 设定扫描条件: 狭缝光谱宽度1.0 nm, 采样间隔0.3 nm。
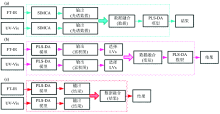
为建立PLS-DA鉴别模型, 不同来源牛肝菌样品FTIR和UV-Vis数据采用Kennard-Stone[23]算法分为校正集(2/3)和验证集(1/3)。 校正集数据用于建立判别模型, 验证集数据对建立模型的预测能力进行外部验证。 光谱原始数据应用SIMCA 13.0基于FTIR、 UV-Vis、 低级、 中级和高级融合数据分别建立牛肝菌种类及产地鉴别模型, 通过比较验证集正确率, 从各模型中选出最适合样品来源鉴别的方法。 低级融合, FTIR和UV-Vis样品信息简单拼接后建立判别模型, 因拼接基于未预处理数据, 过程简单、 快速, 但有可能将噪音信息一并融合。 中级融合选择种类及产地独立判别模型中的潜在变量进行融合后建模。 高级融合通过Zadeh的模糊集合理论实现[24, 25, 26]。 3种多源异构信息融合策略的具体实施方法见图1。
图2为牛肝菌样品种类及产地FTIR和UV-Vis平均光谱图, 由图2中的(a)和(b)可知牛肝菌FTIR主要吸收峰在3 398, 2 927, 1 633, 1 556, 1 383, 1 315, 1 263, 1 205, 1 076, 1 032, 885及615 cm-1处, 出峰位置、 峰数及峰形几乎相同, 但吸光度值存在差异。 3 398 cm-1附近归属为糖、 蛋白质、 纤维素的O— H, N— H伸缩振动; 2 927 cm-1附近为亚甲基不对称伸缩振动; 蛋白质酰胺Ⅰ 带、 酰胺Ⅱ 带和C=N的弯曲振动引起1 633和1 556 cm-1附近的特征吸收; 1 383, 1 315和1 263 cm-1附近为多糖、 蛋白质的C— O— H弯曲振动和— CH2变形振动; 1 205 cm-1归属为碳水化合物吸收峰; 1 076和1 032 cm-1附近为低聚糖、 蛋白质、 纤维素C— O和C— C的伸缩振动; 885及615 cm-1附近归属为α -构型多糖吸收峰[27, 28, 29]。 样品UV-Vis吸收峰主要在274, 283和294 nm处, 峰形相似, 吸收强度有较大差别。 不同来源牛肝菌FTIR和UV-Vis吸收峰的峰位置、 峰形及峰数相似, 但各化学键所引起的特征吸收强度存在差别, 表明不同种类及产地牛肝菌所含成分相似, 但含量有差别。
PLS-DA是基于PLS的多元统计分析法, 能实现海量数据降维并解决回归分析中普遍存在的多重共线性问题[30]。 针对牛肝菌种类及产地的鉴别分别建立基于FTIR、 UV-Vis、 低级、 中级和高级融合数据的PLS-DA模型。 其中决定系数(determination coefficient,
| 表2 PLS-DA模型主要参数 Table 2 Major parameters of PLS-DA models |
| 表3 部分样品的高级数据融合鉴别结果 Table 3 Results of high-level data fusion for some samples |
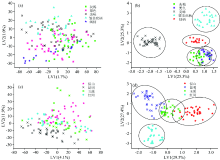
图3为低级和中级融合PLS-DA模型的前两个潜在变量二维散点图。 由图可见, 采用低级融合策略时不同来源样品相互夹杂, 不能实现样品鉴别。 中级融合前两个潜在变量对样品种类原有信息的解释贡献率为50.6%, 对产地原有信息的解释贡献率为56.9%。 相同种类样品成簇, 但栗色牛肝菌与美味牛肝菌有混杂。 玉溪产地样品与来自保山及昆明的牛肝菌均有混合, 而红河产地样品与其余产地样品相距较远, 能较好被区分。 表明, 相较低级融合而言中级数据融合能显示相同来源样品间的相互联系, 初步鉴别区分样品的种类及产地。
为解决客观世界中的边际现象(高与低、 美与丑、 好与坏等), 模糊集合论(Fuzzy set)应运而生。 1965年美国学者Zadeh发表“ 模糊集合” , 标志着其的诞生[34]。 不同于经典二值理论描述事物“ 非此即彼” 的绝对性, 模糊集合理论在“ 是” 与“ 否” 之间加入模糊中介, 弥补了非一即二的判断缺陷[35, 36]。 通过最小值、 最大值、 平均值和乘积4个模糊连接运算符对独立光谱模型鉴别不一致样品重新归类, 样品最终归属为多数投票。 表3为部分样品高级融合的具体内容, 粗体为样品归类。 表4为样品高级数据融合鉴别结果, 数字为计算所得最大值, 粗体为数据融合后归类正确样品。 由表3可知, UV-Vis模型下灰褐牛肝菌错判成美味牛肝菌, FTIR鉴别正确, 经高级融合后样品归类正确。 FTIR模型下实际归属为玉溪市的牛肝菌误认为采自昆明市, UV-Vis分类正确, 经高级数据融合后样品归类错误。 由表4知, 种类鉴别不一致样品经高级数据融合后均正确归类, 而产地鉴别中仍有部分样品归属错误。
图4为各判别分析模型对牛肝菌种类及产地的分类鉴别结果。 由图可见, 种类鉴别中FTIR、 UV-Vis、 低级、 中级和高级数据融合验证集样品预测正确率依次为92.86%, 35.71%, 97.62%, 100%和95.23%, 融合模型均高于独立模型, 其中, 中级融合最佳, 预测正确率高达100%, UV-Vis独立模型最差, 正确率仅为35.71%。 各模型对样品产地的预测正确率依次为71.43%, 61.90%, 61.90%, 97.62%和76.19%, 中级融合和高级融合高于独立模型和低级融合, 中级融合最优, 鉴别正确率为97.62%, UV-Vis和低级融合最差, 正确率为61.90%。 表明: 在牛肝菌种类及产地鉴别中, 数据融合在一定程度上优于独立模型; 3种数据融合方式中, 中级融合具有最佳的样品鉴别正确率, 模型泛化性强, 与Má rquez和Biancolillo等研究结果一致[37, 38]。 值得我们注意的是, UV-Vis独立模型对牛肝菌种类及产地的鉴别正确率分别为35.71%和61.90%, 有较大差距。 推测不同鉴别目的, 同一种统计分析技术可能产生不同的鉴别效果。
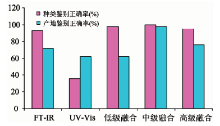 | 图4 各模型对样品种类及产地的鉴别正确率Fig.4 The identification accuracy of each model for the specie and geographic origin |
| 表4 高级数据融合鉴别结果 Table 4 High-level data fusion identification result |
采集牛肝菌样品傅里叶变换红外光谱和紫外可见光谱信息, K-S算法将样品原始数据分为校正集与验证集, 基于FTIR、 UV-Vis、 低级、 中级与高级融合的数据用校正集建立偏最小二乘判别分析模型对样品种类及产地进行鉴别。 FTIR与UV-Vis具有简捷、 高效、 低成本、 绿色等优点。 采用多源异构信息融合技术, 改善样品信息以提高鉴别精度。 实验结果显示: 各模型牛肝菌种类鉴别正确率分别为92.86%, 35.71%, 97.62%, 100%和95.23%, 产地鉴别正确率分别为71.43%, 61.90%, 61.90%, 97.62%和76.19%。 中级融合种类鉴别正确率100%, 产地鉴别正确率97.62%, 实现了样品种类准确鉴别, 产地有效鉴别。 多源异构信息融合策略优于独立模型, FTIR和UV-Vis法结合中级数据融合策略可实现牛肝菌种类及产地的快速有效鉴别, 可作为准确追溯食用菌来源及正确评价其品质的可靠办法。
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|