作者简介: 沈 飞, 1984年生, 南京财经大学食品科学与工程学院副教授 e-mail: shenfei0808@163.com
稻谷是我国主要储粮品种。 为快速、 准确鉴定稻谷霉变状态, 建立了一种基于近红外光谱的稻谷霉菌污染定性、 定量分析方法。 首先, 将四种谷物中常见有害霉菌(黄曲霉3.17、 黄曲霉3.3950、 寄生曲霉3.3950、 灰绿曲霉3.0100)分别接种在灭菌稻谷样品上。 其次, 将接种霉菌样品进行人工模拟储藏(28 ℃、 RH 80%), 并采集不同储藏时间(0, 2, 4, 7和10 d)稻谷的近红外漫反射光谱信号。 最后, 利用主成分分析(PCA)、 判别分析(DA)和偏最小二乘回归(PLSR)方法建立稻谷霉菌污染的快速分析模型。 结果显示, 近红外光谱可有效区分感染不同霉菌的稻谷样品, 平均判别正确率达87.5%。 稻谷霉变随储藏时间逐渐加深, 近红外光谱对感染单一霉菌稻谷样品霉变状态的判别正确率达92.5%, 多种霉菌的判别正确率达87.5%。 稻谷中的菌落总数的PLSR模型定量结果为: 有效决定系数(
China has huge rice reserves. In order to develop a rapid and accurate method for harmful mold infection detection in rice, near infrared (NIR) spectroscopy was applied for qualitative and quantitative analysis of the process of rice mildew in this study. Sterilized rice samples were firstly inoculated with four mold Aspergillus spp. species ( A. flavus 3.17, A. flavus 3.3950, A. parastiticus 3.3950, A. glaucus 3.0100), respectively. Then the rice samples were stored under appropriate conditions (28 ℃, 80% RH) for mould growth. NIR spectra of samples were collected during the storage on different days (0, 2, 4, 7 and 10 d). Analysis models of mold infection in rice were developed by principal component analysis (PCA), discriminant analysis (DA) and partial least squares regression (PLSR), respectively. The results indicated that rice samples infected by different mold species could be effectively distinguished by NIR spectroscopy, and the average classification accuracy was 87.5%. The degree of mildew intensified during storage. The average correct classification accuracy of storage time (mildew degree) was found to be 92.5% for samples infected by one mold species, and 87.5% for samples infected by the four mold species. The PLSR prediction results of mould cell concentration in samples was:
稻谷是关系国计民生的主要储备粮种。 然而, 稻谷含有碳水化合物、 蛋白质等营养成分, 在适宜的温湿度条件下极易受霉菌感染, 导致粮食变质, 降低粮食的食用价值, 带来巨大的经济损失。 此外, 霉菌产生的真菌毒素还可引起人与动物中毒, 严重威胁人畜健康[1]。 因此, 实现稻谷有害霉菌侵染的早期检测显得尤为重要。
目前霉菌等微生物污染的检测方法主要有干片培养法、 荧光分析法、 微生物活性测定法等。 然而, 这些传统的霉变检测方法普遍较为繁琐、 耗时, 误差较大, 难以满足快速准确检测的需要。 因此, 高光谱成像、 红外光谱、 电子鼻等等快速检测方法受到了越来越广泛的关注。 近红外光谱(near infrared spectroscopy, NIRS), 由于具有无需样品预处理、 测定时间短、 不使用化学试剂、 可同时测定多组分等优点[3], 已被广泛应用到农产品/食品品质与安全检测中。 在稻谷研究方面, 已有学者应用NIRS建立了稻谷新陈度及理化指标的定标模型, 并得到了较好的结果。 在食品霉变检测方面, 对鱼类[4]、 花生[5]、 板栗[6]、 鲜枣[7]等的霉变状态进行了快速检测。 Tito等[8]利用近红外光谱成功预测了大西洋鲑鱼的腐败情况, 发现新鲜鲑鱼片和在4 ℃储藏9 d的鲑鱼片的光谱信息存在显著差异。 黄星奕等[9]建立花生不同霉变程度的快速识别模型, 训练集与预测集正确率均达到98.84%。 周竹等[10]建立了基于最小二乘支持向量机分类器等算法的霉变板栗识别模型, 霉变板栗的总体平均识别正确率超过90%。 Zhang等[11]利用NIRS建立了稻米中霉菌菌落总数的预测模型, 发现待测样品中霉菌总数的预测值与实际值之间呈现一定的线性关系, 相关系数达到0.897。
总体而言, 运用近红外技术检测理化指标的研究较多, 但对于霉变研究相对较少, 且主要集中于霉变状态的定性区分, 对受不同霉菌感染从而产生的差异涉及不多。 因此, 本研究拟以接种不同产毒菌株的稻谷样品为研究对象, 追踪检测其在不同储藏阶段(0, 2, 4, 7和10 d)的近红外光谱信息, 建立稻谷受不同霉菌感染以及不同霉变程度的快速判别模型, 并对稻谷中菌落总数进行定量预测, 实现稻谷受有害霉菌侵染的定性与定量分析, 为稻谷储运安全提供技术支撑。
以经15kGay钴60辐照灭菌处理的稻谷(淮稻5号)为研究对象, 调节样品水分至20%, 于4 ℃冷藏储存备用。 选取黄曲霉3.17、 黄曲霉3.3950、 寄生曲霉3.3950、 灰绿曲霉3.0100四种谷物常见有害霉菌, 分别接种于马铃薯葡萄糖琼脂(potato dextrose agar, PDA)培养基, 在28 ℃恒温培养箱中进行培养, 待霉菌生长14 d后产生大量的孢子, 制成浓度约105 CFU· mL-1霉菌的孢子菌液备用。
将灭菌稻谷样品分装为四份, 每份2 400 g, 对每份稻谷样品分别接种四种霉菌菌液20 mL并充分搅拌均匀。 将每份样品用塑料保险盒再分装为六小份, 每份400 g, 共计24份样品, 随后将所有样品放置于28 ℃, 80%相对湿度的人工气候箱中储藏。 从接种霉菌起, 选取时间节点0, 2, 4, 7和10 d, 从每一份样品中取出80 g用于分析, 共计取样五次, 每次取样24个, 合计取样量为120个。 每次取样前后均进行充分搅拌以保证霉菌分布均匀, 样品分析前放入密封袋中于4 ℃冷藏备用。
为实现样品的快速检测, 稻谷样品不进行粉碎等任何前处理, 扫描前将冷藏的样品置于室温下2 h直至样品达到室温。 运用MB3600型近红外光谱仪(ABB-Bomem公司, 魁北克, 加拿大)和InGaAs光电检测器扫描样品, 扫描波数范围为12 000~4 000 cm-1, 分辨率为4 cm-1, 扫描64次, 样品重复扫描3次, 取平均光谱。
采集光谱后, 参照GB/T 4789.15— 2010[12]进行稻谷霉菌菌落总数的测定。 因菌落总数数量级较大, 将得到的结果取对数, 以方便建模分析。
为消除来自高频随机噪声、 基线漂移、 样本不均匀等, 采用多元散射校正(multiplicative signal correction, MSC)、 一阶微分(1st)及Savitzky-Golay平滑对光谱数据进行预处理。
主成分分析(principal component analysis, PCA)是一种降维的多元统计方法, 通过PCA方法可得到样品的最佳描述特征[21]。 因此首先通过主成分分析提取受霉菌侵染稻谷近红外光谱的主成分, 再通过判别分析(discriminant analysis, DA)对接种不同霉菌的稻谷样品及不同储藏阶段稻谷中霉菌的生长状况定性区分。 其次通过偏最小二乘回归(partial least squares regression, PLSR)对稻谷中霉菌总数进行定量分析。 PLSR分析时, 将稻谷的近红外光谱数据作为自变量, 霉菌总数作为因变量进行建模。
本实验随机选取稻谷样品的2/3作为建模集, 1/3作为预测集。 建模集样品用于建立模型, 预测集样品用于对所建模型的验证。 分析均使用TQ Analyst v6.2.1(Thermo Electron Corp., Madison, WI, USA)软件。
图1为接种4种有害霉菌稻谷样品的平均光谱图, 可以看出样品的近红外光谱在9 000~4 000 cm-1波段范围内整体相似, 存在许多共有峰谱, 感染不同灭菌样品未发现显著差异。 绝大多数有机物在近红外谱区的吸收主要是含氢基团伸缩振动的各级倍频和伸缩振动与弯曲振动的合频吸收[13]。 随着霉菌菌落生长, 稻谷中淀粉、 糖类、 水分、 脂质和蛋白质发生变化, 并且霉菌开始产生代谢产物, 从而引起稻谷光谱出现不同程度变化。 其中, 8 334和5 605 cm-1处分别为脂肪族烃C— H伸缩振动二级倍频、 一级倍频; 6 890 cm-1处为芳烃C— H伸缩振动组合频; 5 184 cm-1处为水分子中O— H的组合频; 4 800和4 386 cm-1处为蛋白质中C=O, N— H键的伸缩振动二阶倍频; 4 050 cm-1处为淀粉C— H伸缩, C— C和C— O— C伸缩振动组合频[14]。 未接种霉菌的稻谷整体吸光值较弱, 可能是因为稻谷中本身所含油脂和水分较低, 而受霉菌侵染的稻谷在储藏过程中品质发生变化。 在9 000 cm-1以后样品光谱噪声较大, 对模型的精度及稳定性造成一定影响, 故仅选取9 000~4 000 cm-1范围内进行分析。
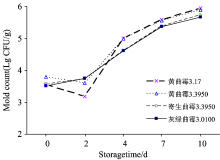
接种四种不同霉菌稻谷样品中菌落总数变化如图2所示。 由图可知, 四种霉菌在0~2 d时生长较为平缓, 甚至有些霉菌的菌落总数有所下降, 可能由于在稻谷表面接种菌液初始, 霉菌需进入稻谷的稻壳内部, 此时的霉菌生长处于延滞期并需要适应新的环境。 但在2~10 d时, 随着贮藏时间的增加, 稻谷中霉菌生长处于对数期, 霉菌菌落总数呈递增趋势, 到后期稻谷样品已发生严重霉变。 四种霉菌生长速度整体差异不大。
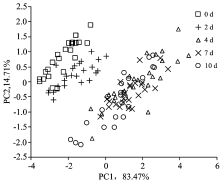
选取9 000~4 000 cm-1波数范围进行主成分分析, 发现第一主成分的贡献率为83.47%, 第二主成分的贡献率为14.71%, 两个主成分贡献之和为98.18%。 第一主成分和第二主成分得分图如图3所示。 总体而言, 不同的储藏阶段稻谷样品存在一定的变化规律。 在霉菌接种早期0和2 d时数据点分布均位于左上侧且区分较为明显。 接种后期样品分布均位于右下侧, 接种7 d样品与接种4和10 d均重叠较为严重, 第4 d和10 d样品有部分重叠现象, 这可能是因为在霉菌生长的中后期生长状况较为相似。 通过主成分分析可以发现, 尽管样品存在部分重叠现象, 但不同储藏阶段样品仍呈现出一定的聚类趋势。
运用从主成分分析提取的前10个主成分得分建立DA判别模型。 表1为在不同储藏阶段稻谷受4种不同有害霉菌感染的判别结果。 结果显示, 在验证组中, 0 d时, 一个黄曲霉3.17组样品误判为黄曲霉3.3950组; 2 d时, 一个黄曲霉3.17组样品误判为灰绿曲霉3.0100组; 4 d时, 一个黄曲霉3.3950组样品误判为灰绿曲霉3.0100组; 7 d时, 一个灰绿曲霉3.0100组样品误判为黄曲霉3.3950组, 一个灰绿曲霉3.0100组样品误判为寄生曲霉3.3950组。 在同一天, 稻谷受霉菌的正确识别率均较高, 建模平均正确率为96.25%, 验证平均正确率为87.5%。 表明不同霉菌所产生的代谢产物存在一定差异, 利用近红外光谱可以用来区分不同霉菌菌属, 以判定稻谷是否受产毒和不产毒霉菌侵染。
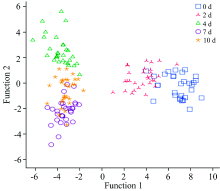
表2为稻谷样品在不同储藏阶段(霉变不同阶段)的DA模型判别统计结果。 对每种霉菌而言, 在五次取样获得的30个样品中选取2/3样品作为建模组, 1/3样品作为验证组。 结果显示, 对四种霉菌在不同储藏阶段的正确识别率均较优, 预测平均正确率为92.5%。 预测集中黄曲霉3.3950组有2个样品被错误区分, 其中一个2 d样品误判为4 d, 一个4 d样品误判为2 d; 灰绿曲霉3.0100组有一个样品被错误区分, 一个4 d样品误判为7 d。 分析发现黄曲霉3.3950组和灰绿曲霉3.0100组被错误区分的样品均处于2和4 d, 可能是因为在储藏前期霉菌刚开始生长时产生的差异并不十分显著。 将所有120个样品一起建立模型, 建模组和验证组的正确率分别为93.75%和87.5%, 表明尽管不同的霉菌生长产生的代谢产物存在一定差异, 但随着储藏过程霉变程度的加深和菌落总数的增加, 近红外光谱可有效获取样品的整体差异信息, 区分样品的霉变状态。 图4为稻谷样品在不同储藏阶段DA模型第一、 第二判别函数得分图; 稻谷样品在0和2 d时聚集较近且有少部分重合, 位于图右侧, 4, 7和10 d时均聚集在图左侧, 区分较为明显。
| 表1 稻谷样品受不同有害霉菌 感染的DA模型判别结果 Table 1 DA discrimination results of paddy samples infected by 4 different fungal species |
| 表2 稻谷样品在不同储藏阶段的DA模型判别结果 Table 2 DA discrimination results of paddy samples at different storage periods |
表3为稻谷中菌落总数的PLSR定量分析统计结果, 较大的
| 表3 稻谷样品感染4种霉菌的菌落总数的PLSR模型预测分析结果 Table 3 PLSR prediction results for mold count in paddy samples infected by 4 different fungal species |
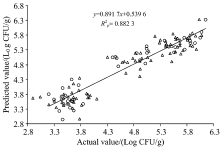 | 图5 稻谷样品中菌落总数与近红外 光谱预测值的相关关系 ○: 建模集; △ : 预测集Fig.5 Mold count of paddy samples using PLSR prediction models on the NIR spectra ○: Calibration set; △ : Prediction set |
对受霉菌侵染的全部稻谷样品建立PLSR分析模型。 预测集的
开发了一种基于近红外光谱技术的稻谷受有害霉菌污染程度的定性、 定量分析方法。 结果表明, 近红外光谱不仅可以用于感染不同霉菌稻谷样品的快速鉴别, 还可有效区分不同霉变程度的稻谷, 总体判别正确率均在85%以上。 运用近红外光谱可实现稻谷中菌落总数的定量分析, 本模型有效决定系数
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|